Результат интеллектуальной деятельности: Способ преимплантационного генетического тестирования синдрома Альпорта
Вид РИД
Изобретение
Изобретение относится к преимплантационному генетическому тестированию моногенных заболеваний. В настоящее время в мире насчитывается более 350 миллионов людей, страдающих редким заболеванием (по данным RARE Project). Общее количество таких заболеваний по подсчетам European Organization for Rare Diseases (EURORDIS) варьируется от 5 до 7 тысяч. При этом около 80% редких заболеваний имеют генетическую причину. Известная генетическая основа заболевания позволяет с высокой точностью предсказать не только здоровье уже родившегося ребенка, но и оценить риск рождения такого ребенка при анализе генотипов родителей, а также провести генетическую диагностику на самых ранних этапах. Преимплантационное генетическое тестирование (ПГТ) моногенного заболевания становится мощным инструментом для профилактики таких заболеваний.
Настоящее изобретение относится к способу преимплантационного генетического тестирования синдрома Альпорта. Семейная гломерулярная гематурия (Синдром Альпорта) - это генетически гетерогенное заболевание, все формы которого обусловлены мутациями в генах, кодирующих коллаген IV типа, который является основным структурным компонентом базальной мембраны. Примерно 85% случаев синдрома Альпорта являются х-сцепленными и около 15% - аутосомно-рецессивными; аутосомно-доминантное наследование встречается редко. Семейная гломерулярная гематурия (Синдром Альпорта) встречается с частотой 1 на 5,000-10,000. Это заболевание приводит к врожденному нарушению базальной мембраны, приводящее к прогрессирующей почечной недостаточности вследствие гломерулонефропатии, вариабельной нейросенсорной тугоухости и вариабельным глазным аномалиям. При Х-сцепленном доминантном типе наследования заболевания вероятность рождения ребенка с этим заболеванием в семье составляет 50%.
К заболеванию семейная гломерулярная гематурия (синдром Альпорта) могут приводить патогенные генетические варианты в гене COL4A5, располагающемся на хромосоме X. [Crockett, D.K et ai. Hum. Mutat, 31: E1652-E1657] Этот ген кодирует белок одна из шести субъединиц коллагена IV типа, основной структурный компонент базальных мембран.
ПГТ синдрома Альпорта проводится для семей, имеющих подтвержденную молекулярно-генетическую природу заболевания. Важно отметить, что обоснование патогенности и каузативности генетических вариантов происходит до проведения ПГТ моногенного заболевания и не входит ни цели и задачи ПГТ моногенного заболевания, ни в комплекс мероприятий по проведению ПГТ моногенного заболевания. Оценку патогенности проводят по международному стандарту - по критериям, описанным в 2015 году Американским Колледжем Медицинской генетики и Геномики (American College of Medical Genetics and Genomics-Association for Molecular Pathology (ACMG-AMP)) в ходе поиска молекулярно-генетической причины заболевания. ПГТ рекомендуется семьям с высоким риском рождения ребенка с тяжелым (неизлечимым) наследственным заболеванием с установленным патогенными вариантом, обуславливающим этот риск. ПГТ позволяет выбрать из всех эмбрионов, полученных при ЭКО (экстракорпоральном оплодотворении), эмбрионы без патогенного варианта и, следовательно, без риска развития заболевания.
Основной проблемой при генетической диагностике эмбрионов является малое исходное количество биоматериала, так как биоптат содержит от одной до трех клеток. В этом случае для повышения эффективности и точности анализа важно как полностью исключить возможность контаминации, так и нивелировать возможный эффект неравномерной и/или неполной амплификации, а также деградации биоматериала. Это требует разработки тест-системы с особыми характеристиками. При этом тест-система разрабатывается с учетом возможности использовать биоматериал разного типа - тотальную дезоксирибонуклеиновую кислоту (ДНК), выделенную из разных тканей, продукт полногеномной амплификации (Whole Genome Amplification, WGA), а также единичные клетки. Сочетание универсальности по отношению к биоматериалу с поэтапной амплификацией целевых фрагментов позволяет проводить анализ нескольких патогенных вариантов для одного образца, в том числе на единичных клетках, а также выявить ситуацию неполной амплификации, контаминации или деградации образца. Еще одной особенностью ПГТ является отсутствие информации о биологических особенностях эмбриона: в отличие от взрослого человека, у эмбриона могут быть любые хромосомные аномалии, которые усложняют задачу оценки статуса эмбриона по конкретному генетическому варианту. Поэтому тест-система для ПГТ моногенного заболевания должна давать возможность выявить такие случаи и оценить их влияние на достоверность результата диагностики.
Описания близкого технического решения не обнаружено в общедоступных источниках.
Представленный нами метод ПГТ синдрома Альпорта решает задачу разработки более точного способа преимплантационного генетического тестирования этого моногенного заболевания без использования дорогостоящих приборов и реагентов, который можно было бы применять на биоматериале различного типа: ДНК, выделенную из разных тканей, продукт полногеномной амплификации (WGA), единичные клетки.
Техническим результатом стало создание тест-системы для диагностики патогенного варианта (номер нуклеотида в референсной последовательности геномной ДНК обозначен префиксом NC, номер нуклеотида в референсной последовательности кодирующего транскрипта обозначен префиксом NM): NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) в гене COL4A5 с двойной системой детекции - прямой и косвенной. Данный вариант известен как патогенный из научной литературы [Kovacs, Gabor et al. PloS one vol. 11,3 e0149241. 2016] Такая система необходима при работе с малым количеством биоматериала, так как нестабильная амплификация может привести к потере информации или сниженной точности анализа. Прямая диагностика подразумевает анализ непосредственно наличия-отсутствия патогенного варианта. В данном случае для генетического варианта NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) типа однонуклеотидный полиморфизм (ОНП, англ. Single nucleotide polymorphism SNP) были подобраны эндонуклеазы рестрикции, которые позволяют произвести детекцию патогенного варианта методом ПЦР-ПДРФ (полиморфизм длин рестриктных фрагментов), основанным на разнице последовательности в сайте рестрикции у различных аллелей. Косвенная диагностика заключается в анализе наследования молекулярно-генетических маркеров, сцепленных с мутацией, т.е. наследуемых вместе с ней. Для этого на расстоянии не более 3 мБ (что соответствует 3% кроссинговера в среднем) от гена COL4A5 в каждую сторону были выбраны полиморфные локусы, называемые STR (short tandem repeat - короткий тандемный повтор), с гетерозиготностью не менее 0,70 для обеспечения максимальной информативности косвенной диагностики. STR представляют собой повторы 2х и более нуклеотидов расположенные друг за другом (например пара аденин-цитозин (АЦ), повторяющаяся несколько раз подряд: АЦАЦАЦАЦ) и в большом количестве присутствуют в геноме человека. Количество повторов в каждом из них может варьироваться от индивидуума к индивидууму, а также может быть разным у одного и того же человека на 2х гомологичных хромосомах. Гетерозиготность выше 0,70 означает, что высока вероятность того, что у одного и того же человека количество повторов нуклеотидов в данном STR на одной хромосоме будет отличаться от количества повторов в этом же STR на гомологичной хромосоме, другими словами, аллели данного маркера у этого человека будут отличаться между собой по длине. При амплификации фрагмента, содержащего такой маркер, будут получены ампликоны двух разных длин. Проанализировав количество повторов в нескольких маркерах, окружающих патогенный вариант, и изучив то, как они наследуются в тестируемой семье, можно установить сцепление между аллелями маркеров и патогенным вариантом. Диагностическая ценность исследования количества повторов в данных маркерах у эмбрионов состоит в том, что по тому, какой аллель каждого из маркеров был унаследован эмбрионом, можно судить о том, унаследовал ли эмбрион ген COL4A5, несущий патогенный вариант, или же он унаследовал ген COL4A5 с другой, гомологичной хромосомы, не содержащий патогенный вариант. Для каждого из этих локусов были подобраны праймеры для амплификации по типу гнездовой или полугнездовой ПЦР в 2 раунда, позволяющей повысить точность и эффективность амплификации. В тест-систему были включены 11 STR локусов для гена COL4A5: DXS106.7, DXS107.2, DXS1105, DXS107.5, DXS107.6, DXS6802, DXS107.9, DXS108.2, DXS108.5, DXS1210, DXS108.9. Праймеры для амплификации находятся на X хромосоме в районе координат 106703115-108989085 (в соответствии с hg19). Амплификация фрагментов ДНК, содержащих полиморфные маркеры, проводилась с помощью праймеров, перечисленных в перечне SEQ ID NO 1-33 в формуле изобретения. Важно отметить, что при подборе праймеров соблюдали ряд особенных требований: длина продукта с внешними праймерами для первого раунда ПЦР не должна превышать 500 п.н. (для наработки с фрагментов, получаемых при полногеномной амплификации), длина продукта с внутренних праймеров для второго раунда ПЦР от 120 до 350 п.н., высокая специфичность внешних праймеров, температура отжига не отличается более, чем на 1°С.
Подготовительный этап ПГТ
На подготовительном этапе проводится отработка тест-системы: подбор условий амплификации, оптимальных для работы праймеров, анализ эффективности и специфичности ПЦР-амплификации в обоих раундах, оценки универсальности тест-системы для биообразцов различного типа (ДНК, продукт WGA, единичные клетки). При отработке тест-системы были приготовлены стоковые разведения праймеров с концентрацией 100 mM, и рабочие разведения комбинаций праймеров (комбинация пар праймеров для 1 и 2 раунда ПЦР) с концентрацией 10 mM каждого праймера в растворе. Так как в рамках диагностики клинического материала могут быть использованы различные типы матриц, при отработке тест-системы были использованы две биопсии единичных клеток, находящихся в специальном лизирующем буфере (1×PCR Buffer, 0,1% Tween-20, 0,1% Triton X-100, 1 μкг Proteinase K), два образца продуктов полногеномной амплификации биопсиий эмбриона (WGA), а также тотальной ДНК членов семьи, выделенной из крови, для составления родословной и выявления сцепления патогенного варианта с аллелями полиморфных маркеров.
В рамках гнездовой и полугнездовой ПЦР амплификация проводится в два этапа. На первом этапе проводится мультиплексная ПЦР со всеми внешними праймерами для всех локусов, входящих в тест-систему, для обогащения образца всеми целевыми фрагментами. На втором этапе проводится индивидуальная амплификация каждого фрагмента с внутренними праймерами.
Полугнездовая ПЦР
Для первого этапа были подобраны внешние высокоспецифичные праймеры для амплификации фрагментов от 300 до 500 п.н. Для второго этапа были подобраны праймеры для амплификации фрагментов длиной не более 350 пар оснований, а также были введены метки для детекции методом фрагментного анализа. Амплификация фрагментов ДНК, содержащих полиморфные маркеры, проводилась с помощью праймеров, перечисленных в перечне SEQ ID NO 1-33 в формуле изобретения. ПЦР-смесь для первого раунда амплификации содержала 1xPCR Buffer with Mg2+ (Евроген, Россия), 0.1 mM каждого деоксинуклеотида, 0.15 μM каждого праймера, 2,5 U/μl of HsTaq DNA-polymerase (Евроген, Россия), 6% of DMSO и 1 мкл тотальной ДНК или 2,5 мкл WGA или 5 мкл лизирующего буфера с образцом в качестве матрицы. Первый этап амплификации проводился по следующему протоколу: этап денатурации 94°С в течение 2 минут, 30 циклов с понижением температуры отжига праймеров с 62 до 45°С в каждом, этап достройки всех матриц 72°С 10 минут. Далее продукты 1-ого этапа были разнесены в индивидуальные пробирки с одной парой праймеров на определенный локус.
В состав ПЦР смеси для второго этапа входили 1xPCR буфер с Mg2+ (Евроген, Россия), 0.5xRediLoad™ загрузочный буфер (Thermo Fisher Scientific, USA), 0.2 mM каждого деоксинуклеотида, 0.2 μM каждого праймера, 1 U/μl ДНК полимеразы HsTaq (Евроген, Россия), 6% диметилсульфоксида (DMSO) и 1 μl ПЦР-продукта первого этапа амплификации в качестве матрицы. Второй этап амплификации проводился по следующему протоколу: этап денатурации 95°С в течение 2 минут, 35 циклов: денатурация 95°С 30 секунд, отжиг праймеров - 57°С 30 секунд, синтез матрицы - 72°С 1 минута, этап достройки всех матриц 72°С 5 минут. Оценку эффективности и специфичности амплификации проводили с помощью электрофореза в 2% агарозном геле. Результат электрофореза в агарозном геле позволяет определить необходимую степень разведения продуктов амплификации для нанесения на фрагментный анализ (продукты амплификации ДНК членов семьи).
Фрагментный анализ продуктов амплификации проводили с помощью капиллярного электрофореза на приборе 3130xl Genetic Analyzer (Applied Biosystems, USA). По результатам фрагментного анализа составляется родословная и отмечаются информативные полиморфные STR-локусы для каждой семьи, которые в дальнейшем будут использованы в клинической диагностике. Локусы делятся на неинформативные (носитель патогенного варианта гомозиготен по этому локусу), полуинформативные (на некоторых и родительских хромосом аллели по этому маркеру совпадают), информативные (на всех хромосомах родителей аллели этого маркера разные, что дает возможность отличить каждую из них при анализе генотипа эмбриона).
Полимеразная цепная реакция - полиморфизм длин рестрикционных фрагментов (ПЦР-ПДРФ)
Полиморфизм длин рестрикционных фрагментов (Restriction fragment length polymorphism, RFLP) - это способ исследования геномной ДНК, путем специфичного расщипления ДНК с помощью эндонуклеаз рестрикции и дальнейшего анализа размеров образующихся фрагментов (рестриктов) путем гель-электрофореза. При использовании данного метода наблюдаются фрагменты различной длины в зависимости от различий в последовательности нуклеотидов в сайте рестрикции, что позволяет детектировать однонуклеотидные варианты, если они располагаются в сайте рестрикции. Более точную детекцию патогенного варианта может обеспечить секвенирование по методу Сэнгера, однако в условиях ПГТ метод ПЦР-ПДРФ оказывается более эффективным из-за сниженной вероятность выпадения аллеля (allele drop out, ADO) и, как следствие, ошибочного результата по оценке статуса эмбриона по патогенному варианту.
Для детекции патогенного варианта NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) была разработана тест-система на основе ПЦР-ПДРФ. Стадия амплификации подробно описана в предыдущем разделе. Были использованы следующие праймеры:
Внешние праймеры:
5'-CTCAAGGGTGAACCAGGATT-3' (Fout),
5'-ACAGAATGGCAGCATAGGGT-3' (Rout),
Внутренние праймеры:
5'-GCTCCCTGGACCAAACG-3' (fin_mm_wt)
5'-GGCTCCCTGGACCAGAAG-3' (fin_mm_mut)
Далее продукты амплификации с внутренних праймеров для детекции патогенного варианта использовали в реакции рестрикции. Эндонуклеаза Taal разрезает только аллель дикого типа, эндонуклеаза Mboll разрезает только мутантный аллель варианта NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A). Детекция проводилась с помощью электрофореза в 12% полиакриламидном геле.
Пример 1
Пациенты А
В ЦГРМ Генетико обратилась семья А, в которой родился ребенок с заболеванием семейная гломерулярная гематурия (синдром Альпорта) с гемизиготным носительством патогенного варианта NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) в гене COL4A5. Паре было рекомендовано проведение ПГТ заболевания семейная гломерулярная гематурия (синдром Альпорта) в рамках ЭКО для отбора эмбрионов, не унаследовавших заболевание.
Гаплотипирование семьи
На первом этапе был получен биоматериал (периферическая кровь) членов семьи для детекции патогенного варианта и выявления групп сцепления аллелей полиморфных маркеров. Амплификация фрагментов ДНК, содержащих полиморфные маркеры, проводилась с помощью праймеров, перечисленных в перечне SEQ ID NO 1-33 в формуле изобретения. Было проанализировано 11 STR локусов. Из них 9 оказались информативными по матери (пациентке). Таким образом, образцы эмбрионов тестировались только на информативные маркеры и на маркеры половых хромосом - SRY и AMEL.
Аллели полиморфных маркеров, совпадающие у родителя-носителя патогенного варианта NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) в гене COL4A5, и у ребенка с заболеванием синдром Альпорта с подтвержденным наличием того же патогенного варианта, признаются сцепленными с патогенным вариантом и друг с другом. Аллели полиморфных маркеров, не совпадающие у этого родителя и ребенка с заболеванием - признаются сцепленными друг с другом и с нормальным аллелем гена COL4A5. Косвенная диагностика позволяет сделать вывод о наследовании нормального или мутантного аллеля гена даже в случае выпадения аллеля или непрохождения реакции при амплификации фрагментов ДНК для прямой диагностики.
В результате гаплотипирования и определения групп сцепления для семьи А из примера были получены результаты, представленные в таблице 1. Аллели, указанные на одной строке, располагаются на одной хромосоме, то есть, представляют группу сцепления. Таким образом для пациентки представлено 2 группы сцепления, соответствующие каждой из 2х X хромосом женщины. У партнера и ребенка с заболеванием представлено по 1 группе сцепления, так как они мужского пола и имеют лишь по одной X хромосоме. Патогенный вариант NC_000023.10:g.107850123G>A (NM_033380.3:c.2395+1G>A) в гене COL4A5 обозначен коротко: COL4A5 c.2395+1G>A. N в таблице обозначает аллель дикого типа, mut - мутантный аллель. Цифрами записаны длины ампликонов в парах нуклеотидов; их длины зависят от количества повторов в маркере STR.

В результате гаплотипирования был сделан вывод, что у пациентки с патогенным вариантом были сцеплены следующие аллели STR-маркеров: DXS106.7 - 187, DXS107.2 - 277, DXS 1105 - 180, DXS 107.5 - 257, DXS107.6 - 238, DXS107.9 - 187, DXS108.2 - 316, DXS1210 - 244, DXS 108.9 - 180.
Преимплантационное генетическое тестирование
В цикле ЭКО было получено 5 эмбрионов, проведена биопсия на 5 день развития (в клинике ЭКО), биоптат в буфере для WGA (1xPBS (Invitrogen, США), 1% поливинилпирролидона (PVP) (Fertipro, Бельгия)) направлен в лабораторию «Генетико». Для контроля контаминации на разных этапах работы с образцом в лаборатории разработана система контролей: контроль контаминации буфера для биопсии, контроль контаминации при транспортировке (одна пробирка с буфером не открывается эмбриологом), контроль контаминации каждого образца (проба среды из последней отмывочной капли биопсиийного материала). Все эти контроли вместе с образцами проходят этап полногеномной амплификации, после которого будет заметно малейшее количество ДНК, контаминировавшей контроли. Полногеномную амлификацию проводили с помощью коммерческого набора SurePlex (Illumina, США).
Продукт полногеномной амплификации, а также ДНК всех членов семьи амплифицировали на 1 этапе в мультиплексной ПЦР с праймерами для детекции патогенного варианта и праймерами для информативных для семьи А полиморфных маркеров в соответствии с разработанным в рамках подготовительного этапа протоколом для тест-системы. На 2 этапе амплификацию проводили для каждого маркера отдельно в соответствии с разработанным протоколом для тест-системы. Таким образом были установлены группы сцепления, унаследованные каждым эмбрионом. Результаты ПГТ-М представлены в таблице 2. Результаты для эмбриона 5 не показаны, так как для этого образца не прошла амплификация.
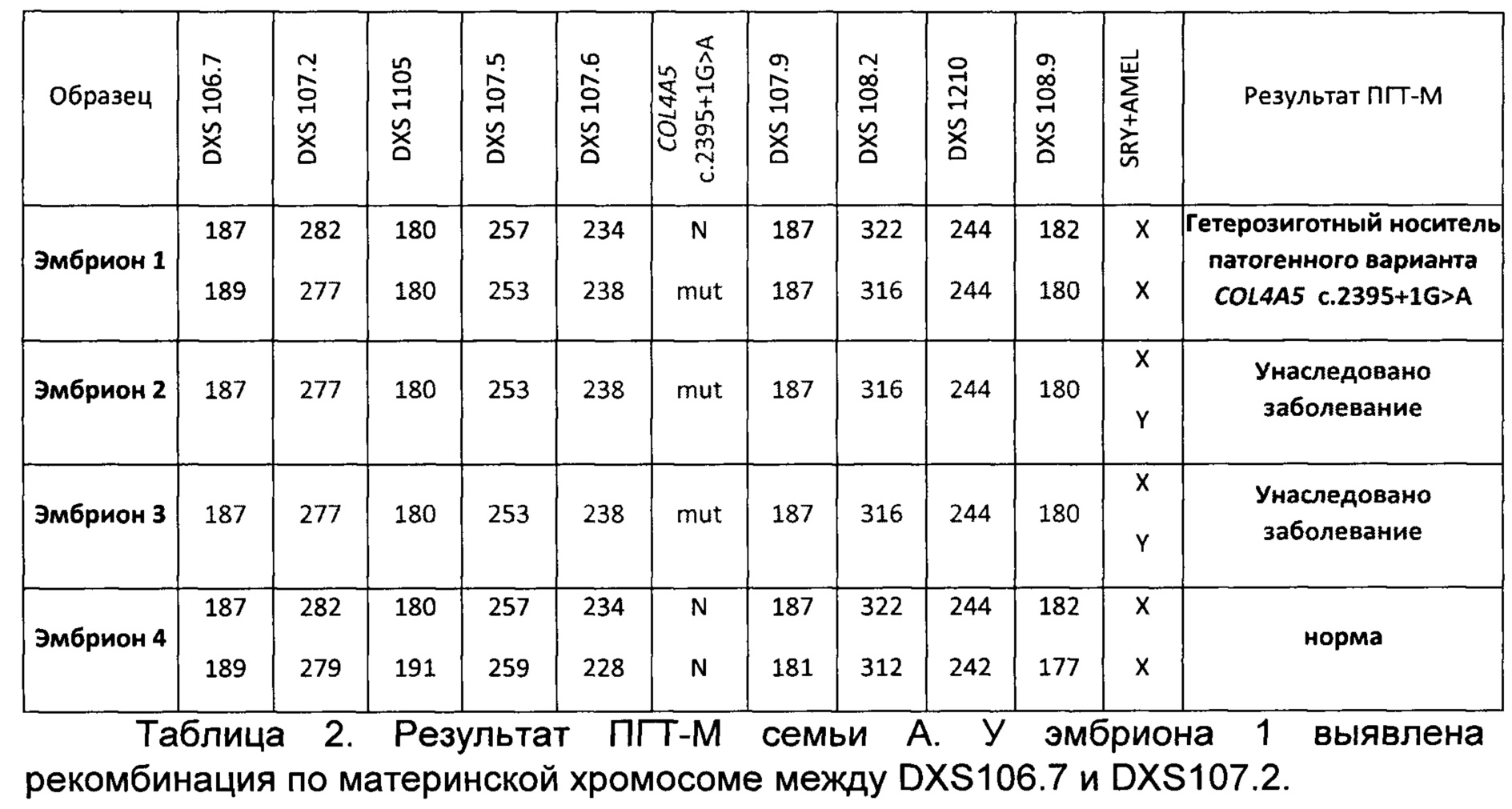
Прямая диагностика выявила у эмбриона 1 гетерозиготное носительство патогенного варианта c.2395+1G>A в гене COL4A5, что было подтверждено косвенной диагностикой. Эмбрион 1 не унаследовал заболевание синдром Альпорта. Без анализа полиморфных маркеров и определения унаследованных групп сцепления у этого эмбриона было бы невозможно отличить гетерозиготное носительство патогенного варианта от контаминации образца нормальным аллелем.
Эмбрионы 2 и 3 унаследовали заболевание синдром Альпорта, что было установлено как прямой, так и косвенной диагностикой. Косвенная диагностика позволила убедиться, что имеет место именно унаследованное заболевание, а не выпадение нормального аллеля гена COL4A5 при амплификации.
Как прямая, так и косвенная диагностика показали, что эмбрион 4 не унаследовал патогенный вариант и, следовательно, не унаследовал заболевание. Без косвенной диагностики невозможно было бы отличить эту ситуацию от выпадения мутантного аллеля гена COL4A5 при амплификации. С согласия родителей для эмбриона, не унаследовавшего заболевание, провели преимплантационный генетический скрининг хромосомных аномалий. По результатам всех проведенных анализов эмбрион 4 был рекомендован к переносу в цикле ЭКО.
--->
<?xml version="1.0" encoding="UTF-8"?>
<!DOCTYPE ST26SequenceListing PUBLIC "-//WIPO//DTD Sequence Listing
1.3//EN" "ST26SequenceListing_V1_3.dtd">
<ST26SequenceListing dtdVersion="V1_3" fileName="Способ
преимплантационного генетического тестирования синдрома Альпорта.xml"
softwareName="WIPO Sequence" softwareVersion="2.2.0"
productionDate="2022-12-21">
<ApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022127644</ApplicationNumberText>
<FilingDate>2022-10-25</FilingDate>
</ApplicationIdentification>
<ApplicantFileReference>2022127644</ApplicantFileReference>
<EarliestPriorityApplicationIdentification>
<IPOfficeCode>RU</IPOfficeCode>
<ApplicationNumberText>2022127644</ApplicationNumberText>
<FilingDate>2022-10-25</FilingDate>
</EarliestPriorityApplicationIdentification>
<ApplicantName languageCode="ru">Акционерное общество "Центр
Генетики и Репродуктивной Медицины "Генетико"
</ApplicantName>
<ApplicantNameLatin>Genetico</ApplicantNameLatin>
<InventionTitle languageCode="ru">Способ преимплантационного
генетического тестирования синдрома Альпорта</InventionTitle>
<SequenceTotalQuantity>27</SequenceTotalQuantity>
<SequenceData sequenceIDNumber="1">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q2">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaacaggcttctccttgct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="2">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q4">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cactctggacacctgcctact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="3">
<INSDSeq>
<INSDSeq_length>18</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..18</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q6">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gaggccgctgattgacag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="4">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q8">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caacagctgtgagagatccag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="5">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q10">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cccaattctctagcctgcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="6">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q12">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caaaggcccaattcaaaact</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="7">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q14">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gcagtgatgggtagtcaaaca</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="8">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q16">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgcacagcaactctctaagct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="9">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q18">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtgttttgtgaggttcaagcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="10">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q20">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agttgttccaagtgacaagcata</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="11">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q22">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gatcttgccacagttccgt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="12">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q24">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cttatgggcaccatgtcaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="13">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q26">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgagaccctgtctccattcc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="14">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q28">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>gtagttgggtcagatgggtctt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="15">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q30">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcattctgacagtctctgcattta</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="16">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q32">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ggagtgtccacaagttggct</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="17">
<INSDSeq>
<INSDSeq_length>19</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..19</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q34">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tccacctcatcccctgagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="18">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q36">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>ctcaataccacagtcttggtgg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="19">
<INSDSeq>
<INSDSeq_length>24</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..24</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q38">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgtaccaaatgtggttttaccagt</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="20">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q40">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tgttcaaatttggatgggtg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="21">
<INSDSeq>
<INSDSeq_length>26</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..26</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q42">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>atggaagttattcatgtagacctcaa</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="22">
<INSDSeq>
<INSDSeq_length>23</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..23</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q44">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cagcagggagatagtcctagaac</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="23">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q46">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>cctgatgtctctgcctttagc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="24">
<INSDSeq>
<INSDSeq_length>21</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..21</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q48">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>tcatctggttgaaaacacacc</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="25">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q50">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>caggccaatgtaagcctctg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="26">
<INSDSeq>
<INSDSeq_length>20</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..20</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q52">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>aagctcctggatgtcctttg</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
<SequenceData sequenceIDNumber="27">
<INSDSeq>
<INSDSeq_length>22</INSDSeq_length>
<INSDSeq_moltype>DNA</INSDSeq_moltype>
<INSDSeq_division>PAT</INSDSeq_division>
<INSDSeq_feature-table>
<INSDFeature>
<INSDFeature_key>source</INSDFeature_key>
<INSDFeature_location>1..22</INSDFeature_location>
<INSDFeature_quals>
<INSDQualifier>
<INSDQualifier_name>mol_type</INSDQualifier_name>
<INSDQualifier_value>unassigned DNA</INSDQualifier_value>
</INSDQualifier>
<INSDQualifier id="q54">
<INSDQualifier_name>organism</INSDQualifier_name>
<INSDQualifier_value>unidentified</INSDQualifier_value>
</INSDQualifier>
</INSDFeature_quals>
</INSDFeature>
</INSDSeq_feature-table>
<INSDSeq_sequence>agggagtccttttgtgactcag</INSDSeq_sequence>
</INSDSeq>
</SequenceData>
</ST26SequenceListing>
<---

