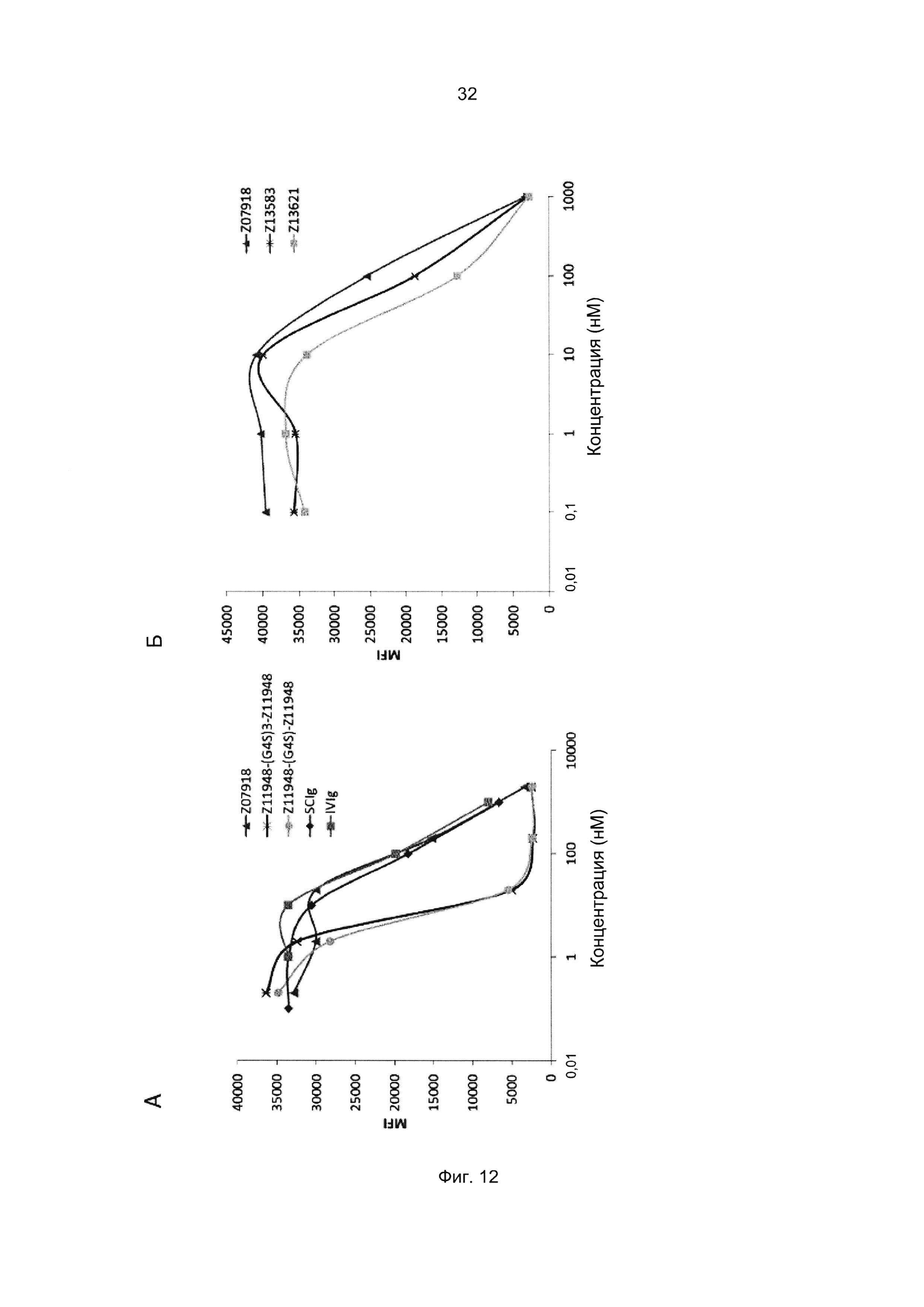
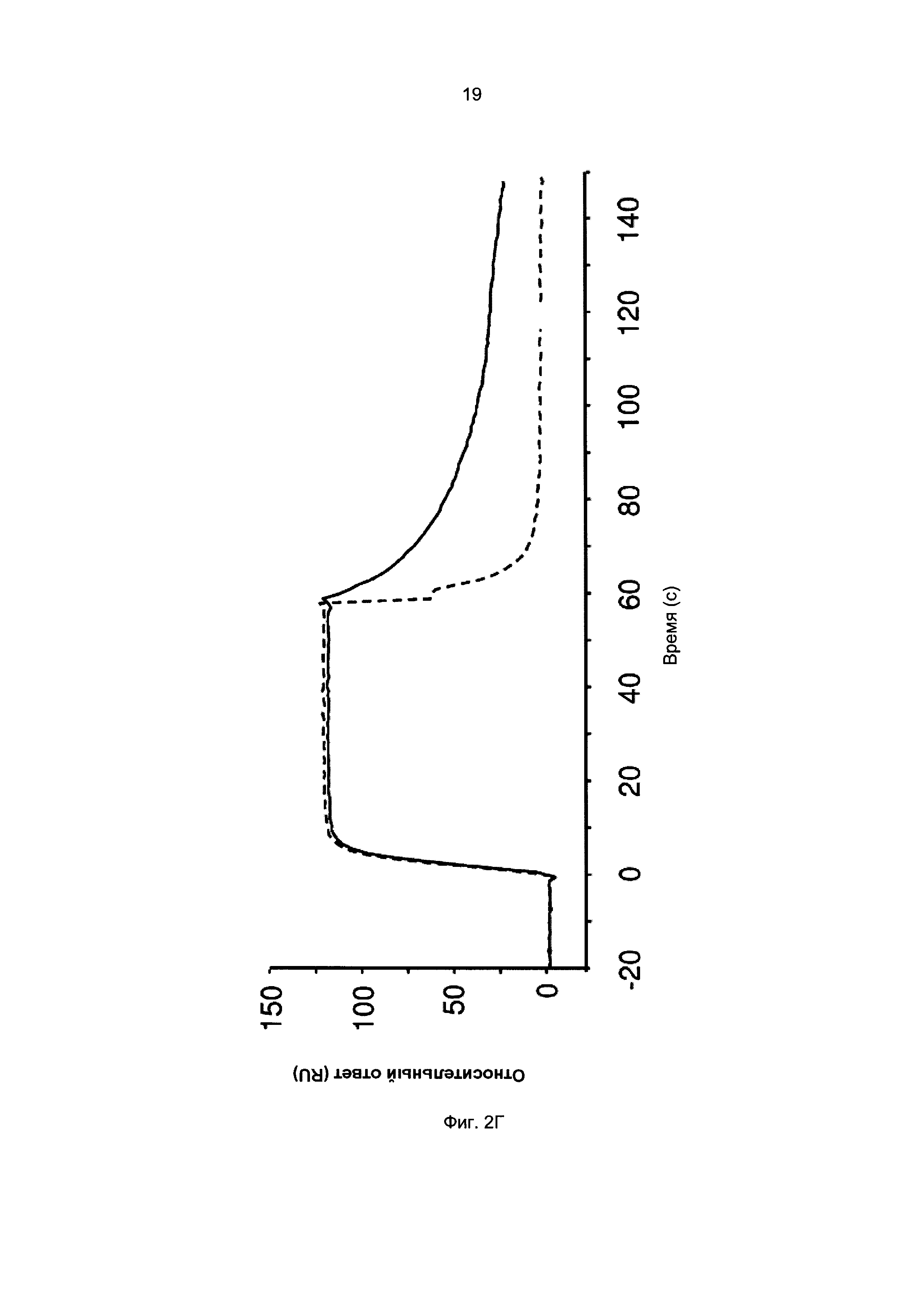
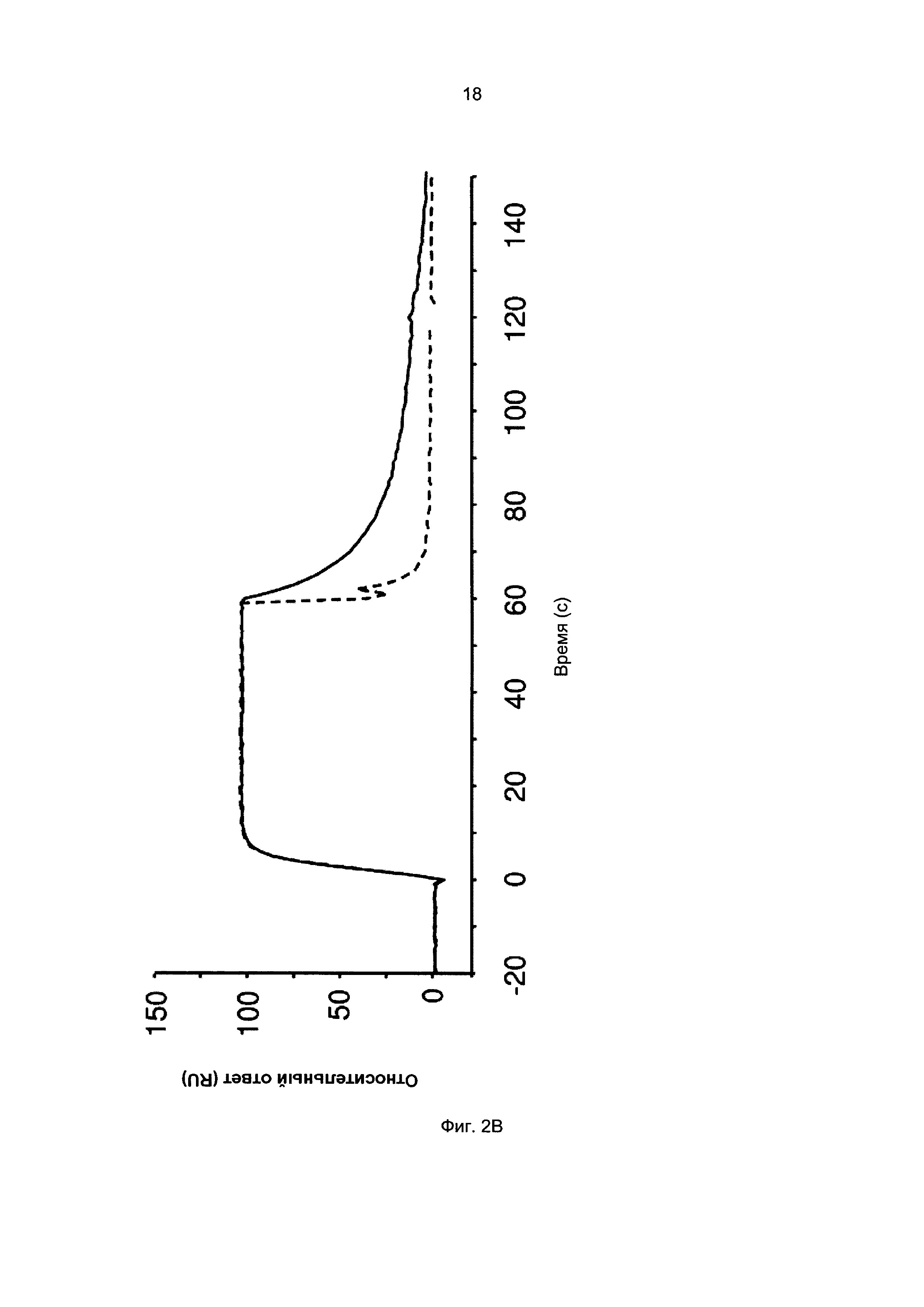
Результат интеллектуальной деятельности: Новые полипептиды
Вид РИД
Изобретение
ОБЛАСТЬ ИЗОБРЕТЕНИЯ
Данное изобретение относится к димерам сконструированных полипептидов, обладающим аффинностью связывания в отношении неонатального Fc рецептора (в дальнейшем обозначаемого FcRn). Данное изобретение также относится к применению таких димеров в качестве агентов для модификации фармакокинетических и фармакодинамических свойств биомолекулы, например фармацевтического средства, а также в качестве терапевтических агентов.
ПРЕДШЕСТВУЮЩИЙ УРОВЕНЬ ТЕХНИКИ
Неонатальный Fc рецептор (FcRn) представляет собой гетеродимерный белок, состоящий из тяжелой цепи, подобной трансмембранному МНС (главный комплекс гистосовместимости) класса I (α-цепь FcRn), и легкой цепи β2-микроглобулина, которая также образует часть молекул МНС класса I (Simister and Mostov (1989) Nature 337: 184-7; Burmeister et al. (1994) Nature 372: 379-83).
FcRn преимущественно локализован в эндосомах и способен связываться с сывороточным альбумином и иммуноглобулином G (IgG) при pH 6,5 или менее и высвобождать их при pH 7,0 или более (обзор представлен Roopenian (2007) Nat Rev Immunol 7: 715-25).
У млекопитающих FcRn выполняет несколько различных задач (Roopenian, см. выше). FcRn задействован в рециркуляции поглощенного путем эндоцитоза IgG и сывороточного альбумина, что позволяет избежать их деградации в лизосомах, обеспечивает им более длительный период полувыведения и большую доступность в крови по сравнению с другими сывороточными белками. Когда IgG, сывороточный альбумин и другие сывороточные белки пассивно поступают в клетку путем пиноцитоза при контакте с кровью, pH в образовавшихся эндосомах постепенно снижается, что позволяет IgG и сывороточному альбумину связываться с FcRn. Затем рецептор вместе с его присоединенным лигандом транспортируется с помощью рециркулирующих эндосом обратно на плазматическую мембрану. После возвращения к плазматической мембране pH возрастает выше 7, в результате чего связанный лиганд высвобождается.
FcRn также известен своей способностью транспортировать IgG через такие барьеры, как плацента, эпителий верхних дыхательных путей, гематоэнцефалический барьер и проксимальный отдел тонкого кишечника.
Благодаря свойствам FcRn у млекопитающих осуществляется трансцитоз IgG через плаценту от матери к плоду и трансцитоз IgG из материнского молока в кровоток младенца в проксимальном отделе тонкого кишечника.
Паттерн экспрессии FcRn различается у разных биологических видов. Однако у большинства видов FcRn широко экспрессируется клетками гематоэнцефалического барьера, эпителия верхних дыхательных путей, почек и эндотелия сосудов, антиген-презентирующими клетками, а также другими клетками гемопоэтического происхождения (Roopenian (2007), см. выше).
Были созданы антитела и пептиды, обладающие аффинностью к FcRn (Liu et al. (2007) J Immunol 179: 2999-3011, Mezo et al. (2008) Proc Natl Acad Sci USA 105: 2337-42) и β2-микроглобулину (Getman and Balthasar (2005) J Pharm Sci 94: 718-29), для ингибирования связывания эндогенных IgG с FcRn. Другой подход заключается в мутировании Fc-области IgG для повышения аффинности к FcRn (Petkova et al. (2006) Int Immunol 18: 1759-69, Vaccaro et al. (2005) Nat Biotechnol 23: 1283-8).
Для увеличения периода полувыведения белков in vivo широко применяют стратегию слияния с Fc доменом или с альбумином. Однако крупный размер таких слитых белков негативно влияет на проникновение в ткани и снижает специфичность к партнеру по слиянию (Valles et al. (2011) J Interferon Cytokine Res 32: 178-184). С другой стороны, для увеличения периода полувыведения антител осуществляли мутации в Fc-фрагменте антител, которые вводили приматам, не являющимся человеком (Hinton et al. (2004) J Biol Chem 279: 6213-6). Однако применение данного подхода ограничено только до применения в отношении терапевтических антител, и его нельзя экстраполировать на другие терапевтические белки, если только эти белки не слиты с Fc-фрагментами, что также приводит к образованию крупных молекул. Для улучшения периода полувыведения белка разработали ряд химических и рекомбинантных способов, таких как ПЭГилирование и рекомбинантное слияние белка с Fc-доменом IgG или с альбумином (обзор представлен Schellenberger et al. (2009) Nat Biotechnol 21: 1186-1190). Опубликованы данные, что ПЭГилирование белков снижает их активность и вносит вклад в их иммунореактивность.
Белки, слитые с Fc, также применяли для пероральной и легочной доставки, опосредованной FcRn (Low et al., (2005) Human reproduction Jul; 20(7): 1805-13), однако из-за размера слитых молекул по-прежнему возникают аналогичные проблемы, связанные с проникновением в ткани и пониженной специфичностью.
Таким образом, в области техники по-прежнему существует большая потребность в молекулах с высокой аффинностью к FcRn. В частности, необходимы небольшие связывающиеся молекулы, которые, будучи партнером по слиянию, не оказывают негативного влияния на свойства молекул, с которыми они слиты, и не способствуют иммунореактивности.
Краткое изложение сущности изобретения
Данное описание основано на неожиданном обнаружении, что FcRn-связывающие полипептиды в димерной форме демонстрируют существенно улучшенные свойства связывания с FcRn по сравнению с соответствующими FcRn-связывающими полипептидами в мономерной форме. Авторы данного изобретения обнаружили, что связывающие свойства указанных FcRn-связывающих полипептидов в димерной форме улучшаются больше, чем ожидается при простом слиянии двух FcRn-связывающих полипептидов.
Таким образом, задачей данного изобретения является обеспечение новых FcRn-связывающих агентов.
Задачей данного изобретения также является обеспечение таких агентов для применения в модификации фармакокинетических и/или фармакодинамических свойств биомолекулы, например лекарственного средства.
Задачей данного изобретения также является обеспечение таких агентов для применения в качестве терапевтических агентов как таковых, в отдельности или в составе комбинированной терапии.
Задачей данного изобретения является обеспечение молекулы, позволяющей эффективно связываться с FcRn, при этом уменьшающей упомянутые выше или другие недостатки существующей терапии.
Эти и другие задачи, очевидные специалисту в области техники на основании данного описания, решаются в различных аспектах изобретения, изложенного в прилагаемой формуле и описанного в общем виде в данном документе.
Так, первым аспектом изобретения является обеспечение димерной формы полипептида, связывающегося с неонатальным Fc рецептором (FcRn), т.е. «FcRn-связывающего димера», содержащей первую мономерную единицу, вторую мономерную единицу и аминокислотный линкер, где каждая из указанных первой и второй мономерных единиц содержит FcRn-связывающий мотив (ВМ), который состоит из аминокислотной последовательности
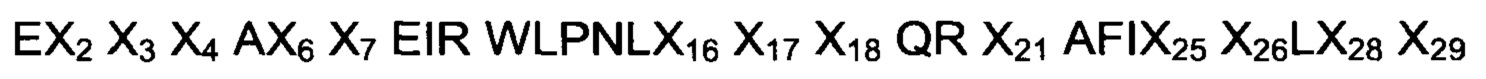
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, G, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х7 выбран из A, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х16 выбран из N и Т;
Х17 выбран из F, W и Y;
Х18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х26 выбран из K и S;
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y; и
Х29 выбран из D и R,
и где указанный FcRn-связывающий димер связывается с FcRn с большей связывающей способностью по сравнению с указанной первой мономерной единицей или указанной второй мономерной единицей в отдельности.
Приведенное выше определение класса FcRn-связывающих мотивов с родственными последовательностями основано на статистическом анализе ряда случайных вариантов полипептидных мономеров с исходным каркасом, которые отобрали благодаря их взаимодействию с FcRn в нескольких различных экспериментах по отбору. Идентифицированный FcRn-связывающий мотив, или «ВМ», соответствует связывающей мишень области исходного каркаса, причем указанная область образует две альфа-спирали в составе белкового домена-трехспирального пучка. В исходном каркасе варьирующие аминокислотные остатки двух спиралей ВМ образуют связывающую поверхность для взаимодействия с константной Fc-частью антител. В данном изобретении случайная вариация остатков связывающей поверхности и последующий отбор вариантов привели к замене способности взаимодействовать с Fc способностью взаимодействовать с FcRn.
В одном воплощении FcRn-связывающий мотив по меньшей мере одной из указанных первой и второй мономерных единиц состоит из аминокислотной последовательности
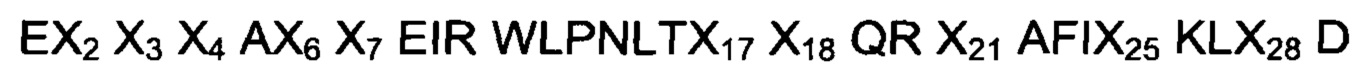
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, G, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х7 выбран из A, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х17 выбран из F, W и Y;
X18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y; и
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y.
В одном воплощении первого аспекта изобретения указанный связывающий мотив по меньшей мере одной из указанных первой и второй мономерных единиц состоит из аминокислотной последовательности
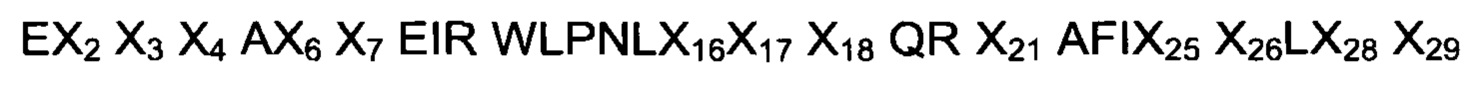
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, G, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из А, Е, F, G, Н, I, K, Q, R, S и V;
Х7 выбран из A, F, Н, K, N, Q, R, S и V;
Х16 выбран из N и Т;
Х17 выбран из F, W и Y;
Х18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из D, Е, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х26 выбран из K и S;
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y; и
Х29 выбран из D и R.
В другом воплощении первого аспекта указанный связывающий мотив по меньшей мере одной из указанных первой и второй мономерных единиц состоит из аминокислотной последовательности, где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из А, Е, F, G, Н, I, K, Q, R и S;
Х7 выбран из A, F, Н, K, N, Q, R, S и V;
X16 выбран из N и Т;
Х17 выбран из F и Y;
X18 представляет собой D;
Х21 представляет собой V;
Х25 выбран из D, Е, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х26 выбран из K и S;
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V и W, и
Х29 выбран из D и R.
В другом воплощении первого аспекта указанный ВМ по меньшей мере одной из указанных первой и второй мономерных единиц состоит из аминокислотной последовательности, выбранной из
(I) 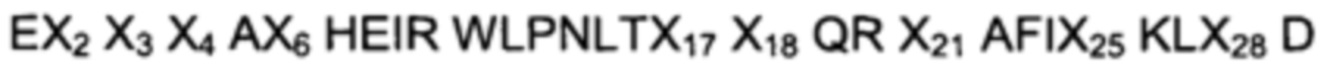
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, G, Н, K, L, М, N, Q, R, S, Т, V и Y;
Х4 выбран из A, D, Е, F, G, I, K, L, N, Q, R, S, Т, V и Y;
Х6 выбран из A, G, K, R, S и V;
Х17 выбран из F, W и Y;
Х18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из D, G, Н, K, L, N, R, V и W;
Х28 выбран из A, D, Е, Н, K, L, N, Q, R, S, Т, W и Y;
и
II) аминокислотной последовательности, идентичной указанной последовательности по меньшей мере на 96%.
В еще одном воплощении указанного аспекта указанный ВМ в последовательности (I) состоит из аминокислотной последовательности, выбранной из
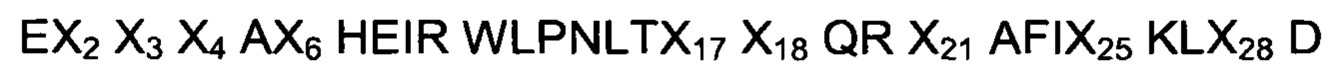
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, N, Q, R, S и W;
Х3 выбран из D, Е, G, Н, K, М, N, Q, S и Т;
Х4 выбран из A, D, Е, G, N, Q, R, S, Т, V и Y;
Х6 выбран из A, G, S и V;
Х17 выбран из F, W и Y;
X18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из D, G, Н, K, L, N, R и V; и
Х28 выбран из А, Е, Н, L, N, Q, R, S, Т, W и Y.
Как известно специалисту в области техники, функция любого полипептида, включая способность предложенного в данном изобретении димера связывать FcRn, зависит от третичной структуры полипептида. Таким образом, возможно произвести минимальные изменения в последовательности аминокислот полипептида, не влияя на его функцию. Таким образом, изобретение охватывает варианты FcRn-связывающего димера, например варианты, в которых по меньшей мере одна из указанных первой и второй мономерных единиц модифицирована, но FcRn-связывающие свойства сохранены.
Таким образом, как указано выше, данное описание также охватывает FcRn-связывающий димер, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит FcRn-связывающий мотив (ВМ), содержащий аминокислотную последовательность, идентичную на 96% или более полипептиду, определенному в i).
В некоторых воплощениях такие изменения могут быть произведены во всех положениях последовательностей ВМ, как описано здесь. В других воплощениях такие изменения могут быть произведены только в невариабельных положениях, также обозначаемых как каркасные аминокислотные остатки. В таких случаях запрещены изменения в вариабельных положениях, то есть в положениях, обозначенных как «X» в последовательности (I). Например, допускается, что аминокислотный остаток, относящийся к определенной функциональной группе аминокислотных остатков (например, гидрофобных, гидрофильных, полярных и т.д.), может быть заменен другим аминокислотным остатком из той же функциональной группы.
Термин «% идентичности», используемый в данном описании, можно рассчитать, например, следующим образом. Искомую последовательность выравнивают с целевой последовательностью с помощью алгоритма CLUSTAL W (Thompson et al. (1994) Nucleic Acids Research 22: 4673-4680). Сравнение выполняют в окне, соответствующем наиболее короткой из выравниваемых последовательностей. В некоторых случаях наиболее короткая из выравниваемых последовательностей может быть целевой последовательностью. В других случаях искомая последовательность может быть наиболее короткой из выравниваемых последовательностей. Сравнивают аминокислотные остатки в каждом положении и количество положений искомой последовательности, имеющих идентичное соответствие в целевой последовательности, выраженное в процентах, указывают как % идентичности.
В данном документе "Xn" и "Xm" используют, чтобы указать аминокислоты в положениях n и m в последовательности ВМ, как определено выше, где n и m являются целыми числами, указывающими положение аминокислоты в указанной последовательности, считая с N-конца указанной последовательности. Например, Х3 и Х7 указывают аминокислоты в положении 3 и 7, соответственно, от N-конца указанного ВМ.
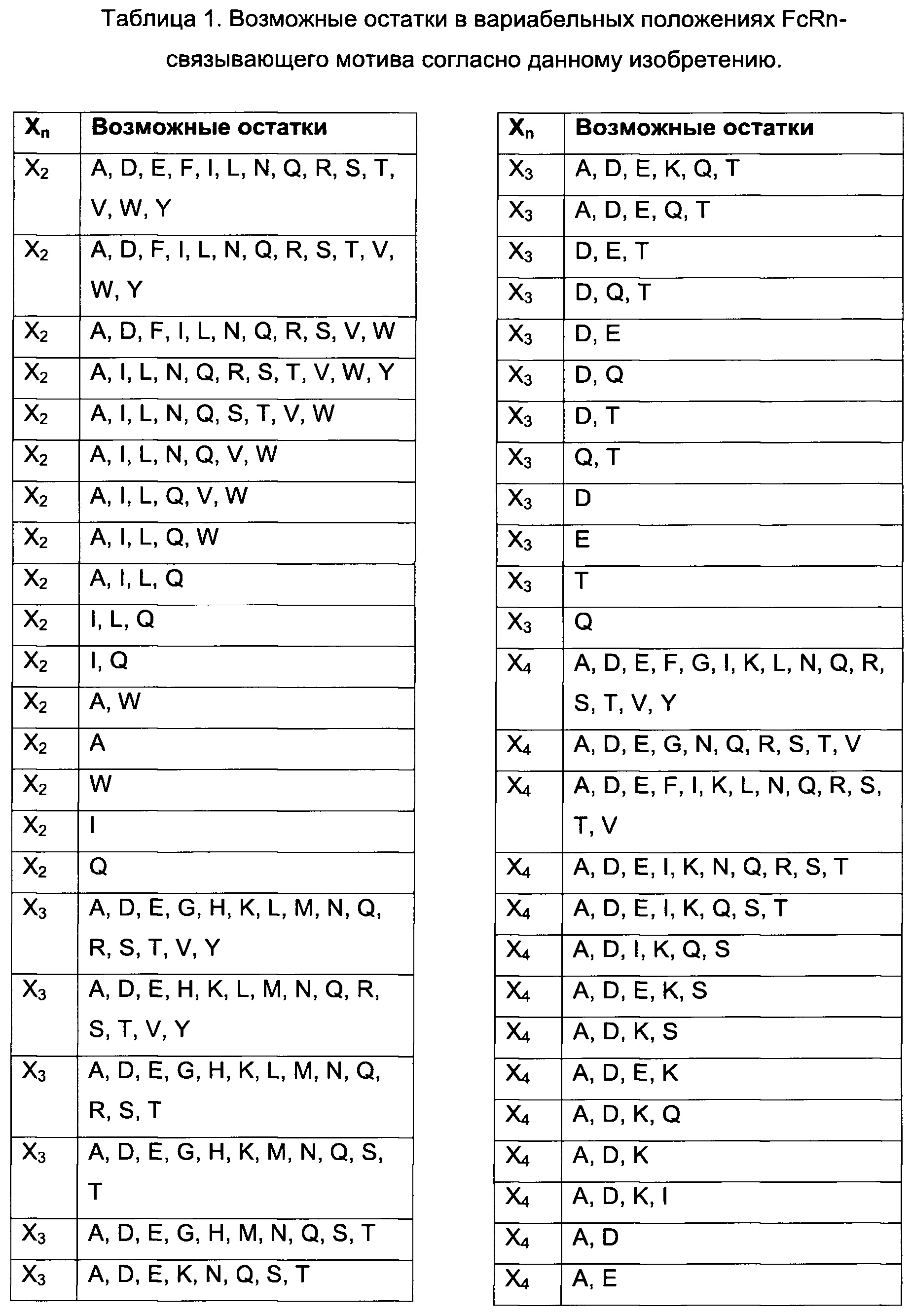
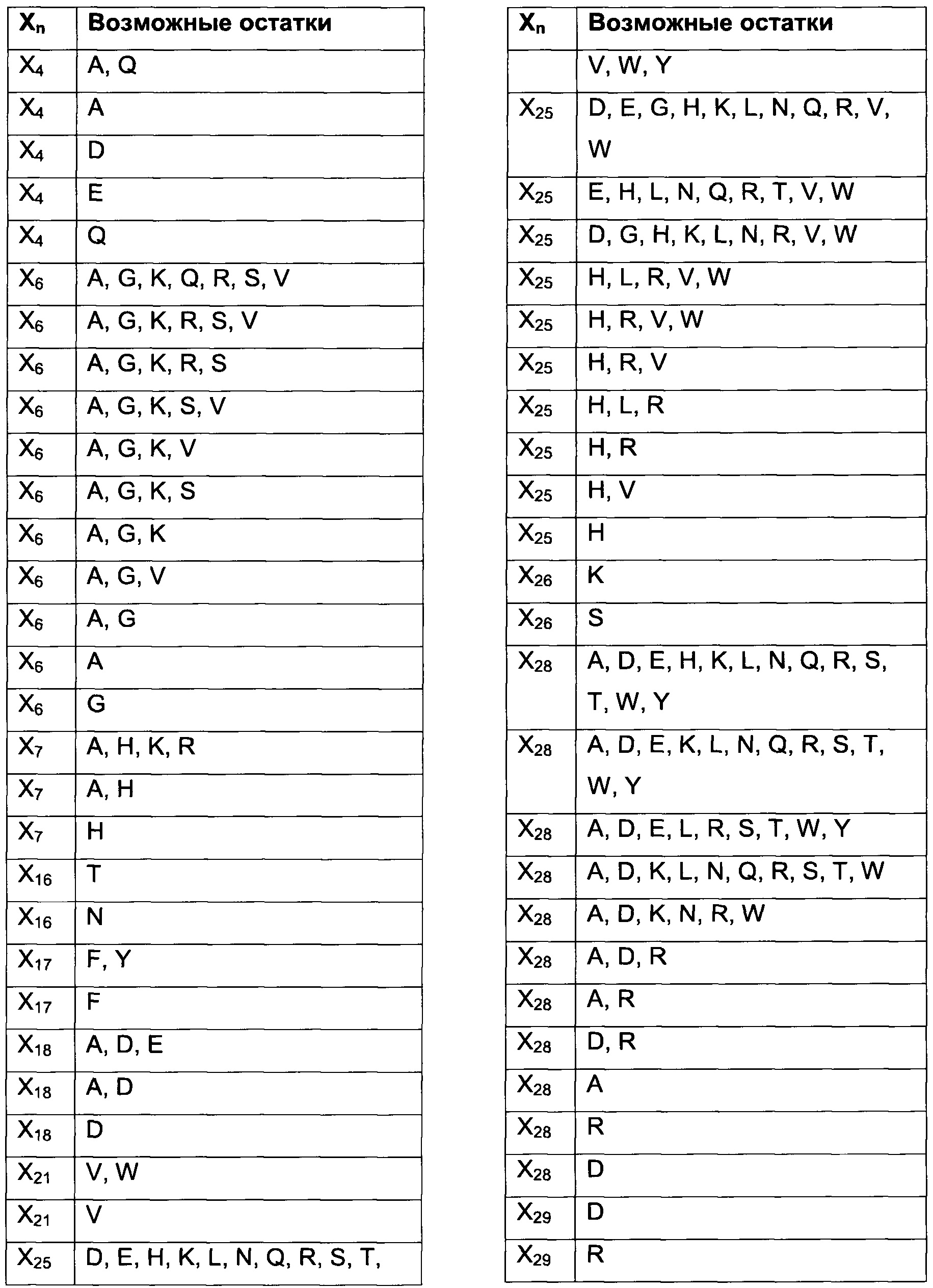
В воплощениях согласно первому аспекту предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит FcRn-связывающий мотив, где Xn независимо выбран из группы возможных остатков согласно Таблице 1. Специалист в области техники поймет, что Xn может быть выбран из любой из указанных групп возможных остатков, и что данный выбор осуществляется независимо от выбора аминокислот в Xm, где n не равно m. Таким образом, любой из перечисленных возможных остатков в положении Xn в Таблице 1 можно независимо скомбинировать с любым из раскрытых здесь возможных остатков в любом другом вариабельном положении в Таблице 1.
Специалист поймет, что Таблицу 1 следует читать следующим образом:
В одном воплощении согласно первому аспекту предложен FcRn-связывающий димер, в котором каждая из указанных первой мономерной единицы и второй мономерной единицы содержит FcRn-связывающий мотив (ВМ), и где аминокислотный остаток «Xn» в ВМ по меньшей мере одной из указанной первой мономерной единицы и указанной второй мономерной единицы независимо выбран из «возможных остатков». Специалист поймет, что аминокислотный остаток «Xn» в ВМ первой мономерной единицы выбран независимо от аминокислотного остатка «Xn» в ВМ второй мономерной единицы. Таким образом, в Таблице 1 представлено несколько конкретных отдельных вариантов первой мономерной единицы и второй мономерной единицы согласно данному изобретению. Например, в одном воплощении предложен FcRn-связывающий димер, содержащий по меньшей мере одну первую или вторую мономерную единицу, где Х2 в ВМ выбран из A, I, L, N, Q, S, Т, V и W, а в другом воплощении предложен FcRn-связывающий димер, содержащий по меньшей мере одну первую или вторую мономерную едииницу, где Х2 в ВМ выбран из A, I, L и Q. Во избежание двусмысленного толкования, указанные первую и вторую мономерные единицы можно свободно комбинировать в других воплощениях. Например, в одном из таких воплощений Х3 выбран из A, D, Е, G, Н, K, L, М, N, Q, R, S и Т, тогда как Х7 выбран из А и Н, а Х25 выбран из Н, L, R, V и W.
*
В одном воплощении предложен FcRn-связывающий димер, где Х6Х7 выбран из АН и GH в по меньшей мере одной из указанных первой и второй мономерных единиц. В одном воплощении Х6Х7 представляет собой АН. В одном воплощении Х6Х7 представляет собой GH. В одном воплощении X17X18 выбран из FD и YD в по меньшей мере одной из указанных первой и второй мономерных единиц. В одном воплощении X17X18 представляет собой FD.
В более конкретном воплощении, определяющем подкласс FcRn-связывающих димеров, последовательность ВМ по меньшей мере одной указанной первой и второй мономерных единиц удовлетворяет по меньшей мере трем из шести условий I-VI:
I. Х6 выбран из A, G, K и S, например, в частности, А;
II. Х7 представляет собой Н;
III. X17 выбран из F и Y, например, в частности, F;
IV. X18 представляет собой D;
V. Х21 выбран из V и W, например, в частности, V;
VI. Х25 выбран из Н и R, например, в частности, Н.
В некоторых примерах связывающего FcRn димера согласно первому аспекту, указанная последовательность удовлетворяет по меньшей мере четырем из шести условий I-VI. Более конкретно, последовательность может удовлетворять по меньшей мере пяти из шести условий I-VI, например всем шести из шести условий I-VI.
В одном воплощении последовательности ВМ указанных первой и второй мономерных единиц являются идентичными. В другом воплощении последовательности ВМ указанных первой и второй мономерных единиц являются различными.
Как подробно описано ниже в экспериментальной части, отбор мономерных единиц FcRn-связывающего полипептида позволил идентифицировать ряд отдельных последовательностей FcRn-связывающих мотивов (ВМ). Эти последовательности составляют отдельные варианты, полезные в качестве первой и второй мономерных единиц, как описано в данном документе. Последовательности отдельных FcRn-связывающих мотивов (ВМ) представлены на Фиг. 1 и соответствуют последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353. Так, в одном воплощении FcRn-связывающего димера согласно первому аспекту, по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353, такой как выбранная из группы, состоящей из SEQ ID NO: 17-352. В одном воплощении указанная последовательность ВМ соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 17-140. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2 и SEQ ID NO: 17-140. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140, такой как группа, состоящая из SEQ NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO.75-77 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73 и SEQ ID NO: 75-77. В другом воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В другом воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В еще одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В еще одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В еще одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В еще одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 75. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 41, группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 44, или группа, состоящая из SEQ ID NO: 41 и SEQ ID NO: 44. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 1 или SEQ ID NO: 23 или SEQ ID NO: 44. В одном воплощении указанная последовательность соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 20 или SEQ ID NO: 41 или SEQ ID NO: 44.
В одном воплощении раскрытого здесь FcRn-связывающего димера обе из указанных первой и второй мономерных единиц содержат ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из одной из групп, указанных выше. В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В одном воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75. В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В другом воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44. В еще одном воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44. В одном конкретном воплощении обе из указанных первой и второй мономерных единиц содержат ВМ, соответствующий последовательности от положения 8 до положения 36 в SEQ ID NO: 1. В одном воплощении указанный ВМ соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 20. В одном воплощении указанный ВМ соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 23. В одном воплощении указанный ВМ соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 41. В одном воплощении указанный ВМ соответствует последовательности от положения 8 до положения 36 в SEQ ID NO: 44.
В некоторых воплощениях данного изобретения ВМ, как определено выше, «является частью» трехспирального пучка белкового домена. Это означает, что последовательность ВМ «встроена» в последовательность или «пересажена» на последовательность исходного домена с трехспиральным пучком, так что ВМ заменяет аналогичный структурный мотив исходного домена. Например, не ограничиваясь какой-либо теорией, предполагают, что ВМ образует две из трех спиралей трехспирального пучка и, таким образом, может заменить такой двухспиральный мотив в составе любого трехспирального пучка. Специалисту в области техники понятно, что замену двух спиралей в трехспиральном пучке домена двумя спиралями ВМ необходимо осуществлять таким образом, чтобы не повлиять на основную структуру полипептида. Таким образом, общая укладка Сα каркаса полипептида согласно данному воплощению изобретения по существу такая же, как укладка белкового домена с трехспиральным пучком, частью которого он является, например, имеет такие же элементы вторичной структуры, расположенные в том же порядке, и т.д. Таким образом, ВМ согласно изобретению, «является частью» домена с трехспиральным пучком, если полипептид согласно данному воплощению аспекта имеет такую же укладку, как и исходный домен, с учетом того, что основные структурные свойства являются общими, например эти свойства обусловливают сходство спектров CD (кругового дихроизма). Специалисту известны другие имеющие значение параметры.
В конкретных воплощениях FcRn-связывающий мотив (ВМ) в по меньшей мере одном из указанных первого и второго мономеров является, таким образом, частью белкового домена с трехспиральным пучком. Например, ВМ может по существу составлять две альфа-спирали с соединительной петлей, в составе указанного белкового домена-трехспирального пучка. В конкретных воплощениях указанный белковый домен с трехспиральным пучком выбран из доменов бактериальных белков-рецепторов. Неограничивающими примерами таких доменов являются пять различных трехспиральных доменов белка А из Staphylococcus aureus, такие как домен В и его производные. В некоторых воплощениях белковый домен с трехспиральным пучком представляет собой вариант белка Z, который является производным домена В стафилококкового белка А.
В воплощениях, где FcRn-связывающий мотив, раскрытый в данном документе, является частью белкового домена с трехспиральным пучком, по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать связывающий модуль (BMod), который состоит из аминокислотной последовательности, выбранной из:
III) 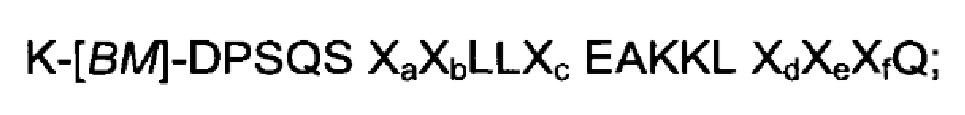
где
[BM] представляет собой FcRn-связывающий мотив, как определено здесь, при условии, что Х29 представляет собой D;
Ха выбран из А и S;
Xb выбран из N и Е;
Хс выбран из A, S и С;
Xd выбран из Е, N и S;
Хе выбран из D, Е и S;
Xf выбран из А и S;
и
IV) аминокислотной последовательности, которая по меньшей мере на 93% идентична последовательности, определенной в (III).
В воплощениях, где FcRn-связывающий мотив, как определено здесь, является частью белкового домена с трехспиральным пучком, по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать связывающий модуль (BMod), который состоит из аминокислотной последовательности, выбранной из:
V) 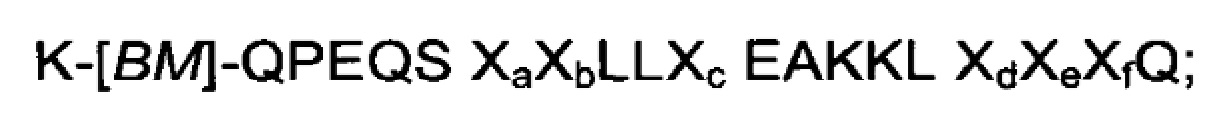
где
[ВМ] представляет собой FcRn-связывающий мотив, как определено здесь, при условии, что Х29 представляет собой R;
Ха выбран из А и S;
Xb выбран из N и Е;
Хс выбран из A, S и С;
Xd выбран из Е, N и S;
Хе выбран из D, Е и S;
Xf выбран из А и S; и
IV) аминокислотной последовательности, которая по меньшей мере на 93% идентична последовательности, определенной в (V).
Как обсуждено выше, полипептиды, содержащие незначительные изменения по сравнению с указанными выше аминокислотными последовательностями, которые не влияют в большой степени на их третичную структуру и функцию, также входят в объем данного изобретения. Так, в некоторых воплощениях, последовательность (IV) или последовательность (VI) по меньшей мере на 95%, например по меньшей мере на 97%, идентична последовательности, определенной в (III) и (V), соответственно.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Ха представляет собой А. В альтернативном воплощении Ха в последовательности (III) или (V) представляет собой S. В одном воплощении Ха в последовательности (III) или (V) представляет собой А. В одном воплощении Ха в последовательности ((III) или (V) представляет собой S.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Xb представляет собой N. В одном воплощении Xb в последовательности (III) или (V) представляет собой Е.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Хс представляет собой А. В одном воплощении Хс в последовательности (III) или (V) представляет собой S. В одном воплощении Хс в последовательности (III) или (V) представляет собой С.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Xd представляет собой Е. В одном воплощении Xd в последовательности (III) или (V) представляет собой N. В одном воплощении Xd в последовательности (III) или (V) представляет собой S.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Хе представляет собой D. В одном воплощении Хе в последовательности (III) или (V) представляет собой Е. В одном воплощении Хе в последовательности (III) или (V) представляет собой S.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где XdXe выбрано из ЕЕ, ES, SD, SE и SS. В одном воплощении XdXe в последовательности (III) или (V) представляет собой ES. В одном воплощении XdXe в последовательности (III) или (V) представляет собой SE. В одном воплощении XdXe в последовательности (III) или (V) представляет собой SD.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Xf представляет собой А. В одном воплощении Xf в последовательности (III) или (V) представляет собой S.
В одном воплощении предложен FcRn-связывающий димер, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (III) или (V), где Ха представляет собой А, Xb представляет собой N; Хс представляет собой А и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой А и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой С и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой S и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой С и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой A; XdXe представляет собой ND и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой A; XdXe представляет собой ND и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой С; XdXe представляет собой ND и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой S; XdXe представляет собой ND и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой С; XdXe представляет собой ND и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой A; XdXe представляет собой SE и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой A; XdXe представляет собой SE и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой С; XdXe представляет собой SE и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой S; XdXe представляет собой SE и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой С; XdXe представляет собой SE и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой A; XdXe представляет собой ES и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой A; XdXe представляет собой ES и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой С; XdXe представляет собой ES и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой S; XdXe представляет собой ES и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой С; XdXe представляет собой ES и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой A; XdXe представляет собой SD и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой A; XdXe представляет собой SD и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой А; Xb представляет собой N; Хс представляет собой С; XdXe представляет собой SD и Xf представляет собой А.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой S; XdXe представляет собой SD и Xf представляет собой S.
В одном воплощении в последовательности (III) или (V) Ха представляет собой S; Xb представляет собой Е; Хс представляет собой С; XdXe представляет собой SD и Xf представляет собой S.
В одном воплощении FcRn-связывающего димера согласно первому аспекту по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod согласно последовательности (III), соответствующей последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353, SEQ ID NO: 358 и SEQ ID NO: 360-364. Таким образом, в одном воплощении FcRn-связывающего димера согласно первому аспекту по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-352 и SEQ ID NO: 360-364. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140, SEQ ID NO: 353, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-140 и SEQ ID NO: 360-364. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-140, SEQ ID NO: 358 и SEQ ID NO: 360-364. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125, SEQ ID NO: 127-140, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125, SEQ ID NO: 127-140 и SEQ ID NO: 360-364. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77, SEQ ID NO: 353, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 360-364. В другом воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73, SEQ ID NO: 75-77, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 360-364. В другом воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В другом воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 77, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 360-364. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 360-364. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 75 и SEQ ID NO: 360-364. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75. В еще одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 75, SEQ ID NO: 358, SEQ ID NO: 361 и SEQ ID NO: 364, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 75, SEQ ID NO: 361 и SEQ ID NO: 364. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 75.
В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 360, SEQ ID NO: 362 и SEQ ID NO: 363, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 360 и SEQ ID NO: 362; группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 44, SEQ ID NO: 360 и SEQ ID NO: 363; или группа, состоящая из SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 362 и SEQ ID NO: 363.
В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 358, SEQ ID NO: 361 и SEQ ID NO: 363, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 361 и SEQ ID NO: 363. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 1 или SEQ ID NO: 23 или SEQ ID NO: 44. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 20 или SEQ ID NO: 41 или SEQ ID NO: 44. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 360 или SEQ ID NO: 362 или SEQ ID NO: 363.
В одном воплощении FcRn-связывающего димера согласно данному описанию обе из указанных первой и второй мономерных единиц содержат BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из одной из групп, указанных выше. В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 358 и SEQ ID NO: 360-364. В одном воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 75 и SEQ ID NO: 360-364. В одном воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75. В другом воплощении указанная группа состоит из SEQ ID NO: 360-364. В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В другом воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44. В одном воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 360, SEQ ID NO: 362 и SEQ ID NO: 363. В другом воплощении указанная группа состоит из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44. В другом воплощении указанная группа состоит из SEQ ID NO: 360, SEQ ID NO: 362 и SEQ ID NO: 363. В одном конкретном воплощении обе из указанных первой и второй мономерных единиц содержат BMod, соответствующий последовательности от положения 7 до положения 55 в SEQ ID NO: 1 или SEQ ID NO: 358. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 20 или SEQ ID NO: 360. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 23 или SEQ ID NO: 361. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 41 или SEQ ID NO: 362. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 44 или SEQ ID NO: 363. В одном воплощении указанный BMod соответствует последовательности от положения 7 до положения 55 в SEQ ID NO: 75 или SEQ ID NO: 364.
Также в дополнительном воплощении предложен FcRn-связывающий димер, как определено выше, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
VII) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и Хс выбран из A, S и С; и
VIII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (VII).
В другом воплощении предложен FcRn-связывающий димер, как определено выше, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
IX) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и Хс выбран из A, S и С; и
X) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (IX).
В одном воплощении Хс в последовательности, определенной в (IX), представляет собой S.
В альтернативном варианте предложен FcRn-связывающий димер, как определено выше, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XI) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и Хс выбран из А и С; и
XII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XI).
Как обсуждено выше, полипептиды, имеющие незначительные изменения по сравнению с указанными выше аминокислотными последовательностями, которые не влияют в большой степени на их третичную структуру и функцию, также входят в объем данного изобретения. Так, в некоторых воплощениях FcRn-связывающий димер, как определено выше, может содержать последовательность (VIII), (X) или (XII), которая по меньшей мере на 96%, например по меньшей мере на 98% идентична последовательности, определенной в (VII), (IX) или (XI), соответственно.
В некоторых воплощениях FcRn-связывающего димера по меньшей мере одна из указанных первой и второй мономерных единиц может содержать аминокислотную последовательность, выбранную из


где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше.
В одном воплощении по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать аминокислотную последовательность, выбранную из:
XIII) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и
XIV) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XIII).
В одном воплощении последовательность (XIII) выбрана из группы, состоящей из SEQ ID NO: 354-357, в частности, выбрана из группы, состоящей из SEQ ID NO: 354 и 357.
В одном воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XIII), выбранную из группы, состоящей из SEQ ID NO: 354-357, в частности, выбрана из группы, состоящей из SEQ ID NO: 354 и 357. В одном воплощении указанная последовательность (XIII) представляет собой SEQ ID NO: 354 в обеих указанных первой и второй мономерных единицах. В одном воплощении указанная последовательность (XIII) представляет собой SEQ ID NO: 357 в обеих указанных первой и второй мономерных единицах.
В одном воплощении по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать аминокислотную последовательность, выбранную из:
XV) 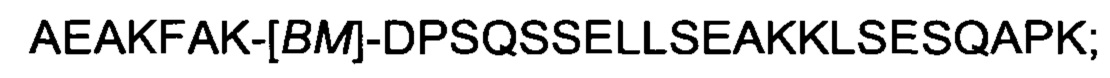
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и
XVI) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XV).
В одном воплощении последовательность (XV) выбрана из группы, состоящей из SEQ ID NO: 365-367. В одном воплощении последовательность (XV) представляет собой SEQ ID NO: 365, SEQ ID NO: 366 или SEQ ID NO: 367.
В одном воплощении по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать аминокислотную последовательность, выбранную из:
XVII) 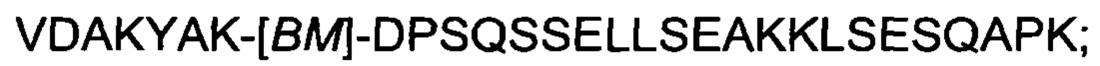
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и
XVIII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XVII).
В одном воплощении последовательность (XVII) выбрана из группы, состоящей из SEQ ID NO: 360-364. В одном воплощении последовательность (XVII) представляет собой SEQ ID NO: 360, SEQ ID NO: 361. SEQ ID NO: 362, SEQ ID NO: 363 или SEQ ID NO: 364.
В одном воплощении по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать аминокислотную последовательность, выбранную из:
XIX) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и
XX) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XIX).
В одном воплощении последовательность (XIX) представляет собой SEQ ID NO: 359.
В одном воплощении по меньшей мере одна из указанных первой и второй мономерных единиц FcRn-связывающего димера может содержать аминокислотную последовательность, выбранную из:
XXI) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено выше, и
XXII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XXI).
И вновь, полипептиды, имеющие незначительные изменения по сравнению с указанными выше аминокислотными последовательностями, которые не влияют в большой степени на их третичную структуру и функцию, также входят в объем данного изобретения. Так, в некоторых воплощениях FcRn-связывающий димер, как определено выше, может содержать последовательность (XIV), (XVI), (XVIII), (XX) или (XXII), которая по меньшей мере на 96%, например по меньшей мере на 98% идентична последовательности, определенной в (XIII), (XV), (XVII), (XIX) или (XXI), соответственно.
В одном воплощении FcRn-связывающего димера согласно первому аспекту по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1-353, такой как из группа, состоящая из SEQ ID NO: 17-352. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 17-140. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1-2 и SEQ ID NO: 17-140. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140, такой как группа, состоящая из SEQ NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73 и SEQ ID NO: 75-77. В другом воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В другом воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77. В еще одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77. В еще одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В еще одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75. В еще одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 75. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 41, группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 44, или группа, состоящая из SEQ ID NO: 41 и SEQ ID NO: 44. В одном воплощении указанная последовательность (XXI) представляет собой последовательность, выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 1 или представляет собой SEQ ID NO: 20, или представляет собой SEQ ID NO: 23, или представляет собой SEQ ID NO: 41, или представляет собой SEQ ID NO: 44.
В одном воплощении FcRn-связывающего димера согласно данному описанию обе из указанных первой и второй мономерных единиц содержат последовательность (XXI) или (XIII), выбранную из одной из групп, указанных выше. В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 354 и SEQ ID NO: 357, как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 354 и SEQ ID NO: 357, как группа, состоящая из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44 или группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 354 и SEQ ID NO: 357.
В одном воплощении FcRn-связывающего димера, как определено здесь, обе из указанных первой и второй мономерных единиц содержат последовательность (XIII), (XV), (XVII), (XIX) или (XXI), выбранную из одной из групп, указанных выше.
В одном воплощении указанная группа состоит из SEQ ID NO: 1, SEQ ID NO: 20; SEQ ID NO: 23, SEQ ID: 41; SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 354, SEQ ID NO: 357 и SEQ ID NO: 360-367, как группа, состоящая из SEQ ID NO: 20; SEQ ID NO: 23, SEQ ID: 41; SEQ ID NO: 44, SEQ ID NO: 75, SEQ ID NO: 357 и SEQ ID NO: 360-367, как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 357, SEQ ID NO: 360, SEQ ID NO: 362, SEQ ID NO: 363, SEQ ID NO: 365, SEQ ID NO: 366 и SEQ ID NO: 367, как группа, состоящая из SEQ ID NO: 357, SEQ ID NO: 360, SEQ ID NO: 362, SEQ ID NO: 363, SEQ ID NO: 365, SEQ ID NO: 366 и SEQ ID NO: 367. В одном конкретном воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44. В одном конкретном воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XXI), соответствующую SEQ ID NO: 1. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 20. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 23. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 41. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 44. В одном воплощении указанная последовательность (XXI) представляет собой SEQ ID NO: 75.
В другом воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XIII), соответствующую SEQ ID NO: 354. В одном конкретном воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XIX), соответствующую SEQ ID NO: 360. В одном воплощении указанная последовательность (XIX) представляет собой SEQ ID NO: 361. В одном воплощении указанная последовательность (XIX) представляет собой SEQ ID NO: 362. В одном воплощении указанная последовательность (XIX) представляет собой SEQ ID NO: 363. В одном воплощении указанная последовательность (XIX) представляет собой SEQ ID NO: 364. В другом конкретном воплощении обе из указанных первой и второй мономерных единиц содержат последовательность (XV), соответствующую SEQ ID NO: 365. В одном воплощении указанная последовательность (XV) представляет собой SEQ ID NO: 366. В одном воплощении указанная последовательность (XV) представляет собой SEQ ID NO: 367.
В конкретном воплощении связывающего FcRn димера первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 1 и SEQ ID NO: 23; SEQ ID NO: 1 и SEQ ID NO: 44; SEQ ID NO: 1 и SEQ ID NO: 65; SEQ ID NO: 1 и SEQ ID NO: 75; SEQ ID NO: 1 и SEQ ID NO: 354; SEQ ID NO: 1 и SEQ ID NO: 357; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 23 и SEQ ID NO: 44; SEQ ID NO: 23 и SEQ ID NO: 65; SEQ ID NO: 23 и SEQ ID NO: 75; SEQ ID NO: 23 и SEQ ID NO: 354; SEQ ID NO: 23 и SEQ ID NO: 357; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 44 и SEQ ID NO: 65; SEQ ID NO: 44 и SEQ ID NO: 75; SEQ ID NO: 44 и SEQ ID NO: 354; SEQ ID NO: 44 и SEQ ID NO: 357; SEQ ID NO: 65 и SEQ ID NO: 65; SEQ ID NO: 65 и SEQ ID NO: 75; SEQ ID NO: 65 и SEQ ID NO: 354; SEQ ID NO: 65 и SEQ ID NO: 357; SEQ ID NO: 75 и SEQ ID NO: 354; SEQ ID NO: 75 и SEQ ID NO: 357; SEQ ID NO: 354 и SEQ ID NO: 354; SEQ ID NO: 354 и SEQ ID NO: 357; или SEQ ID NO: 357 и SEQ ID NO: 357, соответственно. В одном воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 1 и SEQ ID NO: 23; SEQ ID NO: 1 и SEQ ID NO: 44; SEQ ID NO: 1 и SEQ ID NO: 354; SEQ ID NO: 1 и SEQ ID NO: 357; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 23 и SEQ ID NO: 44; SEQ ID NO: 23 и SEQ ID NO: 354; SEQ ID NO: 23 и SEQ ID NO: 357; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 44 и SEQ ID NO: 354; SEQ ID NO: 44 и SEQ ID NO: 357; SEQ ID NO: 354 и SEQ ID NO: 354; SEQ ID NO: 354 и SEQ ID NO: 357; или SEQ ID NO: 357 и SEQ ID NO: 357, соответственно. В другом воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 354 и SEQ ID NO: 354; или SEQ ID NO: 357 и SEQ ID NO: 357, соответственно. В еще одном воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 44 и SEQ ID NO: 44; или SEQ ID NO: 357 и SEQ ID NO: 357, соответственно.
В конкретном воплощении FcRn-связывающего димера первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 1 и SEQ ID NO: 20; SEQ ID NO: 1 и SEQ ID NO: 23; SEQ ID NO: 1 и SEQ ID NO: 41; SEQ ID NO: 1 и SEQ ID NO: 44; SEQ ID NO: 1 и SEQ ID NO: 65; SEQ ID NO: 1 и SEQ ID NO: 75; SEQ ID NO: 1 и SEQ ID NO: 354; SEQ ID NO: 1 и SEQ ID NO: 357; SEQ ID NO: 1 и SEQ ID NO: 365; SEQ ID NO: 1 и SEQ ID NO: 366; SEQ ID NO: 1 и SEQ ID NO: 367; SEQ ID NO: 20 и SEQ ID NO: 20; SEQ ID NO: 20 и SEQ ID NO: 23; SEQ ID NO: 20 и SEQ ID NO: 41; SEQ ID NO: 20 и SEQ ID NO: 44; SEQ ID NO: 20 и SEQ ID NO: 357; SEQ ID NO: 20 и SEQ ID NO: 365; SEQ ID NO: 20 и SEQ ID NO: 366; SEQ ID NO: 20 и SEQ ID NO: 367; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 23 и SEQ ID NO: 41; SEQ ID NO: 23 и SEQ ID NO: 44; SEQ ID NO: 23 и SEQ ID NO: 65; SEQ ID NO: 23 и SEQ ID NO: 75; SEQ ID NO: 23 и SEQ ID NO: 354; SEQ ID NO: 23 и SEQ ID NO: 357; SEQ ID NO: 23 и SEQ ID NO: 365; SEQ ID NO: 23 и SEQ ID NO: 366; SEQ ID NO: 23 и SEQ ID NO: 367; SEQ ID NO: 41 и SEQ ID NO: 41; SEQ ID NO: 41 и SEQ ID NO: 44; SEQ ID NO: 41 и SEQ ID NO: 357; SEQ ID NO: 41 и SEQ ID NO: 365; SEQ ID NO: 41 и SEQ ID NO: 366; SEQ ID NO: 41 и SEQ ID NO: 367; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 44 и SEQ ID NO: 65; SEQ ID NO: 44 и SEQ ID NO: 75; SEQ ID NO: 44 и SEQ ID NO: 354; SEQ ID NO: 44 и SEQ ID NO: 357; SEQ ID NO: 44 и SEQ ID NO: 365; SEQ ID NO: 44 и SEQ ID NO: 366; SEQ ID NO: 44 и SEQ ID NO: 367; SEQ ID NO: 65 и SEQ ID NO: 65; SEQ ID NO: 65 и SEQ ID NO: 75; SEQ ID NO: 65 и SEQ ID NO: 354; SEQ ID NO: 65 и SEQ ID NO: 357; SEQ ID NO: 75 и SEQ ID NO: 354; SEQ ID NO: 75 и SEQ ID NO: 357; SEQ ID NO: 354 и SEQ ID NO: 354; SEQ ID NO: 354 и SEQ ID NO: 357; SEQ ID NO: 357 и SEQ ID NO: 357; SEQ ID NO: 357 и SEQ ID NO: 365; SEQ ID NO: 357 и SEQ ID NO: 366; SEQ ID NO: 357 и SEQ ID NO: 367; SEQ ID NO: 365 и SEQ ID NO: 365; SEQ ID NO: 365 и SEQ ID NO: 366; SEQ ID NO: 365 и SEQ ID NO: 367; SEQ ID NO: 366 и SEQ ID NO: 366; SEQ ID NO: 366 и SEQ ID NO: 367; или SEQ ID NO: 367 и SEQ ID NO: 367, соответственно. В одном воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 1 и SEQ ID NO: 20; SEQ ID NO: 1 и SEQ ID NO: 23; SEQ ID NO: 1 и SEQ ID NO: 41; SEQ ID NO: 1 и SEQ ID NO: 44; SEQ ID NO: 1 и SEQ ID NO: 354; SEQ ID NO: 1 и SEQ ID NO: 357; SEQ ID NO: 1 и SEQ ID NO: 365; SEQ ID NO: 1 и SEQ ID NO: 366; SEQ ID NO: 1 и SEQ ID NO: 367; SEQ ID NO: 20 и SEQ ID NO: 20; SEQ ID NO: 20 и SEQ ID NO: 23; SEQ ID NO: 20 и SEQ ID NO: 41; SEQ ID NO: 20 и SEQ ID NO: 44; SEQ ID NO: 20 и SEQ ID NO: 357; SEQ ID NO: 20 и SEQ ID NO: 365; SEQ ID NO: 20 и SEQ ID NO: 366; SEQ ID NO: 20 и SEQ ID NO: 367; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 23 и SEQ ID NO: 41; SEQ ID NO: 23 и SEQ ID NO: 44; SEQ ID NO: 23 и SEQ ID NO: 354; SEQ ID NO: 23 и SEQ ID NO: 357; SEQ ID NO: 23 и SEQ ID NO: 365; SEQ ID NO: 23 и SEQ ID NO: 366; SEQ ID NO: 23 и SEQ ID NO: 367; SEQ ID NO: 41 и SEQ ID NO: 41; SEQ ID NO: 41 и SEQ ID NO: 44; SEQ ID NO: 41 и SEQ ID NO: 357; SEQ ID NO: 41 и SEQ ID NO: 365; SEQ ID NO: 41 и SEQ ID NO: 366; SEQ ID NO: 41 и SEQ ID NO: 367; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 44 и SEQ ID NO: 354; SEQ ID NO: 44 и SEQ ID NO: 357; SEQ ID NO: 44 и SEQ ID NO: 365; SEQ ID NO: 44 и SEQ ID NO: 366; SEQ ID NO: 44 и SEQ ID NO: 367; SEQ ID NO: 354 и SEQ ID NO: 354; SEQ ID NO: 354 и SEQ ID NO.357; SEQ ID NO: 357 и SEQ ID NO: 357; SEQ ID NO: 357 и SEQ ID NO: 365; SEQ ID NO: 357 и SEQ ID NO: 366; SEQ ID NO: 357 и SEQ ID NO: 367; SEQ ID NO: 365 и SEQ ID NO: 365; SEQ ID NO: 365 и SEQ ID NO: 366; SEQ ID NO: 365 и SEQ ID NO: 367; SEQ ID NO: 366 и SEQ ID NO: 366; SEQ ID NO: 366 и SEQ ID NO: 367; или SEQ ID NO: 367 и SEQ ID NO: 367, соответственно. В другом воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 1 и SEQ ID NO: 1; SEQ ID NO: 20 и SEQ ID NO: 20; SEQ ID NO: 23 и SEQ ID NO: 23; SEQ ID NO: 41 и SEQ ID NO: 41; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 354 и SEQ ID NO: 354; SEQ ID NO: 357 и SEQ ID NO: 357; SEQ ID NO: 365 и SEQ ID NO: 365; SEQ ID NO: 366 и SEQ ID NO: 366; или SEQ ID NO: 367 и SEQ ID NO: 367, соответственно. В еще одном воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 20 и SEQ ID NO: 20; SEQ ID NO: 41 и SEQ ID NO: 41; SEQ ID NO: 44 и SEQ ID NO: 44; SEQ ID NO: 357 и SEQ ID NO: 357; SEQ ID NO: 365 и SEQ ID NO: 365; SEQ ID NO: 366 и SEQ ID NO: 366; или SEQ ID NO: 367 и SEQ ID NO: 367, соответственно.
В еще одном воплощении первая и вторая мономерные единицы содержат SEQ ID NO: 365 и SEQ ID NO: 365; SEQ ID NO: 366 и SEQ ID NO: 366; или SEQ ID NO: 367 и SEQ ID NO: 367, соответственно.
В целях ясности, обозначение первой и второй мономерных единиц, которое используется во всем данном описании, сделано во избежание двусмысленного толкования, для их различения, без намерения указать фактический порядок мономерных единиц в полипептидной цепи FcRn-связывающего димера. Так, например, указанная первая мономерная единица может располагаться в направлении N-конца или C-конца полипептидной цепи относительно указанной второй мономерной единицы.
Как поймет специалист в данной области, в конструкции слитого белка между используемыми функциональными группировками зачастую применяют линкеры. Специалисту в области техники знакомы различные типы линкеров с разными свойствами, такие как гибкие аминокислотные линкеры, жесткие аминокислотные линкеры и расщепляемые аминокислотные линкеры. Линкеры применяли, например, для повышения стабильности или улучшения фолдинга слитых белков, для повышения экспрессии, улучшения биологической активности, обеспечения нацеливания и изменения фармакокинетики слитых белков.
Так, в одном воплощении первого аспекта предложен FcRn-связывающий димер, как определено здесь, в котором указанный линкер выбран из группы, состоящей из гибких аминокислотных линкеров, жестких аминокислотных линкеров и расщепляемых аминокислотных линкеров. В одном воплощении FcRn-связывающего димера согласно данному описанию указанный линкер расположен между первой мономерной единицей и второй мономерной единицей. Специалист в области техники поймет, что присутствие линкера, расположенного между первой мономерной единицей и второй мономерной единицей, не исключает присутствия дополнительных линкеров.
Гибкие линкеры часто используют в области техники, когда требуется определенная степень подвижности или взаимодействия между соединяемыми доменами, и они могут быть особенно полезны в некоторых воплощениях FcRn-связывающего димера. Такие линкеры обычно состоят из небольших неполярных (например, G) или полярных (например, S или Т) аминокислот. Некоторые гибкие линкеры преимущественно состоят из последовательности остатков G и S, например (GGGGS)p и (SSSSG)p. Подбор числа копий "р" позволяет оптимизировать линкер для достижения надлежащего разделения функциональных группировок или поддержания необходимого взаимодействия между группировками. Помимо G и S линкеров в области техники известны другие гибкие линкеры, такие как G и S линкеры, содержащие дополнительные аминокислотные остатки, такие как Т, А, K и Е, для поддержания гибкости, а также полярные аминокислотные остатки для улучшения растворимости.
Дополнительные неиограничивающие примеры линкеров включают 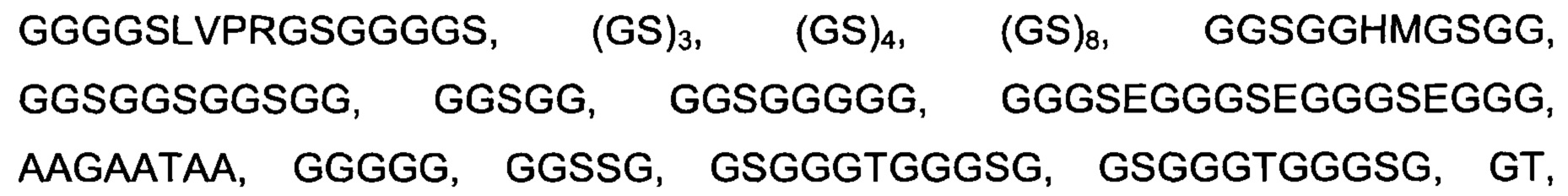
 и
и  Специалисту известны другие подходящие линкеры.
Специалисту известны другие подходящие линкеры.
В одном воплощении указанный линкер представляет собой гибкий линкер, содержащий остатки глицина (G), серина (S) и/или треонина (Т). В одном воплощении указанный линкер имеет общую формулу, выбранную из (GnSm)p и (SnGm)p, где, независимо, n равно от 1 до 7, m равно от 0 до 7, сумма n и m составляет не более 8 и р равно от 1 до 7. В одном воплощении n равно от 1 до 5. В одном воплощении m равно от 0 до 5. В одном воплощении р равно от 1 до 5. В более конкретном воплощении n равно 4, m равно 1 и р равно от 1 до 4. В одном воплощении указанный линкер выбран из группы, состоящей из S4G, (S4G)3 и (S4G)4. В одном воплощении указанный линкер выбран из группы, состоящей из GS, G4S и (G4S)3. В одном конкретном воплощении указанный линкер представляет собой G4S, а в другом воплощении указанный линкер представляет собой (G4S)3.
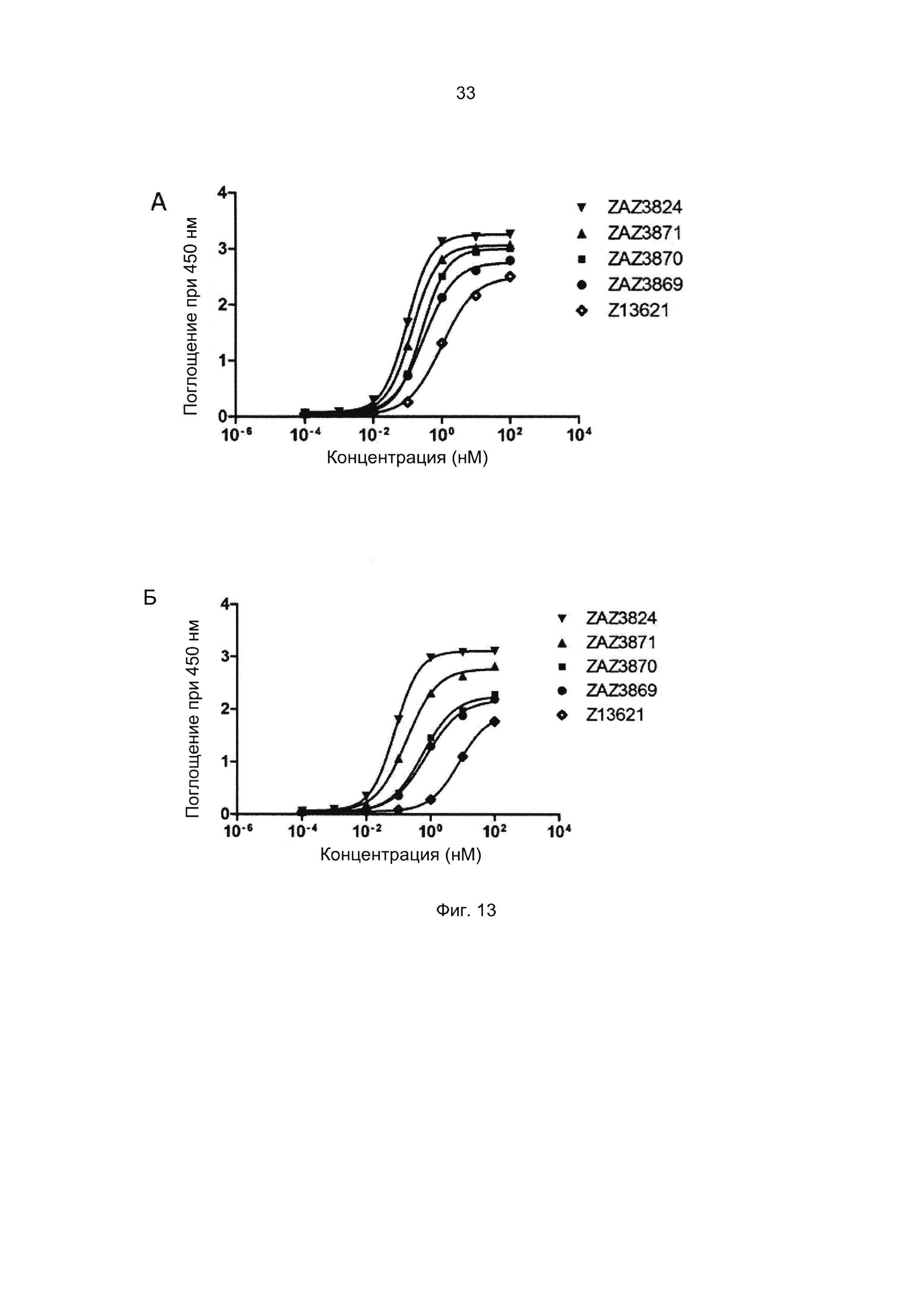
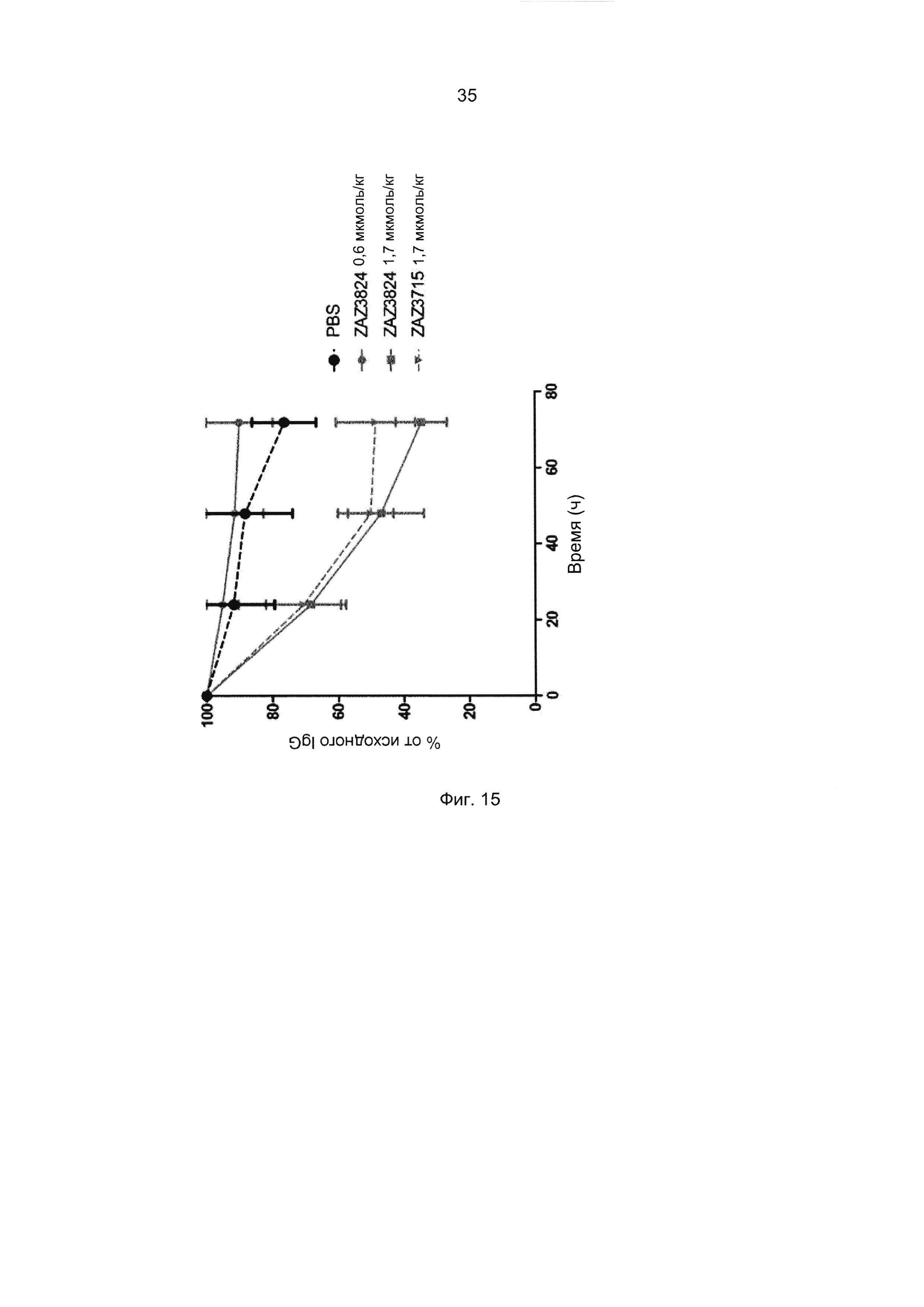
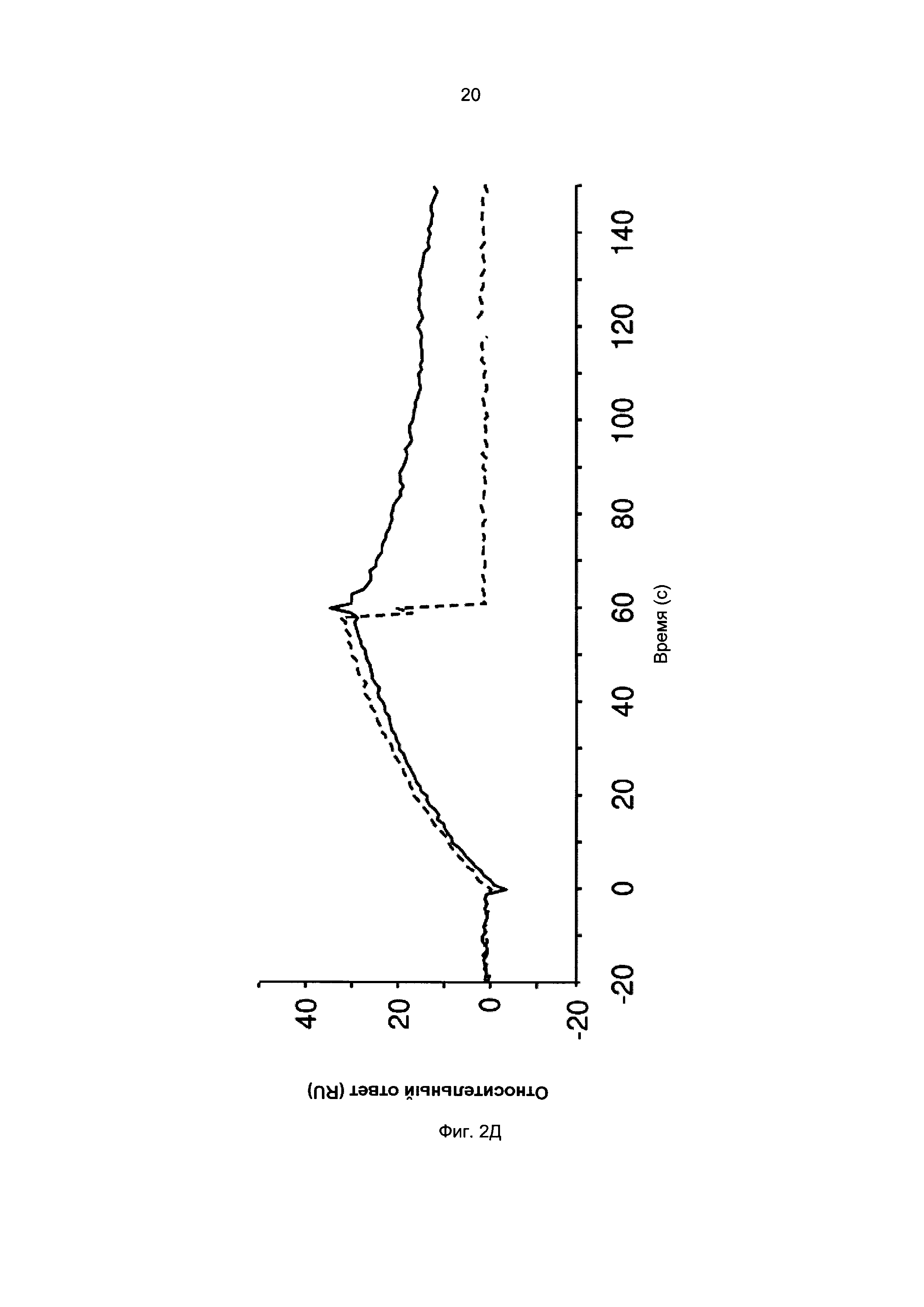
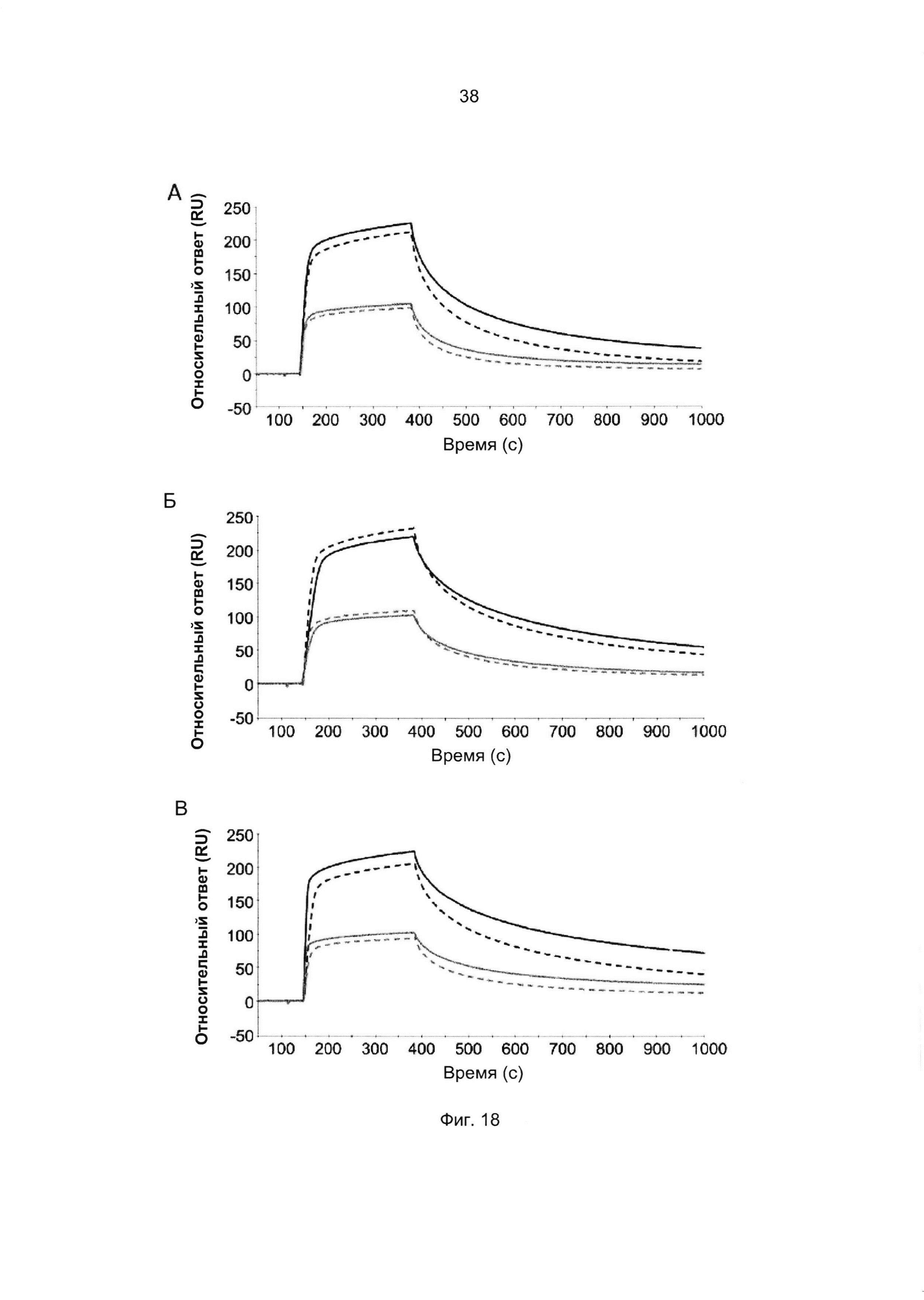
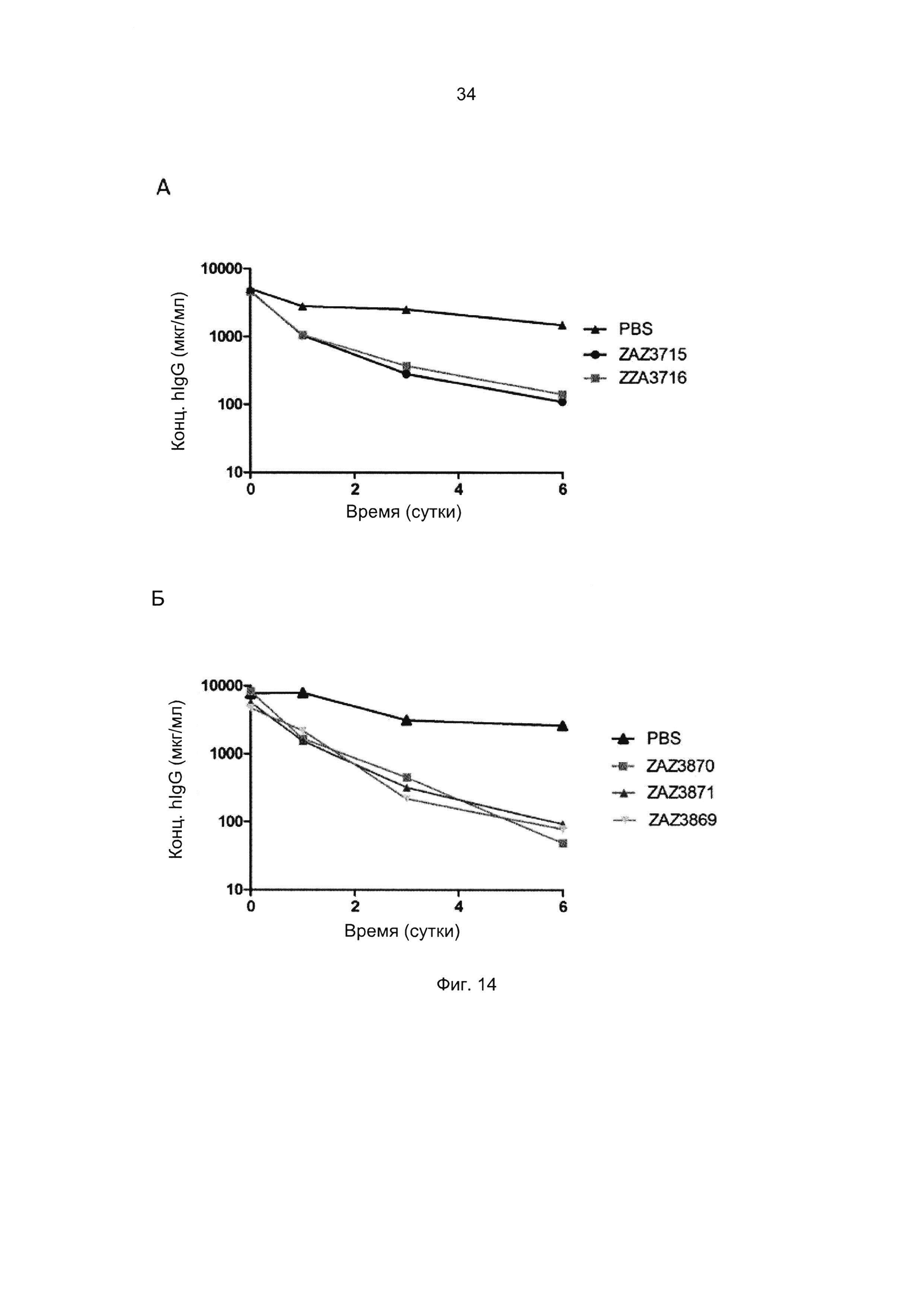
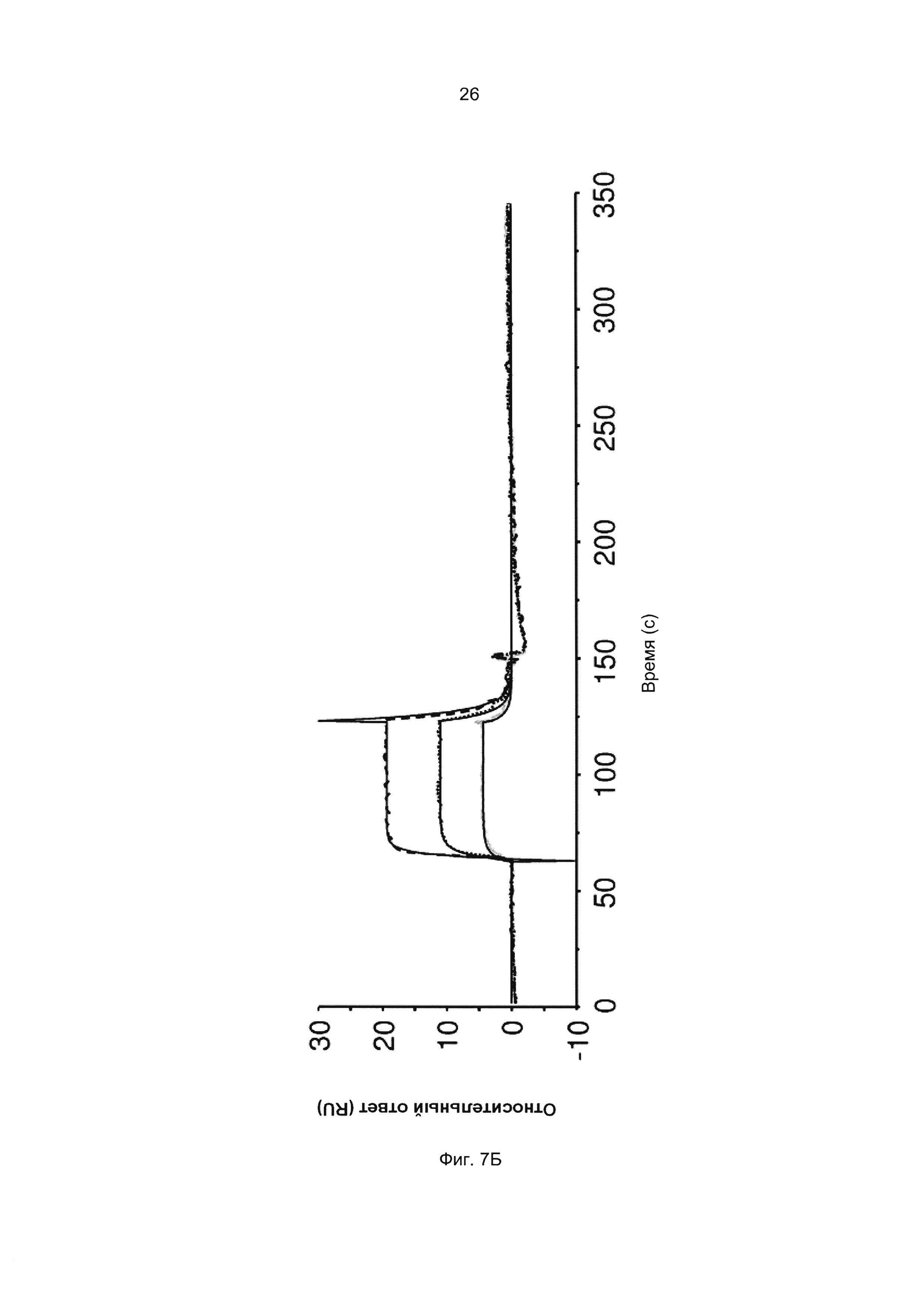
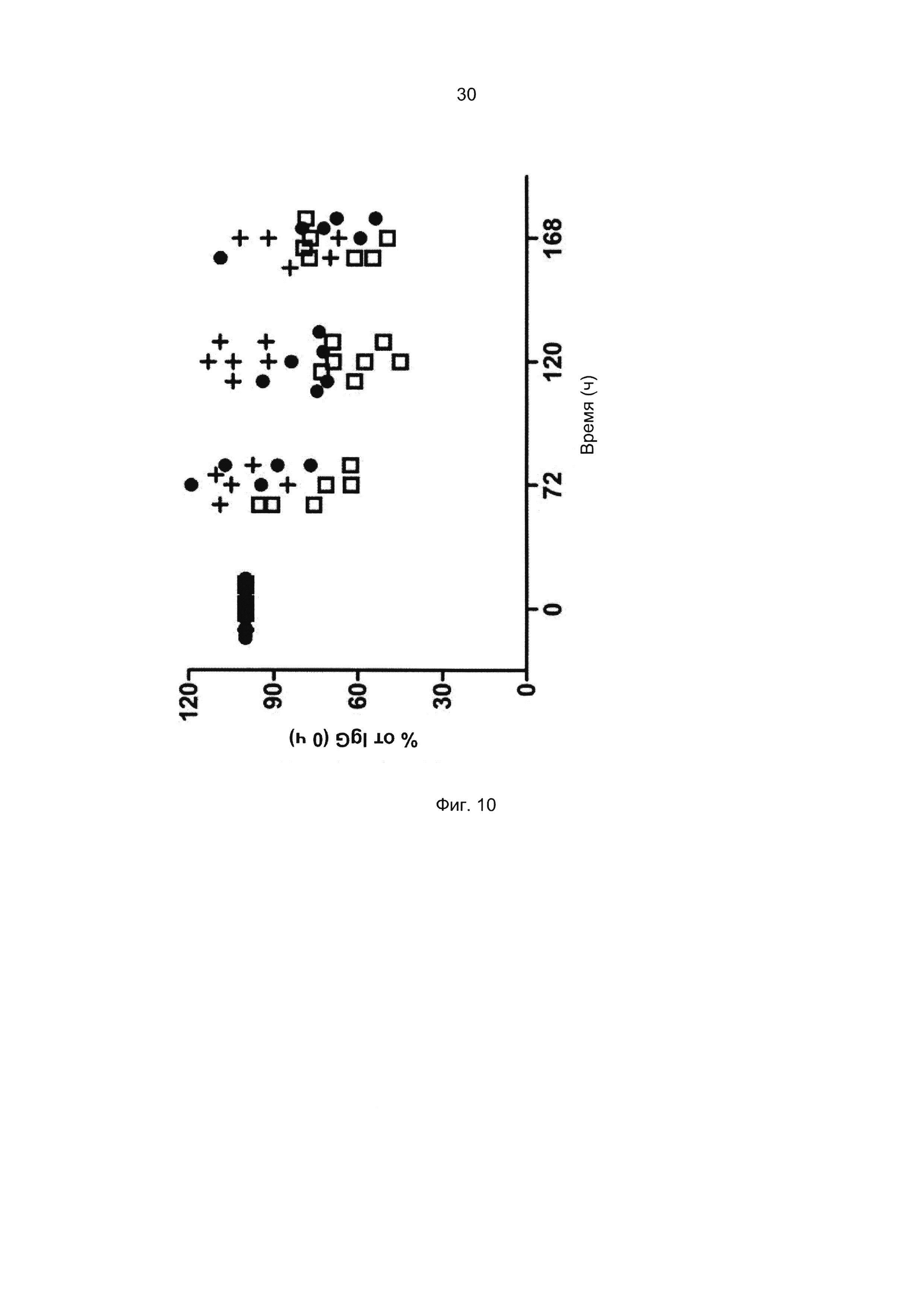
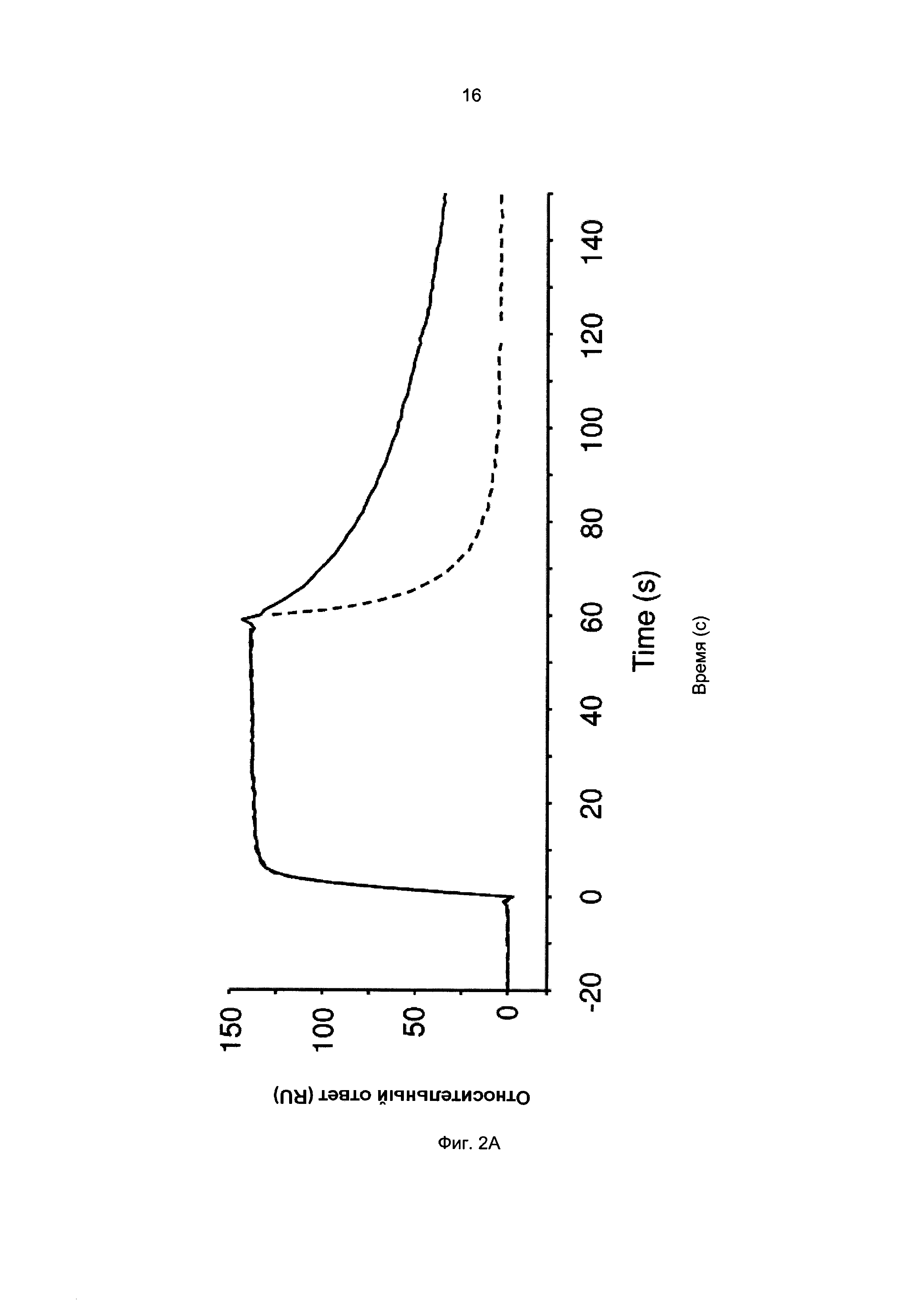
Термины «связывание FcRn» и «аффинность связывания с FcRn», используемые в данном описании, относятся к свойству полипептида, которое можно исследовать, например, используя методику поверхностного плазмонного резонанса (SPR) или твердофазный иммуноферментный анализ (ELISA).
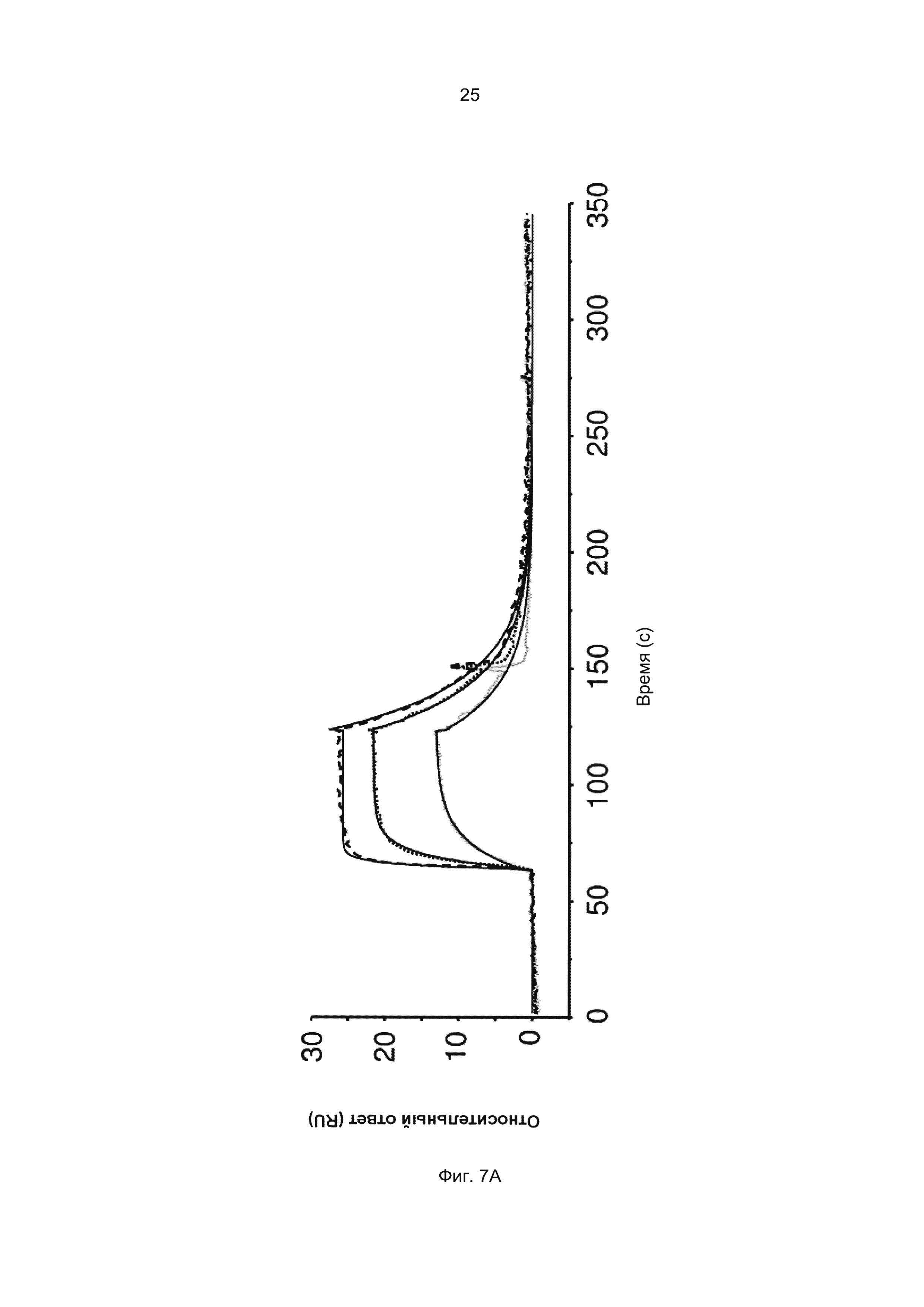
Например, как описано в приведенных ниже примерах, аффинность связывания с FcRn можно исследовать в эксперименте, в котором FcRn или его фрагмент, имеющий правильную укладку, иммобилизован на сенсор-чипе прибора, а образец, содержащий исследуемый полипептид, пропускают над чипом. В альтернативном варианте исследуемый полипептид иммобилизован на сенсор-чипе прибора, а образец, содержащий FcRn или его фрагмент, имеющий правильную укладку, пропускают над чипом. Специалист в области техники может затем интерпретировать результаты, полученные в таких экспериментах, по меньшей мере для качественной оценки аффинности связывания полипептида с FcRn. Если необходимо количественное определение, например определение значения KD взаимодействия, также можно использовать методику поверхностного плазмонного резонанса. Показатели связывания можно, например, определять с помощью аппаратов Biacore (GE Healthcare) или ProteOn XPR 36 (Bio-Rad). FcRn удобно иммобилизовать на сенсор-чипе прибора, а образцы полипептидов, аффинность которых необходимо определить, готовят в виде серии разведений и вводят в случайном порядке. Затем по полученным результатам можно рассчитать значения KD, например, используя модель связывания 1:1 по Лангмюру в программе BIAevaluation 4.1 или другой подходящей программе, предоставляемой производителями оборудования.
В альтернативном варианте, как описано в приведенных ниже примерах, аффинность связывания FcRn можно исследовать в экспериментах, в которых образцы полипептида захватываются антителом, котрым покрыты планшеты для ELISA, и к ним добавляют биотинилированный FcRn с последующим внесением стрептавидина, конъюгированного с пероксидазой хрена (HRP). Добавляют субстрат тетраметилбензидин (ТМВ) и измеряют поглощение при 450 нм с помощью считывающего устройства для многолуночных планшетов, такого как Victor3 (Perkin Elmer). Специалист в области техники может интерпретировать результаты, полученные в таких экспериментах, по меньшей мере для качественной оценки аффинности связывания полипептида с FcRn. Если необходимо количественное определение, например определение значения KD взаимодействия (полумаксимальной эффективной концентрации), также можно использовать ELISA. Ответ полипептидов на серию разведений биотинилированного FcRn измеряют с помощью ELISA, как описано выше. Специалист в области техники затем может интерпретировать результаты, полученные в таких экспериментах и по полученным результатам рассчитать значения KD, например, используя программу GraphPad Prism 5 и нелинейную регрессию.
В альтернативном варианте аффинность к FcRn можно исследовать непосредственно по способности FcRn-связывающего полипептида блокировать связывание IgG с FcRn. Так, специалист в области техники поймет, что способность FcRn-связывающего полипептида блокировать указанное связывание коррелирует со способностью FcRn-связывающего полипептида связываться с FcRn, при условии, что FcRn-связывающий димер взаимодействует с FcRn в той же самой или по меньшей мере частично перекрывающейся области FcRn, что и IgG. Таким образом, чем выше способность полипептида связывать FcRn, тем лучше способность блокировать связывание IgG с FcRn.
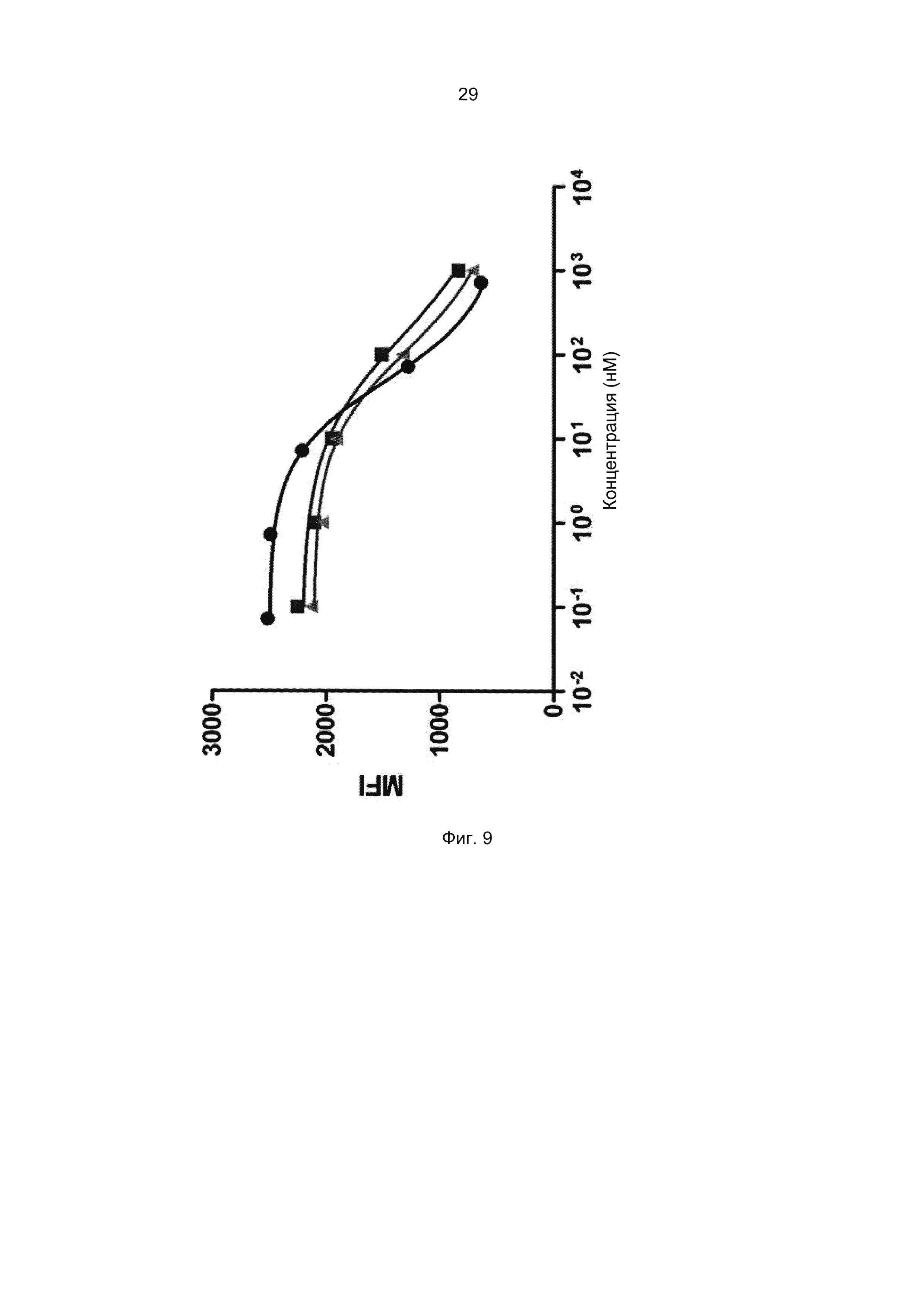
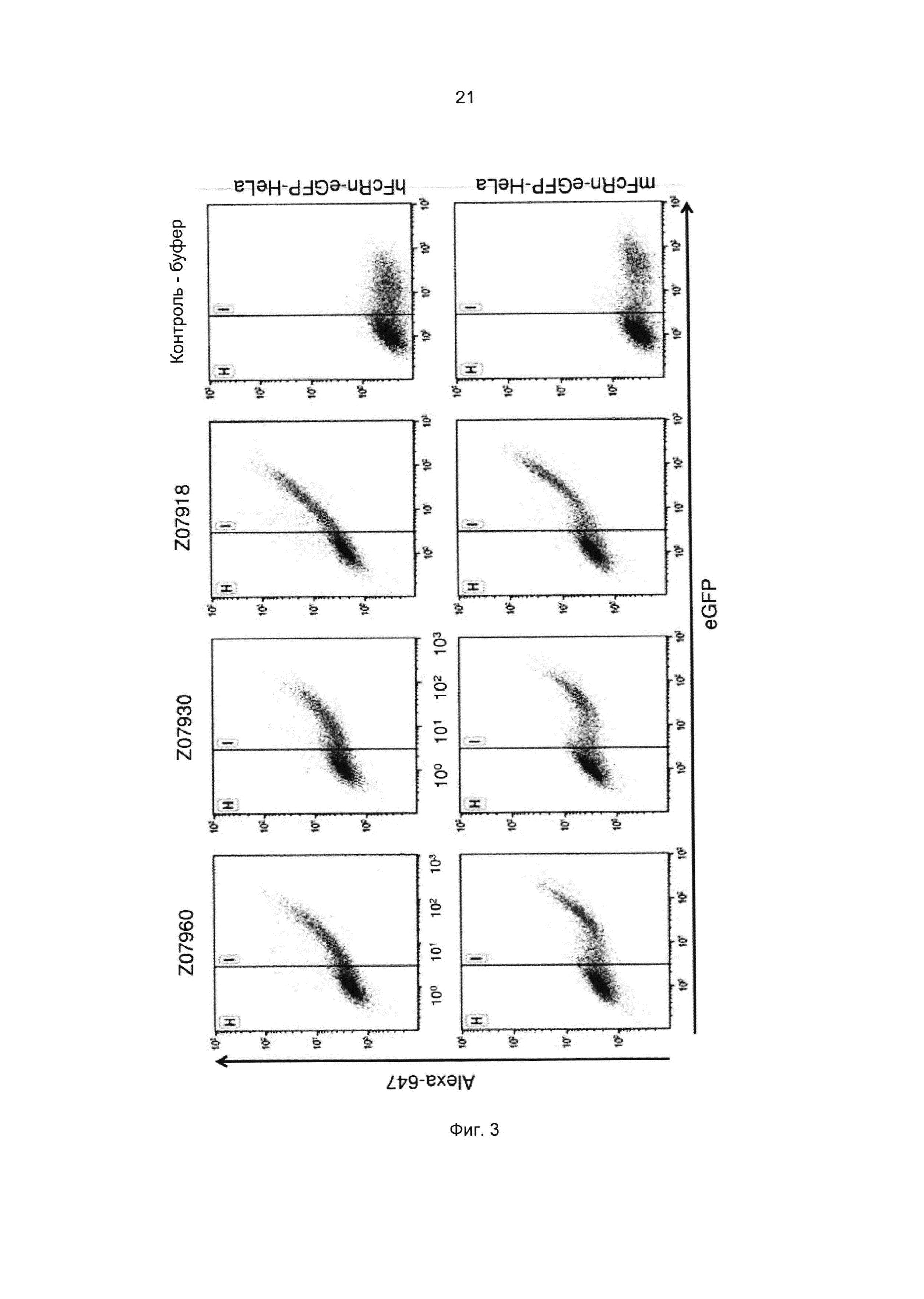
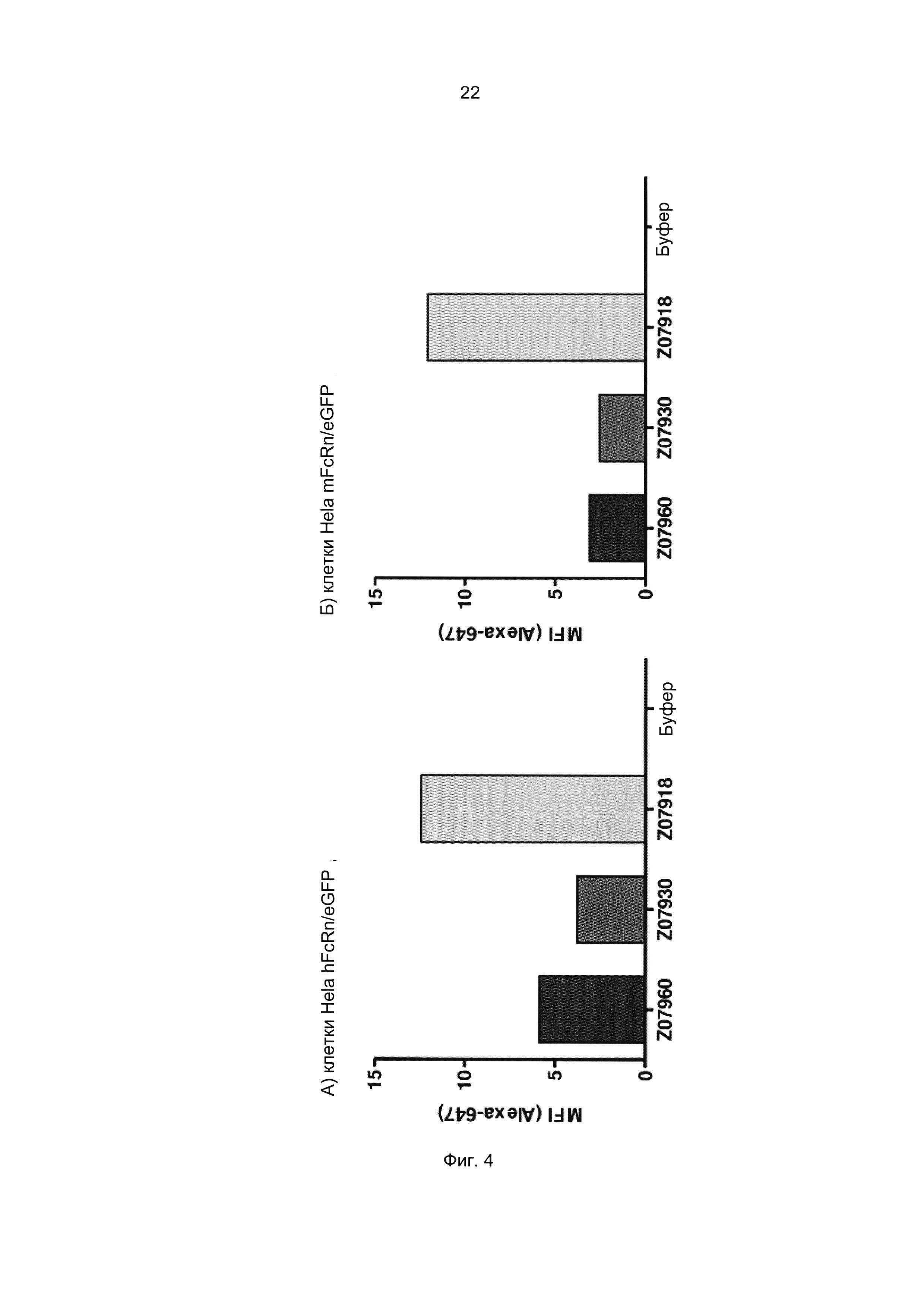
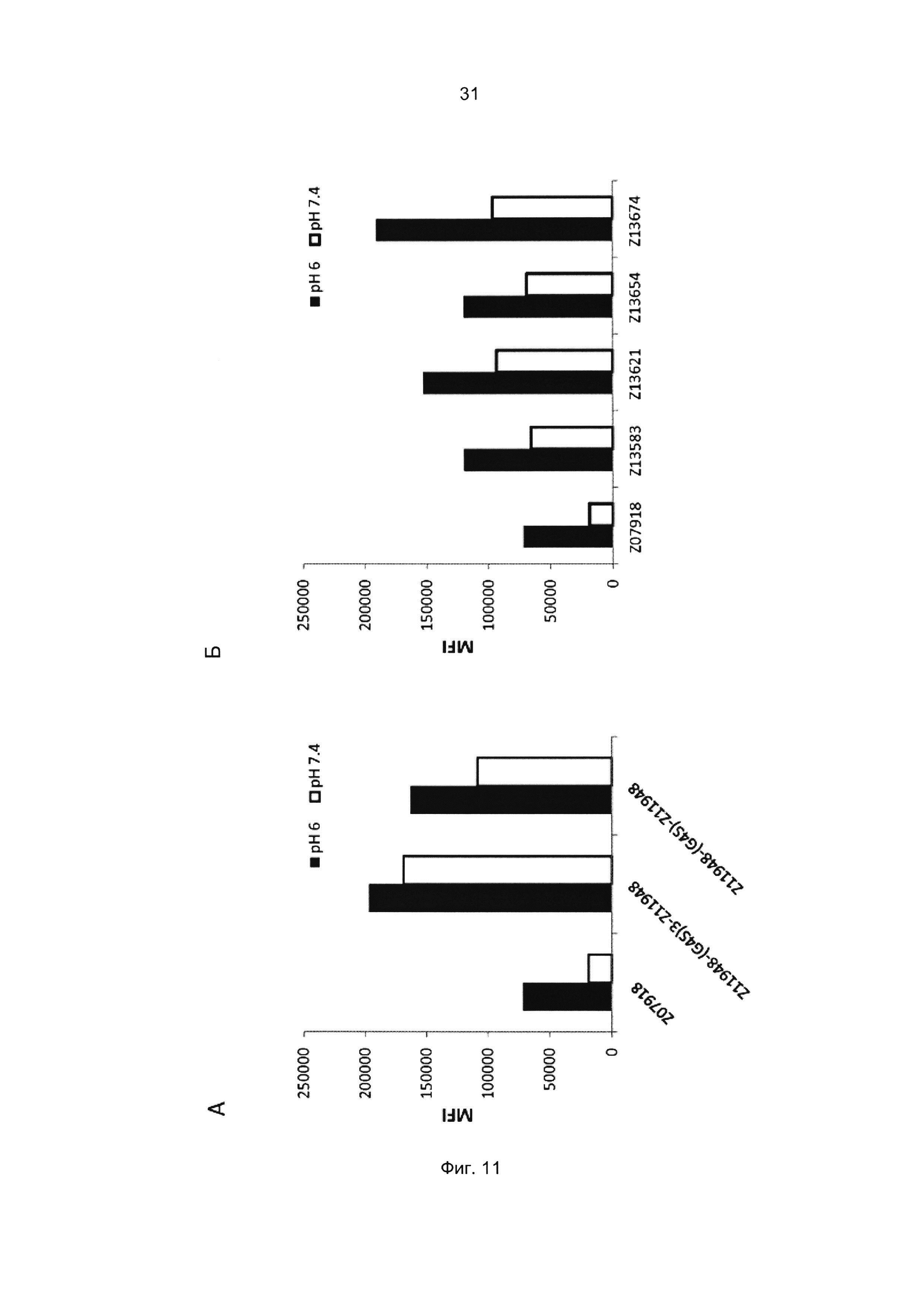
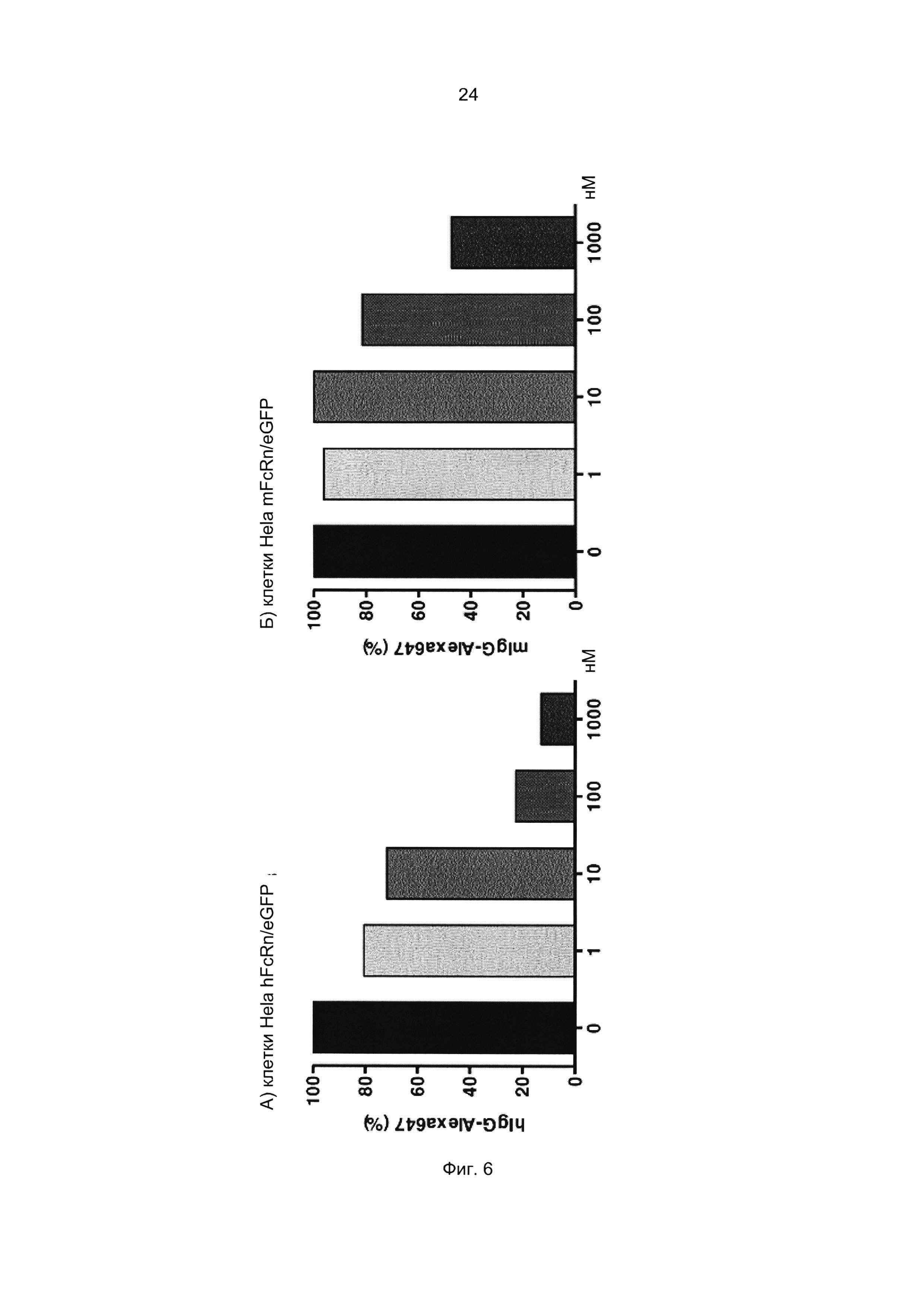
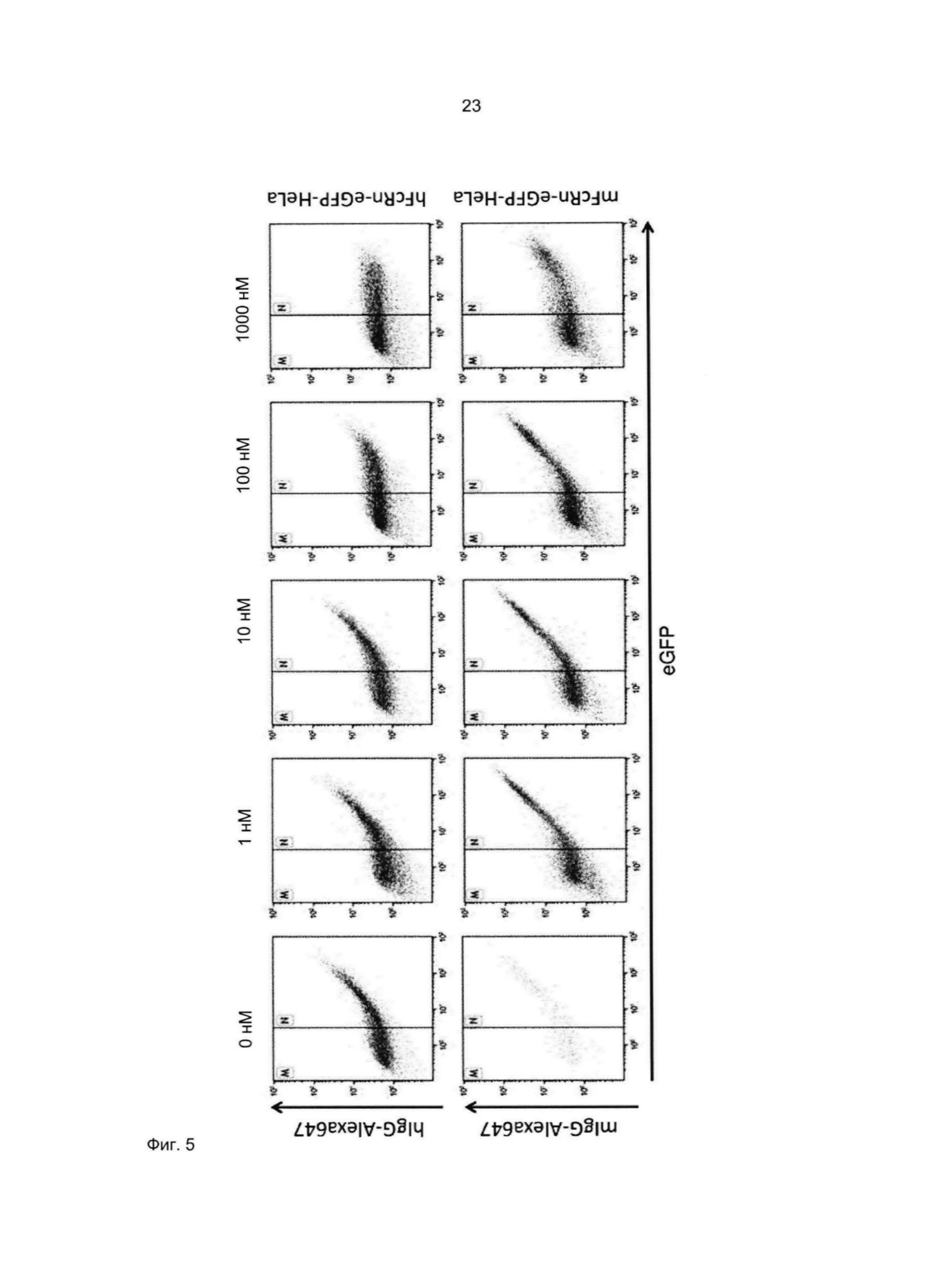
Специалист в области техники также поймет, что взаимодействие FcRn-связывающего полипептида и FcRn можно оценить с помощью FACS-анализа (сортировки клеток, активированную флуоресценцией), когда полученное значение средней интенсивности флуоресценции (MFI) является непрямым показателем силы связывания исследуемого полипептида относительно других исследуемых полипептидов в том же эксперименте. Так, более высокое значение MFI коррелирует с более высокой относительной аффинностью, а более низкое значение MFI коррелирует с более низкой относительной аффинностью.
В данном описании термин «более высокая связывающая способность» применительно к аффинности связывания с FcRn или к связыванию FcRn следует интерпретировать в контексте любого из упомянутых выше анализов для прямой или опосредованной оценки аффинности.
Как определено здесь, FcRn-связывающий димер связывается с FcRn с более высокой связывающей способностью по сравнению с указанными первой или второй мономерной единицей в отдельности. В одном воплощении FcRn-связывающий димер может связываться с FcRn со связывающей способностью, которая по меньшей мере в 2 раза, например по меньшей мере в 3 раза, например по меньшей мере в 4 раза, например по меньшей мере в 5 раз, например по меньшей мере в 6 раз, например по меньшей мере в 7 раз, например по меньшей мере в 8 раз, например по меньшей мере в 9 раз, например по меньшей мере в 10 раз, например по меньшей мере в 25 раз, например по меньшей мере в 50 раз, например по меньшей мере в 100 раз выше, чем у соответствующей первой мономерной единицы или второй мономерной единицы в отдельности. Данное соотношение может быть справедливо как при pH 6,0 и pH 7,4, так и только при pH 6,0 или только при pH 7,4.
В некоторых воплощениях, как объясняется ниже, FcRn-связывающий димер ингибирует связывание IgG с FcRn. В таких воплощениях указанный FcRn-связывающий димер может связываться с FcRn, так что способность FcRn-связывающего димера блокировать связывание IgG с FcRn по меньшей мере в 2 раза выше, например по меньшей мере в 3 раза выше, например по меньшей мере в 4 раза выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз выше, например по меньшей мере в 15 раз выше, например по меньшей мере в 20 раз выше, например по меньшей мере в 25 раз выше по сравнению с блокирующей способностью соответствующей первой или второй мономерной единицы в отдельности.
В некоторых воплощениях указанный FcRn-связывающий димер может связываться с FcRn, так что значение MFI взаимодействия между FcRn и FcRn-связывающим димером по меньшей мере в 2 раза выше, например по меньшей мере в 3 раза выше, например по меньшей мере в 4 раза выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз выше по сравнению со значением MFI взаимодействия между FcRn и соответствующей первой или второй мономерной единицей в отдельности.
В некоторых воплощениях указанный FcRn-связывающий димер может связываться с FcRn, так что значение KD взаимодействия между FcRn и FcRn-связывающим димером по меньшей мере в 2 раза ниже, например по меньшей мере в 3 раза ниже, например по меньшей мере в 4 раза ниже, например по меньшей мере в 5 раз ниже, например по меньшей мере в 10 раз ниже, например по меньшей мере в 25 раз ниже, например по меньшей мере в 50 раз ниже, например по меньшей мере в 100 раз ниже, например по меньшей мере в 1000 раз ниже по сравнению со значением KD взаимодействия между FcRn и соответствующей первой или второй мономерной единицей в отдельности.
В одном воплощении предложен FcRn-связывающий димер, который способен связываться с FcRn при pH 6,0, так что значение KD взаимодействия составляет самое большее 1×10-7 М, например самое большее 1×10-8 М, например самое большее 1×10-9 М, например самое большее 1×10-10 М, например самое большее 1×10-11 М, например самое большее 1×10-12 М. FcRn-связывающий димер по данному воплощению будет связывать или оставаться связанным с FcRn при кислых значениях pH, таких как pH 6,0, например в эндосомах. Если такой полипептид окажется во все более и более кислой внутриклеточной среде, он вновь вернется на плазматическую мембрану благодаря своему взаимодействию с FcRn и, таким образом, избежит деградации.
В одном воплощении значение KD взаимодействия между FcRn-связывающим димером и FcRn при pH 7,4 превышает значение KD указанного взаимодействия при pH 6,0. Таким образом, FcRn-связывающий полипептид будет связываться с FcRn при pH 6,0 с более высокой аффинностью, чем при pH 7,4. В одном воплощении значение KD указанного взаимодействия при pH 7,4 по меньшей мере в 2 раза выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз выше, например по меньшей мере в 25 раз выше, например по меньшей мере в 50 раз выше, например по меньшей мере в 100 раз выше, например по меньшей мере в 1000 раз выше, чем значение KD указанного взаимодействия при pH 6,0.
Как упоминалось выше, для анализа взаимодействия между FcRn-связывающим димером и FcRn можно использовать FACS-анализ. Так, можно оценивать взаимодействие между FcRn-связывающим димером и FcRn при pH 6,0 и pH 7,4 и сравнивать значение MFI при pH 6,0 и pH 7,4, как описано в экспериментальной части ниже. Полученное более высокое относительное значение MFI соответствует более высокой аффинности, а более низкое относительное значение MFI соответствует более низкой аффинности, при условии, что указанные значения MFI сравнивают в том же самом эксперименте. Так, в одном воплощении FcRn-связывающий димер связывается FcRn при pH 6,0 с более высокой аффинностью, чем при pH 7,4, например по меньшей мере на 10% выше, например по меньшей мере на 20% выше, например по меньшей мере на 35% выше, например по меньшей мере на 50% выше, например по меньшей мере на 100% выше.
В одном воплощении значение KD взаимодействия между FcRn-связывающим димером и FcRn при pH 7,4 составляет по меньшей мере 1×10-10 М, например по меньшей мере 1×10-9 М, например по меньшей мере 1×10-8 М, например по меньшей мере 1×10-7 М, например по меньшей мере 1×10-6 М, например по меньшей мере 1×10-5 М. В некоторых воплощениях единственным критерием взаимодействия между FcRn-связывающим димером и FcRn при pH 7,4 является то, что FcRn-связывающий димер, который связался с FcRn в более кислых условиях, быстрее отсоединяется от FcRn при повышении значения pH.
В альтернативном воплощении предложен FcRn-связывающий димер, у которого KD указанного взаимодействия при pH 7,4 является таким же или более низким, чем KD указанного взаимодействия при pH 6,0. FcRn-связывающий димер согласно данному воплощению будет связываться или оставаться связанным с FcRn в услоыиях с кислым pH (т.е. будет иметь достаточно низкую скорость диссоциации при pH 6,0, что не позволит произойти высвобождению), например в эндосоме, а также в среде с нейтральным или слегка щелочным pH, например на плазматической мембране. В более конкретном воплощении значение KD указанного взаимодействия при pH 7,4 по меньшей мере в 2 раза ниже, например по меньшей мере в 5 раз ниже, например по меньшей мере в 10 раз ниже, например по меньшей мере в 50 раз ниже, например по меньшей мере в 100 раз ниже, чем значение KD указанного взаимодействия при pH 6,0.
В другом воплощении предложен FcRn-связывающий димер, который способен связываться с FcRn при pH 7,4, так что значение KD взаимодействия составляет самое большее 1×10-7 М, например самое большее 1×10-8 М, например самое большее 1×10-9 М, например самое большее 1×10-10 М, например самое большее 1×10-11 М, например самое большее 1×10-12 М. FcRn-связывающий димер согласно данному воплощению будет связываться или оставаться связанным с FcRn в течение более продолжительного времени при нейтральных или слегка щелочных значениях pH, таких как pH 7,4, например на плазматической мембране. Термин «оставаться связанным» обозначает взаимодействие, характеризуемое низкой скоростью диссоциации в заданных условиях.
В общем, специалисту в области техники известно, что значение KD взаимодействия определяют как отношение между скоростью диссоциации (koff) и скоростью ассоциации (kon). Так, высокое значение KD может быть следствием либо высокого koff, либо низкого kon, либо и того, и другого, и наоборот, низкое значение KD может быть следствием либо низкого koff, либо высокого kon, либо и того, и другого.
Специалист в области техники поймет, что можно осуществить различные модификации и/или добавления к FcRn-связывающему димеру согласно любому описанному здесь аспекту для оптимизации полипептида для конкретного практического применения, без отклонения от сущности данного изобретения.
Например, в одном воплощении предложен FcRn-связывающий димер, как описано здесь, в котором по меньшей мере одна из указанных первой и второй мономерных единиц содержит по меньшей мере одну дополнительную аминокислоту на C-конце и/или N-конце. Такой полипептид следует рассматривать, как полипептид, который имеет один или более дополнительных аминокислотных остатков в самом первом и/или самом последнем положении в полипептидной цепи по меньшей мере одной из указанных первой и второй мономерных единиц. Так, по меньшей мере одна из указанных мономерных единиц FcRn-связывающего димера согласно данному описанию может содержать любое подходящее число дополнительных аминокислотных остатков, например по меньшей мере один дополнительный аминокислотный остаток. Каждый дополнительный аминокислотный остаток может быть добавлен отдельно или совместно, например для улучшения или упрощения получения, очистки, стабилизации in vivo или in vitro, присоединения или выявления полипептида. Такие дополнительные аминокислотные остатки могут содержать один или несколько аминокислотных остатков, добавленных для химического присоединения. Одним из таких примеров является добавление остатка цистеина. Такие дополнительные аминокислотные остатки могут также создавать «метку» для выделения или выявления полипептида, такие как His6-метка или «myc» (с-myc)-метка или «FLAGw-метка для взаимодействия с антителами, специфичными в отношении метки, или металл-аффинной хроматографии (IMAC) в случае гексагистидиновой метки.
Как обсуждено выше, дополнительные аминокислоты можно присоединять к FcRn-связывающему димеру или к любой из указанных первой и второй мономерных единиц или к обеим из них посредством химической конъюгации (с применением известных методов органической химии) или любым другим способом, таким как экспрессия FcRn-связывающего димера в виде слитого белка или присоединения любым другим образом, напрямую или через линкер, например аминокислотный линкер, как обсуждено выше.
Как обсуждено выше, дополнительные аминокислоты могут, например, содержать один или более доменов полипептида. Дополнительный полипептидный домен может придавать FcRn-связывающему димеру другую функцию, например такую как другая связывающая функция или ферментативная функция, или токсическая функция, или функция передачи флуоресцентного сигнала, или их комбинацию.
Так, во втором аспекте данного описания предложен слитый белок или конъюгат, содержащий первую группировку, состоящую из FcRn-связывающего димера согласно первому аспекту, и вторую группировку, состоящую из полипептида, обладающего желаемой биологической активностью. В другом воплощении указанный слитый белок или конъюгат может дополнительно содержать дополнительные группировки, имеющие желаемые биологические активности, которые могут быть такими же, как биологическая активность второй группировки, или отличаться от нее.
В одном воплощении указанного слитого белка или конъюгата общий размер молекулы меньше предельного размера для эффективного почечного клиренса при введении субъекту-млекопитающему.
В другом воплощении указанного слитого белка или конъюгата общий размер молекулы больше предельного размера для эффективного почечного клиренса при введении субъекту-млекопитающему.
В одном воплощении указанного слитого белка или конъюгата период полувыведения указанного слитого белка или конъюгата in vivo дольше, чем период полувыведения in vivo полипептида, обладающего желаемой биологической активностью, самого по себе.
Неограничивающие примеры желаемой биологической активности включают терапевтическую активность, связывающую активность и ферментативную активность.
В одном воплощении указанная желаемая биологическая активность представляет собой связывающую активность с выбранной мишенью.
Одним из примеров такой связывающей активности является связывающая активность, которая увеличивает период полувыведения слитого белка или конъюгата in vivo. Такой слитый белок или конъюгат может содержать по меньшей мере одну дополнительную группировку. В одном конкретном воплощении указанная мишень представляет собой альбумин, связывание с которым увеличивает период полувыведения указанного слитого белка или конъюгата in vivo. В одном воплощении указанная альбумин-связывающая активность обеспечивается альбумин-связывающим доменом (ABD) белка G стрептококка или его производным. Например, указанный слитый белок или конъюгат, содержащий по меньшей мере одну дополнительную группировку, может содержать [FcRn-связывающий димер] - [альбумин-связывающую группировку] - [группировку, обладающую аффинностью к выбранной мишени]. Кроме того, следует принимать во внимание, что указанный слитый белок или конъюгат может содержать альбумин-связывающую группировку или группировку, связывающую другую мишень, расположенную между двумя связывающими FcRn мономерными единицами, образующими FcRn-связывающий димер, как описано здесь, и, таким образом, в качестве неограничивающего примера, могут быть расположены следующим образом: [связывающая FcRn мономерная группировка] - [альбумин-связывающая группировка] - [связывающая FcRn мономерная группировка] - [группировка, обладающая аффинностью к выбранной мишени] или следующим образом: [связывающая FcRn мономерная группировка] - [группировка, обладающая аффинностью к выбранной мишени] - [связывающая FcRn мономерная группировка] - [альбумин-связывающая группировка]. Следует понимать, что группировки в составе слитого белка или конъюгата могут свободно располагаться в любом порядке от N-конца к C-концу полипептида. В одном воплощении указанный период полувыведения in vivo увеличен по меньшей мере в 10 раз, например по меньшей мере в 25 раз, например по меньшей мере в 50 раз, например по меньшей мере в 75 раз, например по меньшей мере в 100 раз по сравнению с периодом полувыведения in vivo слитого белка или конъюгата самого по себе.
В одном воплощении, когда комплекс между мишенью и слитым белком или конъюгатом согласно данному описанию образуется (или сохраняется) при кислых значениях pH, таких как pH 6,0, мишень избегает элиминации, происходящей посредством деградации в лизосомах. Таким образом, период полувыведения мишени увеличивается. Увеличение периода полувыведения предполагает, что скорость элиминации мишени ниже при взаимодействии с указанным слитым белком или конъюгатом, чем скорость элиминации молекулы-мишени в отсутствие указанного слитого белка или конъюгата. Кроме того, в данном воплощении желательно, чтобы связывание мишени со слитым белком или конъюгатом не мешало существенным образом функции мишени.
С другой стороны, когда при кислых значениях pH комплекс между мишенью и слитым белком или конъюгатом согласно данному описанию не сохраняется или не образуется, мишень поступает во внутриклеточные лизосомы, где подвергается деградации.
В одном воплощении предложен слитый белок или конъюгат, где скорость элиминации выбранной нежелательной мишени у субъекта повышена. Усиленная элиминация нежелательной мишени означает повышенную скорость элиминации мишени из тела многоклеточного организма по сравнению с «нормальной» скоростью элиминации молекулы-мишени самой по себе, то есть без предварительного взаимодействия со слитым белком или конъюгатом.
В другом воплощении связывание выбранной нежелательной мишени может инактивировать функцию мишени, таким образом блокируя ее биологическую активность в ситуациях, когда это желательно. Такая биологическая активность может, например, представлять собой активацию или блокирование рецепторов или ферментативную или, в ином случае, токсическую или нежелательную активность. Такой нежелательной мишенью может быть эндогенный гормон, фермент, цитокин, хемокин или мишень, имеющая некоторую другую биологическую активность. Посредством инактивирующего связывания мишени биологическая активность блокируется до тех пор, пока мишень не поступит на деградацию и не высвободится при низком значении pH, а связывающий мишень слитый белок не вернется в циркуляцию. Такая рециркуляция слитого белка, связывающего мишень (посредством его FcRn-связывающей группировки), позволяет «катализировать» удаление более чем одной молекулы выбранной нежелательной мишени.
Нежелательными мишенями могут, например, быть чужеродные белки и соединения или естественным образом экспрессируемые белки, уровень которых в плазме повышается при медицинских состояниях, когда терапевтический эффект может быть достигнут посредством элиминации указанного белка. Нежелательная мишень не обязательно равномерно распределена в плазме, но может концентрироваться в определенных участках, например вокруг опухоли или в местах воспаления.
Неограничивающие примеры мишеней выбраны из группы, состоящей из аллергенов, амилоидов, антител, аутоантигенов, факторов свертывания крови, гормонов, опухолевых клеток, молекул лекарств, цитокинов, хемокинов, протеаз, медиаторов гиперчувствительности, провоспалительных факторов, токсинов, таких как бактериальные токсины и яды змей, загрязнителей, металлов и антиоксидантов.
При определенных условиях, например при некоторых видах раковых заболеваний, желательно удалять эндогенные молекулы, например VEGF (фактор роста эндотелия сосудов), PDGF (фактор роста тромбоцитов), HGF (фактор роста гепатоцитов) и другие стимулирующие рост гормоны. Такие молекулы могут также служить мишенями для функции связывания указанного слитого белка или конъюгата.
При других условиях, например при некоторых иммунологических заболеваниях, может быть желательным транзиторное удаление эндогенных молекул, таких как определенные интерлейкины или TNF (фактор некроза опухолей). Такие молекулы могут также служить мишенями для функции связывания указанного слитого белка или конъюгата.
В одном воплощении вторая группировка, обладающая желаемой биологической активностью, представляет собой терапевтически активный полипептид. Неограничивающими примерами терапевтически активных полипептидов являются биомолекулы, такие как молекулы, выбранные из группы, состоящей из ферментов, например альгазидазы α и β, глюкоцереброзидазы, ларонидазы, арилсульфатазы, аглюкозидазы-α, аспарагиназы, фактора VII, фактора VIII, фактора IX и фактора Ха; гормонов и факторов роста, например гормона роста, трансформирующего фактора роста-β2, эритропоэтина, инсулина, инсулин-подобного фактора роста-1, миостатина, фактора роста костной ткани и глюкагон-подобного пептида-1; хемокинов, например CCL17, CCL19, CCL20, CCL21, CCL22, CCL27, XCL1 и CXC3CL1; и цитокинов, например интерлейкина (IL)-2, IL-4, IL-7, IL-10, IL-12, IL-15, IL-18, IL-22, IL-27, интерферона (IFN)-α, IFN-β, IFN-γ, фактора некроза опухоли (TNF), гранулоцитарного колониестимулирующего фактора (G-CSF), макрофагального CSF и гранулоцитарно/макрофагального CSF.
Специалисту в области техники понятно, что FcRn-связывающий димер согласно первому аспекту может быть полезен в слитом белке или в качестве партнера по конъюгации с любой другой группировкой. Таким образом, приведенный выше перечень терапевтически активных полипептидов не следует рассматривать как ограничивающий каким-либо образом.
Изобретение также предусматривает другие возможности создания слитых белков или конъюгатов. Так, FcRn-связывающий димер согласно первому аспекту изобретения может быть ковалентно связан со второй или дополнительной группировкой или группировками, которые проявляют другие функции дополнительно к связыванию с мишенью или вместо связывания с мишенью. Одним примером является слияние одного или более FcRn-связывающих димеров и ферментативно активных полипептидов, служащих рецептором или эффекторной группировкой.
Применительно к приведенному выше описанию слитых белков или конъюгатов, включающих FcRn-связывающий димер согласно изобретению, следует отметить, что обозначение первой, второй и последующих группировок употребляется в целях ясности для различения между FcRn-связывающим димером согласно изобретению с одной стороны, и группировками, проявляющими другие функции, с другой стороны. Данные обозначения не относятся к фактическому порядку различных доменов в полипептидной цепи слитого белка или конъюгата. Так, например, указанная первая группировка может без ограничений располагаться на N-конце, в середине или на C-конце слитого белка или конъюгата. Более того, FcRn-связывающий димер согласно данному описанию может содержать вторую группировку, расположенную между двумя связывающими FcRn мономерными единицами FcRn-связывающего димера.
Концентрация полумаксимального ингибирования (IC50) является показателем эффективности вещества при ингибировании конкретной количественно характеризуемой биологической или биохимической функции. Данный количественный показатель указывает, сколько конкретного вещества необходимо для ингибирования определенной биологической функции на 50%, и является общеупотребительным в области техники. В одном конкретном воплощении предложен FcRn-связывающий димер, слитый белок или конъюгат, как определено здесь, способный блокировать связывание IgG с FcRn, так что концентрация полумаксимального ингибирования (IC50) блокирования составляет самое большее 1×10-8 М, например самое большее 6×10-9 М, например самое большее 4×10-9 М, например самое большее 1×10-9 М, например самое большее 1×10-10 М, например самое большее 1×10-11 М. В одном воплощении предложен FcRn-связывающий димер, слитый белок или конъюгат, как определено здесь, способный блокировать связывание IgG с FcRn, так что концентрация полумаксимального ингибирования (IC50) блокирования по меньшей мере в 10 раз меньше, например по меньшей мере в 100 раз меньше, например по меньшей мере в 1000 раз меньше по сравнению с IC50 блокирования соответствующей первой или второй мономерной единицей в отдельности.
Ингибирование может происходить вследствие связывания FcRn-связывающего димера, слитого белка или конъюгата с той же или по меньшей мере частично перекрывающейся областью FcRn, что и в случае IgG. В альтернативном варианте FcRn-связывающий димер, слитый белок или конъюгат могут связываться с другой областью FcRn, чем IgG, но создавать пространственное препятствие связыванию IgG с FcRn. Так, скорость элиминации или клиренса IgG из системы циркуляции будет повышаться вследствие усиленной деградации IgG в лизосомах, поскольку опосредованная FcRn рециркуляция IgG будет полностью или частично недоступна, поскольку сайты связывания FcRn будут заняты FcRn-связывающим димером согласно данному изобретению. Другими словами, введение FcRn-связывающего димера, слитого белка или конъюгата согласно данному изобретению будет усиливать катаболизм циркулирующих IgG-антител.
В одном воплощении значение KD взаимодействия между FcRn-связывающим димером, слитым белком или конъюгатом и FcRn ниже, чем значение KD указанного взаимодействия между IgG и FcRn. Данное соотношение может быть справедливо при pH 6,0 и pH 7,4, или только при pH 6,0.
Вышеупомянутые аспекты также охватывают полипептиды, в которых FcRn-связывающий димер согласно первому аспекту или FcRn-связывающий димер, содержащийся в слитом белке или конъюгате согласно второму аспекту, дополнительно содержит метку, такую как метка, выбранная из группы, состоящей из флуоресцентных красителей и металлов, хромофорных красителей, хемилюминесцентных соединений и биолюминесцентных белков, ферментов, радионуклидов и радиоактивных частиц. Такие метки, например, можно применять для выявления полипептида.
В других воплощениях меченый FcRn-связывающий димер присутствует в виде группировки в составе слитого белка или конъюгата, также содержащего вторую группировку, обладающую желаемой биологической активностью и/или функцией связывания, как описано выше. В некоторых случаях метка может быть присоединена только к FcRn-связывающему димеру (например, к одной, двум или к обеим из указанных первой и второй мономерных единиц), а в некоторых случаях и к FcRn-связывающему димеру, и ко второй группировке конъюгата или слитого белка. Кроме того, возможно, что метка присоединена только ко второй группировке, но не к связывающей FcRn группировке. Так, в еще одном воплощении предложен FcRn-связывающий димер, содержащий вторую группировку, где указанная метка присоединена только ко второй группировке.
Когда упоминается меченый полипептид, это следует понимать как ссылку на все аспекты FcRn-связывающего димера согласно данному описанию, включая слитые белки и конъюгаты, содержащие FcRn-связывающий димер и вторую и возможные дополнительные группировки. Так, меченый полипептид может содержать только FcRn-связывающий димер и, например, терапевтический радионуклид, который может быть хелатирован или ковалентно связан с FcRn-связывающим димером, или содержать FcRn-связывающий димер, терапевтический радионуклид и вторую группировку, такую как малая молекула, обладающая желаемой биологической активностью, например обеспечивающая терапевтическую эффективность.
В воплощениях, где FcRn-связывающий димер, слитый белок или конъюгат являются радиоактивно меченными, такой радиоактивно меченный полипептид может содержать радионуклид. Большинство радионуклидов являются металлами по природе, используются в ионизированной форме и обычно не способны образовывать стабильные ковалентные связи с элементами, присутствующими в белках и пептидах. По этой причине мечение белков и пептидов радиоактивными металлами осуществляют с использованием хелаторов, то есть многозубчатых лигандов, которые образуют с ионами металлов нековалентные соединения, называемые хелатами. В воплощении FcRn-связывающего димера, слитого белка или конъюгата встраивание радионуклида возможно благодаря обеспечению хелатирующей среды, посредством которой радионуклид может координироваться, подвергаться хелатированию или образовывать комплекс с полипептидом.
Одним из примеров хелатора является хелатор полиаминополикарбоксилатного типа. Можно выделить два класса таких полиаминополикарбоксилатных хелаторов: макроциклические и ациклические хелаторы.
В одном воплощении FcRn-связывающий димер, слитый белок или конъюгат содержит хелатирующую среду, обеспечиваемую полиаминополикарбоксилатным хелатором, присоединенным к FcRn-связывающему димеру через тиоловую группу остатка цистеина или эпсилон-аминогруппу остатка лизина. В альтернативном варианте полиаминополикарбоксилатный хелатор может быть присоединен к любой части слитого белка или конъюгата согласно данному описанию, например ко второй или последующей группировке указанного слитого белка или конъюгата.
Наиболее часто используемыми макроциклическими хелаторами для радиоизотопов индия, галлия, иттрия, висмута, радиоактивных актиноидов и радиоактивных лантаноидов являются различные производные DOTA (1,4,7,10-тетраазациклододекан-1,4,7,10-тетрауксусной кислоты). В одном воплощении хелатирующая среда FcRn-связывающего димера, слитого белка или конъюгата обеспечивается DOTA или его производным. Более конкретно, в одном воплощении хелатирующие полипептиды, охватываемые данным изобретением, получают при помощи реакции производного DOTA, 1,4,7,10-тетраазациклододекан-1,4,7-трис-уксусной кислоты-10-малеимидоэтилацетамида (малеимидомоноамида-DOTA) с указанным полипептидом.
Кроме того, в качестве хелаторов также можно использовать 1,4,7-триазациклононан-1,4,7-триуксусную кислоту (NOTA) и ее производные. Так, в одном воплощении предложен FcRn-связывающий димер, слитый белок или конъюгат, где полиаминополикарбоксилатный хелатор представляет собой 1,4,7-триазациклононан-1,4,7-триуксусную кислоту или ее производное.
Наиболее часто используемыми ациклическими полиаминополикарбоксилатными хелаторами являются различные производные DTPA (диэтилентриамин-пентауксусной кислоты). Так, полипептиды, имеющие хелатирующую среду, обеспечиваемую диэтилентриамин-пентауксусной кислотой или ее производными, также входят в объем данного изобретения.
В дополнительном воплощении FcRn-связывающий димер, полученный рекомбинантно посредством экспрессии полинуклеотида или синтетически, конъюгирован с одним или более синтетическими полимерами, например, для увеличения его гидродинамического радиуса. Для этого часто используют полиэтиленгликоль (PEG, ПЭГ), но в области техники также применяли и другие полимеры. Такое «ПЭГилирование» можно применять для увеличения размера FcRn-связывающего димера, слитого белка или конъюгата согласно данному описанию до размера, превышающего пороговый для эффективной почечной экскреции.
В одном воплощении синтетический полимер конъюгирован с одним или более синтезированными химическим путем FcRn-связывающими димерами. С тем же самым синтетическим полимером могут быть конъюгированы и другие функциональные группы. Если FcRn-связывающий димер и другие компоненты синтезированы химическим путем, нет необходимости получать какой-либо из компонентов в биологической системе, если это не желательно.
В предпочтительном воплощении один или более полученных синтетическим или биологическим способом FcRn-связывающих димеров конъюгированы с синтетическим полимером для достижения размера, превышающего размер, ассоциированный с эффективным почечным клиренсом, и используются для блокирования связывания IgG с FcRn. Для сайт-специфического конъюгирования можно использовать единственный цистеин в мономерной единице FcRn-связывающего димера, например расположенный на C-конце цистеин, встроенный для этой цели. В случае разветвленного синтетического полимера, для повышения авидности и, следовательно, блокирующей способности с одним и тем же полимером могут быть конъюгированы более двух связывающих FcRn группировок.
В третьем аспекте данного изобретения предложен полинуклеотид, кодирующий FcRn-связывающий димер или слитый белок согласно данному описанию. Данное изобретение также охватывает способ получения FcRn-связывающего димера или слитого белка, как описано выше, включающий экспрессию полинуклеотида; вектор экспрессии, содержащий полинуклеотид, и клетку-хозяина, содержащую вектор экспрессии.
Изобретение также охватывает способ получения FcRn-связывающего димера или слитого белка, включающий культивирование указанной клетки-хозяина в условиях, позволяющих указанному полипептиду экспрессироваться с вектора экспрессии, и выделение полипептида.
В альтернативном варианте FcRn-связывающий димер или слитый белок согласно данному изобретению можно получать при помощи небиологического пептидного синтеза с использованием аминокислот и/или производных аминокислот, имеющих защищенные реакционноспособные боковые цепи, причем небиологический пептидный синтез включает
- пошаговое присоединение аминокислот и/или производных аминокислот для образования FcRn-связывающего димера или слитого белка, имеющего защищенные реакционноспособные боковые цепи,
- удаления защитных групп с реакционноспособных боковых цепей FcRn-связывающего димера или слитого белка и
- фолдинг FcRn-связывающего димера или слитого белка в водном растворе.
В четвертом аспекте изобретения предложена композиция, содержащая FcRn-связывающий димер, слитый белок или конъюгат согласно данному описанию и по меньшей мере один фармацевтически приемлемый эксципиент или носитель. В одном воплощении указанная композиция дополнительно содержит по меньшей мере один дополнительный активный агент, например по меньшей мере два дополнительных активных агента, например по меньшей мере три дополнительных активных агента. Неограничивающими примерами дополнительных активных агентов, которые могут оказаться полезными в такой комбинации, являются иммуносупрессорные агенты, противовоспалительные агенты, антимикробные агенты и ферменты.
В одном воплощении данного аспекта указанная композиция выполнена с возможностью введения способом, выбранным из группы, состоящей из перорального введения, интраназального введения, легочного введения, вагинального введения, ректального введения, внутривенной инъекции, внутрибрюшинной инъекции, внутримышечной инъекции, подкожной инъекции и внутрикожной инъекции.
В данном описании термин «системное введение» относится к способу введения, при котором представляющее интерес вещество попадает в систему кровообращения, так что воздействие оказывается на весь организм. Специалисту в области техники известно, что системное введение можно осуществлять посредством энтерального введения (абсорбции лекарственного средства в желудочно-кишечном тракте) или парентерального введения (обычно инъекции, инфузии или имплантации).
В одном воплощении указанная композиция выполнена с возможностью системного или местного введения. В некоторых воплощениях можно применять системное введение указанной композиции. В другом воплощении указанная композиция выполнена с возможностью местного введения. Например, местное введение может быть поверхностным, в виде мази, пасты, пены или крема. В другом воплощении указанная композиция выполнена с возможностью введения через слой эндотелия или эпителия. В этом случае может осуществляться трансцитоз композиции через указанный слой.
В одном воплощении скорость поглощения композиции, содержащей слитый белок или конъюгат согласно данному описанию, выше, чем скорость поглощения полипептидов, соответствующих второй или последующим группировкам, самих по себе. В одном воплощении скорость поглощения по меньшей мере в 2 раза выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз выше, например по меньшей мере в 25 раз выше, чем скорость поглощения второй или последующих группировок самих по себе.
С учетом приведенного выше описания следует понимать, что FcRn-связывающий димер, слитый белок или конъюгат или композиция согласно данному описанию могут, например, быть полезными в качестве терапевтического агента и/или в качестве средства увеличения периода полувыведения in vivo партнера по слиянию и/или в качестве средства увеличения скорости элиминации нежелательных мишеней.
Так, в пятом аспекте данного изобретения предложен FcRn-связывающий димер, слитый белок, конъюгат или композиция согласно данному описанию для применения в качестве лекарственного средства.
В связанном с ним шестом аспекте данного изобретения предложен способ лечения или профилактики субъекта, нуждающегося в этом, включающий стадию введения терапевтически или профилактически активного количества FcRn-связывающего димера, слитого белка, конъюгата или композиции согласно данному описанию.
В одном воплощении любого их этих двух последних аспектов лекарственное средство или способ предназначены для снижения уровня IgG у субъекта, нуждающегося в этом.
В одном воплощении любого из указанных двух последних аспектов лекарственное средство или способ предназначены для лечения или профилактики, в которых используют способность FcRn-связывающего димера по меньшей мере частично блокировать связывание IgG с FcRn, например для лечения или профилактики, при которых желателен усиленный катаболизм IgG антител.
В другом воплощении, когда используют блокирующую IgG способность, введение FcRn-связывающих димеров согласно данному описанию повышает эффективность лекарственного средства путем блокирования антител, проявляющих свойства, направленные против лекарственного средства. В частности, таким способом можно улучшить действие лекарственных средств, подвергающихся клиренсу антителами, или против которых вырабатываются нейтрализующие антитела, путем введения FcRn-связывающих димеров перед введением лекарственного средства, представляющего интерес.
В другом воплощении, когда используют блокирующую IgG способность, введение FcRn-связывающих димеров согласно данному описанию снижает неблагоприятное воздействие антител путем их удаления или уменьшения времени их циркуляции в кровотоке субъекта. Например, радиоактивно меченные или конъюгированные с токсином антитела можно удалять путем последующего введения FcRn-связывающих димеров согласно данному описанию. В альтернативном варианте, в случаях, когда нежелательные токсические эффекты возникают как реакция на лекарственное средство, представляющее собой используемое в терапевтических целях антитело, такой нежелательный эффект может быть ослаблен или нейтрализован путем последующего введения FcRn-связывающего димера для удаления таких антител или ограничения времени их циркуляции.
В одном воплощении условием, при котором может быть показано такое лечение или профилактика, является аутоиммунное состояние. В качестве неограничивающих примеров указанных состояний следует упомянуть острый диссеминированный энцефаломиелит (ADEM), острый некрозирующий геморрагический лейкоэнцефалит, болезнь Аддисона, агаммаглобулинемию, гнездную алопецию, амилоидоз, ANCA (антитела к цитоплазме нейтрофилов)-ассоциированный васкулит, анкилозирующий спондилит, нефрит, вызываемый анти-GBM (клубочковая базальная мембрана) / анти-ТВМ (базальная мембрана канальцев) антителами, антифосфолипидный синдром (APS), аутоиммунный ангионевротический отек, аутоиммунную апластическую анемию, аутоиммунную дизавтономию, аутоиммунный гепатит, аутоиммунную гиперлипидемию, аутоиммунный иммунодефицит, аутоиммунное заболевание внутреннего уха (AIED), аутоиммунный энцефалит с поражением лимбической системы, аутоиммунный миокардит, аутоиммунный панкреатит, аутоиммунную ретинопатию, аутоиммунную тромбоцитопеническую пурпуру (АТР), аутоиммунное заболевание щитовидной железы, аутоиммунные невропатии, связанные с крапивницей, аксональные и анальные, болезнь Бало, болезнь Бехчета, буллезный пемфигоид, кардиомиопатию, болезнь Кастлемана, целиакию, болезнь Шагаса, хроническую воспалительную демиелинизирующую полиневропатию (CIDP), хронический рецидивирующий мультифокальный остеомиелит (CRMO), синдром Черга-Штраусса, рубцующийся пемфигоид/доброкачественный пемфигоид слизистой, болезнь Крона, синдром Когана, болезнь холодовых агглютининов, врожденную блокаду сердца, миокардит Коксаки, болезнь CREST (кальциноз, синдром Рейно, нарушение моторики пищевода, склеродактилия и телеангиэктазия), эссенциальную смешанную криоглобулинемию, демиелинизирующие невропатии, герпетиформный дерматит, дерматомиозит, болезнь Девика (нейромиелит зрительного нерва), дилатационную кардиомиопатию, дискоидную волчанку, синдром Дресслера, эндометриоз, эозинофильный ангиоцентрический фиброз, эозинофильный фасцит, приобретенный буллезный эпидермолиз, нодозную эритему, экспериментальный аллергический энцефаломиелит, синдром Эванса, фиброзирующий альвеолит, гигантоклеточный артериит (височный артериит), гломерулонефрит, синдром Гудпасчера, гранулематоз с полиангиитом (GPA; Вегенера), болезнь Грейвса, синдром Гийена-Барре, энцефалит Хашимото, тиреоидит Хашимото, гемолитическую анемию, пурпуру Геноха-Шенлейна, гестационный герпес, гипогаммаглобулинемию, идиопатический тубулоинтерстициальный нефрит с гипокомплементемией, идиопатическую мембранозную нефропатию, идиопатическую тромбоцитопеническую пурпуру (ITP), IgA-нефропатию, IgG4-связанное заболевание, IgG4-связанное склерозирующее заболевание, иммунорегуляторные липопротеины, воспалительную аневризму аорты, воспалительную псевдоопухоль, миозит с тельцами включений, инсулинозависимый диабет (1 типа), интерстициальный цистит, ювенильный артрит, ювенильный диабет, синдром Кавасаки, опухоль Кюттнера, синдром Ламберта-Итона, лейкоцитокластический васкулит, плоский лишай, склеротический лишай, деревянистый конъюнктивит, болезнь линейных цепей IgA (LAD), болезнь Лайма, хронический фиброз средостения, болезнь Меньера, микроскопический полиангиит, синдром Микулича, смешанную болезнь соединительных тканей (MCTD), язву Мурена, синдром Морвана, болезнь Мухи-Хабермана, пемфигоид слизистых оболочек, многоочаговый фибросклероз, рассеянный склероз, тяжелую миастению, миозит, нарколепсию, нейромиелит зрительного нерва (Девика), нейромиотонию (синдром Исаакса), нейтропению, рубцующийся пемфигоид зрачка, нейрит зрительного нерва, болезнь Ормонда (ретроперитонеальный фиброз), палиндромный ревматизм, PANDAS (детские аутоиммунные нейропсихиатрические расстройства, ассоциированные со стрептококковой инфекцией), паранеопластическую дегенерацию мозжечка, парапротеинемические полинейропатии, пароксизмальную ночную гемоглобинурию (PNH), синдром Парри-Ромберга, синдром Персонейджа-Тернера, pars planitis (периферический увеит), гестационный пемфигоид, обыкновенную пузырчатку, периаортит, периартериит, периферическую нейропатию, околовенозный энцефаломиелит, пернициозную анемию, синдром POEMS (полинейропатия, органомегалия, моноклональная гаммапатия, кожные изменения), нодозный полиартериит, полиартрит, аутоиммунные полигландулярные синдромы типа I, II и III, ревматическая полимиалгия, полимиозит, постинфарктный синдром, постперикардиотомный синдром, дерматит, вызываемый прогестероном, первичный билиарный цирроз, первичный склерозирующий холангит, псориаз, псориатический артрит, идиопатический фиброз легких, гангренозную пиодермию, истинную эритроцитарную аплазию, болезнь Рейно, рефлекторную симпатическую дистрофию, синдром Рейтера, рецидивирующий полихондрит, синдром «беспокойных ног», ретроперитонеальный фиброз (болезнь Ормонда), ревматическую лихорадку, ревматоидный артрит, тиреоидит Риделя, саркоидоз, синдром Шмидта, склерит, склеродермию, синдром Шегрена, аутоиммунную реакцию, направленную на антигены спермы и яичек, синдром мышечной скованности, подострый бактериальный эндокардит (SBE), синдром Сусака, симпатическую офтальмию, артериит Такаясу, системную красную волчанку (SLE), височный артериит/гигантоклеточный артериит, тромботическую тромбоцитопеническую пурпуру (ТРР), синдром Толосы-Ханта, поперечный миелит, язвенный колит, недифференцированное заболевание соединительной ткани (UCTD), увеит, васкулит, везикулобуллезный дерматоз, витилиго, макроглобулинемию Вальденстрема и тепловую идиопатическую гемолитическую анемию.
В другом воплощении пятого и шестого аспектов состоянием, при котором может быть показано такое лечение или профилактика, является аллоиммунное состояние. В качестве неограничивающих примеров указанных состояний следует отметить несовпадение донора при трансплантации, обусловленное антителами против HLA; аллоиммунную тромбоцитопению плода и новорожденного FNAIT (или неонатальную аллоиммунную тромбоцитопению NAITP или NAIT или NAT, или аллоиммунную тромбоцитопению вследствие антигенной несовместимости плода и матери, FMAITP или FMAIT).
В другом воплощении пятого и шестого аспектов состояние, при котором может быть показано такое лечение или профилактика, выбрано из группы, состоящей из аутоиммунных полиэндокринных синдромов 1 типа (APECED, или синдрома Уитакера) и 2 типа (синдрома Шмидта), генерализованной алопеции, миастенического криза, тиреотоксического криза, заболеваний глаз, ассоциированных с тиреоидной патологией, тиреоидной офтальмопатии, аутоиммунного диабета, энцефалита и/или энцефалопатии, ассоциированных с аутоантителами, листовидной пузырчатки, буллезного эпидермолиза, герпетиформного дерматита, хореи Сиденгама, острой моторной аксональной нейропатии (AMAN), синдрома Миллера-Фишера, мультифокальной моторной нейропатии (MMN); опсоклонуса; воспалительной миопатии; синдрома Исаакса (аутоиммунной нейромиотонии), паранеопластических синдромов и энцефалита с поражением лимбической системы.
В другом воплощении пятого и шестого аспектов состояние, при котором может быть показано такое лечение или профилактика, выбрано из эпилепсии и судорог.
В другом воплощении предложен FcRn-связывающий димер, слитый белок, конъюгат или композиция согласно данному описанию для применения в блокировании или удалении нежелательной мишени из циркуляции. В одном воплощении указанная нежелательная мишень выбрана из группы, включающей аллергены, амилоиды, антитела, аутоантигены, факторы свертывания крови, гормоны, опухолевые клетки, молекулы лекарственных средств, цитокины, хемокины, медиаторы гиперчувствительности, провоспалительные факторы, токсины, такие как бактериальные токсины и яды змей, загрязнители, металлы и антиоксиданты.
Несмотря на то, что изобретение описано с указанием различных приведенных в качестве примера аспектов и воплощений, специалисты в области техники поймут, что можно осуществлять различные изменения и заменять эквиваленты их элементами, при этом оставаясь в рамках изобретения. Кроме того, можно осуществлять множество модификаций для адаптации конкретной ситуации или молекулы к идеям изобретения, при этом оставаясь в рамках изобретения. Таким образом, изобретение не ограничивается каким-либо конкретным рассмотренным воплощением, но охватывает все воплощения, находящиеся в рамках приложенной формулы изобретения.
КРАТКОЕ ОПИСАНИЕ ГРАФИЧЕСКИХ МАТЕРИАЛОВ
На Фиг. 1 представлен перечень аминокислотных последовательностей примеров связывающих FcRn полипептидов в мономерой форме (SEQ ID NO: 1-367) и связывающих FcRn полипептидов в димерной форме (SEQ ID NO: 368-376), а также аминокислотные последовательности варианта альбумин-связывающего полипептида РР013 (SEQ ID NO: 377), связывающего Taq полимеразу Z варианта Z03638 (SEQ ID NO: 378), αFcRn человека (SEQ ID NO: 379), αFcRn мыши (SEQ ID NO: 384), β2-микроглобулина человека (SEQ ID NO: 380), β2-микроглобулина мыши (SEQ ID NO: 381), αFcRn человека (SEQ ID NO: 382), когда он представлен человеческим FcRn-eGFP (усиленный зеленый флуоресцентный белок), и αFcRn мыши (SEQ ID NO: 383), когда он представлен мышиным FcRn-eGFP. Предсказанные FcRn-связывающие мотивы (ВМ) FcRn-связывающих полипептидов согласно данному описанию располагаются между остатком 8 и остатком 36 в последовательностях SEQ ID NO: 1-367. Аминокислотные последовательности полипептидов длиной 49 аминокислотных остатков (BMod), которые, как предсказано, составляют полный трехспиральный пучок в составе каждого из этих Z вариантов, располагаются между остатком 7 и остатком 55.
На Фиг. 2А-2Д показано связывание с FcRn человека при pH 6,0 и диссоциация при pH 6,0 и 7,4 для Z вариантов, несущих His6-метку, и для IgG, как описано в Примере 3. Наложенные сенсограммы, полученные с использованием прибора Biacore, демонстрирующие инъекцию при pH 6,0 с последующей диссоциацией при pH 6,0 (сплошная линия) и инъекции при pH 6,0 с последующей диссоциацией при pH 7,4 (пунктирная линия), приведены для: (A) Z07918 (SEQ ID NO: 1), (Б) Z07960 (SEQ ID NO: 4), (В) Z10109 (SEQ ID NO: 3), (Г) Z10193 (SEQ ID NO: 2) и (Д) IgG.
На Фиг. 3 показаны точечные диаграммы, полученные при анализе связывания FcRn-связывающего Z варианта с клетками HeLa, экспрессирующими человеческий (верхняя панель) и мышиный (нижняя панель) FcRn-eGFP, методом проточной цитометрии, как описано в Примере 4. Из-за гетерогенной экспрессии FcRn-eGFP у клеток HeLa гейты для клеток строили по уровню экспрессии FcRn-eGFP. Клетки в гейте Н считали FcRn-eGFP-отрицательными, а клетки в гейте I считали положительными. Инкубация с Z вариантами, меченными Alexa Fluor® 647, приводила к популяциям, положительным как по Alexa Fluor® 647, так и по eGFP, в то время как инкубация с буфером (контроль-буфер) не приводила. На Фиг. 3 показано, что все три варианта Z07960 (SEQ ID NO: 4), Z07930 (SEQ ID NO: 6) и Z07918 (SEQ ID NO: 1) связываются с FcRn человека и FcRn мыши. На оси у показана интенсивность Alexa Fluor® 647, а на оси х показана активность eGFP.
На Фиг. 4 показаны значения средней интенсивности флуоресценции (MFI) Z07960 (SEQ ID NO: 4), Z07930 (SEQ ID NO: 6) и Z07918 (SEQ ID NO: 1), меченных Alexa Fluor® 647, которую измеряли в исследовании связывания клеток, как описано в Примере 4. На диаграмме (А) показана MFI клеток HeLa, трансдуцированных человеческим FcRn-eGFP, а на диаграмме (Б) показана MFI клеток HeLa, трансдуцированных мышиным FcRn-eGFP.
На Фиг. 5 показаны точечные диаграммы, полученные при анализе связывания меченных Alexa Fluor® 647 мышиных или человеческих IgG с клетками HeLa, экспрессирующими человеческий (верхняя панель) и мышиный (нижняя панель) FcRn-eGFP, методом проточной цитометрии, как описано в Примере 5. Из-за гетерогенной экспрессии FcRn-eGFP в клетках HeLa гейты для клеток строили по количеству FcRn-eGFP на клеточной поверхности. Клетки в гейте М считали FcRn-eGFP-отрицательными, а клетки в гейте N считали положительными. Связывание 100 нМ меченных Alexa Fluor® 647 человеческих или мышиных IgG с клетками HeLa, трансдуцированными FcRn, показано на левой панели (0 нМ). На Фиг. показано, что связывание IgG блокировалось Z07918, несущим His6-метку (SEQ ID NO: 1), дозозависимым образом (1, 10, 100 и 1000 нМ). На оси у показана интенсивность Alexa Fluor® 647, а на оси х показана активность eGFP.
На Фиг. 6 показаны значения средней интенсивности флуоресценции (MFI) при связывании FcRn с IgG, меченными Alexa Fluor® 647, в присутствии различных концентраций Z07918 с His6-меткой (SEQ ID NO: 1) на: (А) клетках HeLa, трансдуцированных человеческим FcRn-eGFP, и (Б) клетках HeLa, трансдуцированных мышиным FcRn-eGFP, как описано в Примере 5. На Фиг. 6 показано дозозависимое блокирование связывания IgG-FcRn под воздействием Z варианта.
На Фиг. 7А-7 В показана кинетика связывания трех Z вариантов с FcRn человека при pH 6,0, как описано в Примере 6, с использованием прибора Biacore. Приведены сенсограммы для серии концентраций (A) Z11948 (SEQ ID NO: 354), (Б) Z11946 (SEQ ID NO: 355) и (В) Z11947 (SEQ ID NO: 356), соответственно, слитых с альбумин-связывающим полипептидом РР013 (SEQ ID NO: 377), а также для Z варианта Z03638, использованного в качестве контрольной молекулы (SEQ ID NO: 378; не специфический в отношении FcRn). Кинетику связывания анализировали по кривым для 640 нМ (пунктирная линия), 160 нМ (точечная пунктирная линия) и 40 нМ (сплошная серая линия) с использованием модели связывания 1:1 по Лангмюру. Кинетические параметры и аффинности, рассчитанные по аппроксимированным кривым (сплошные черные линии), приведены в Таблице 6.
На Фиг. 8 показаны фармакокинетические профили для трех Z вариантов, связывающих FcRn, слитых с альбумин-связывающим полипептидом РР013, полученные, как описано в Примере 6. Все Z варианты Z11947 (SEQ ID NO: 356, незаштрихованные квадраты), Z11946 (SEQ ID NO: 355, незаштрихованные треугольники) и Z11948 (SEQ ID NO: 354, незаштрихованные ромбы) демонстрировали увеличенный период полувыведения по сравнению с отрицательным контролем Z03638-PP013 (незаштрихованные круги).
На Фиг. 9 показано блокирование взаимодействия IgG с человеческим FcRn в присутствии His6-Z07918 (SEQ ID NO: 1; черные круги), IVIg (серые квадраты) и SCIg (серые треугольники), соответственно, которое исследовали, как описано в Примере 10.
На Фиг. 10 показано, что блокирование взаимодействия IgG-FcRn специфическими в отношении FcRn Z вариантами у мышей приводит к снижению уровней IgG. Как описано далее в Примере 11, мыши получали ежедневно пять инъекций носителя (+), слитого с ABD Z варианта Z07918-РР013 (незаштрихованный квадрат) и Z11948 (SEQ ID NO: 354; заштрихованный круг). Концентрацию эндогенного IgG определяли при помощи ELISA. Концентрацию IgG у отдельных мышей через 24, 72, 120 и 168 ч соотносили с уровнем в момент времени 0 ч и, таким образом, представляли результаты в процентах от IgG в момент времени 0 ч.
На Фиг. 11 показаны значения средней интенсивности флуоресценции (MFI) меченных Alexa Fluor® 647 димерных и мономерных полипептидов, связывающхся с трансфицированными человеческим FcRn-eGFP клетками HeLa, полученные, как описано в Примере 13. (А) Димеры Z11948-(G4S)3-Z11948 (SEQ ID NO: 369) и Z11948-(G4S)-Z11948 (SEQ ID NO: 368) и соответствующий мономер Z варианта, Z07918 (SEQ ID NO: 1), связывание с FcRn при pH 6 (черные столбики) и pH 7,4 (белые столбики). (Б) Мономеры первичного Z варианта Z07918 (SEQ ID NO: 1) и мономеры зрелых Z вариантов Z13583 (SEQ ID NO: 23), Z13621 (SEQ ID NO: 44), Z13654 (SEQ ID NO: 65) и Z13674 (SEQ ID NO: 75), связывание с FcRn при pH 6 (черные столбики) и pH 7,4 (белые столбики).
На Фиг. 12 показано блокирование связывания IgG человека с FcRn человека димерными и мономерными полипептидами, которое исследовали, как описано в Примере 14. (А) Димеры Z11948-(G4S)3-Z11948 (SEQ ID NO: 369) и Z11948-(G4S)-Z11948 (SEQ ID NO: 368); соответствующий мономер Z варианта, Z07918 (SEQ ID NO: 1), SCIg и IVIg. (Б) Мономеры первичного Z варианта Z07918 (SEQ ID NO: 1) и мономеры зрелых Z вариантов Z13583 (SEQ ID NO: 23) и Z13621 (SEQ ID NO: 44).
На Фиг. 13 показано pH-зависимое связывание полипептидов с hFcRn, которое исследовали методом ELISA, как описано в Примере 17. (А) Связывание указанных полипептидов при pH 6. (Б) Связывание указанных полипептидов при pH 7,4. При обоих значениях pH у димерных полипептидов (ZAZ####) наблюдали более эффективное связывание по сравнению с мономерным Z вариантом (Z13621).
На Фиг. 14 показано снижение уровней hIgG у трансгенных по FcRn мышей, получавших димерные полипептиды, как описано в Примере 20. (А) Снижение уровней hIgG было одинаково эффективным как при расположении альбумин-связывающего домена РР013 (SEQ ID NO: 377) между двумя Z группировками (ZAZ3715; SEQ ID NO: 371), так и при расположении РР013 на C-конце полипептида (ZZA3716; SEQ ID NO: 372). (Б) Одинаковое снижение уровней hIgG было получено с полипептидами ZAZ3869 (SEQ ID NO: 374), ZAZ3870 (SEQ ID NO: 375) и ZAZ3871 (SEQ ID NO: 376).
На Фиг. 15 показано дозозависимое снижение уровней hIgG у мышей NMRI, получавших димерные полипептиды ZAZ3715 (SEQ ID NO: 371) и ZAZ3824 (SEQ ID NO: 373), как описано в Примере 21.
На Фиг. 16 показаны сывороточные концентрации ZAZ3715 (SEQ ID NO: 371) и ZAZ3824 (SEQ ID NO: 373), соответственно, измеренные в том же исследовании катаболизма IgG, которое представлено на Фиг. 15.
На Фиг. 17 приведено изображение геля SDS-PAGE (полиакриламидный гель с додецилсульфатом натрия), на котором показаны исходные и мутированные FcRn-связывающие Z варианты до (0) и через 2 недели после (2w) проведения теста на стабильность. Дорожка 1: Z11948 (0), дорожка 2: Z11948 (2w), дорожка 3: Маркер мол. массы, дорожка 4: Z17347 (0), дорожка 5: Z17347 (2w), дорожка 6: Z17348 (0), дорожка 7: Z17348 (2w). Маркер молекулярной массы (Mw) представлял собой предварительно окрашенный белковый стандарт Novex® Sharp (216, 160, 110, 80, 60, 50, 40, 30, 20, 15, 10, 3,5 кДа). Диагональные полосы, заметные на Фиг. 17 , являются артефактом, отпечатком второго геля, который окрашивался в том же контейнере.
На Фиг. 18 показано связывание с FcRn человека и яванского макака при pH 6,0, как описано в Примере 25. Показаны наложенные сенсограммы, полученные на приборе Biacore, представляющие ответ на инъецирование 90 нМ Z варианта, несущего His6-метку, вдоль поверхности с адсорбированным hFcRn (черные линии) и cFcRn (серые линии) для: (A) Z13578 (сплошная линия) и Z18632 (пунктирная линия), (В) Z13616 (сплошная линия) и Z18633 (пунктирная линия) и (С) Z13621 (сплошная линия) и Z18634 (пунктирная линия).
Примеры
Краткое описание
В следующих Примерах изложена разработка новых молекул Z вариантов, нацеленных на неонатальный Fc рецептор (FcRn). Z варианты были получены с использованием технологии фагового дисплея. Гены, кодирующие описанные здесь FcRn-связывающие полипептиды, и соответствующие аминокислотные последовательности приведены на Фиг. 1 и обозначены идентификаторами SEQ ID NO: 1-353. Предсказанные FcRn-связывающие мотивы (ВМ) описанных здесь FcRn-связывающих полипептидов располагаются между остатком 8 и остатком 36 в последовательностях SEQ ID NO: 1-353. Кроме того, исследовали свойства связывания с FcRn и способность блокировать связывание IgG с FcRn указанных полипептидов в димерной форме.
Пример 1
Получение αFcRn человека и β2-микроглобулина человека (В2М)
В данном Примере внеклеточный домен (ECD) αFcRn человека (SEQ ID NO: 379) в комплексе с β2-микроглобулином человека (SEQ ID NO: 380) (комплекс обозначен как FcRn) и β2-микроглобулин человека в не связанной в комплекс форме (обозначен как В2М) получили в виде растворимых белков. Человеческие FcRn и В2М, полученные в данном Примере, использовали для отбора фагов, анализов ELISA и Biacore в Примерах 2 и 3.
Материалы и методы
Конструирование плазмид, содержащих гены αFcRn человека и β2-микроглобулина человека для применения при совместной экспрессии: Гены, кодирующие αFcRn человека (Genbank ВС008734.2) и β2-микроглобулин человека (В2М) (Genbank ВС032589.1), получили из OpenBiosystems. Фрагмент гена, кодирующий аминокислоты 24-290 αFcRn человека (αFcRnECD) (SEQ ID NO: 379), амплифицировали, используя ПЦР с перекрывающимися праймерами, для получения конструкции, состоящей из: сайта attB1/последовательности Козак, за которыми располагался ген, кодирующий лидерную последовательность каппа-цепи Ig, hFcRnECD, GS-линкер и flag-метку, за которыми располагался сайт attB2. Создали аналогичную конструкцию, содержащую фрагмент гена, кодирующий аминокислоты 21-119 В2М человека (SEQ ID NO: 380), однако flag-метку заменили на His6-метку. Конструкции встроили в плазмиду pDONOR221 (Invitrogen, кат. номер 12536-017) посредством рекомбинации с использованием системы Gateway (Invitrogen, кат. номер 11789020, смесь ферментов Gateway® BP Clonase® II) согласно рекомендациям производителя. После подтверждения правильности последовательностей конструкцию αFcRnECD человека встроили в 2K7bsd (Suter et al. (2006) Stem Cells 24: 615-623) посредством многосайтового клонирования Gateway вместе с плазмидой pENTR-CMV, содержащей промотор (Tai et al. (2012) PLoS One 7(9): е46269), с получением вектора 2K7bsd-CMV-hFcRnECD-Конструкцию гена В2М встроили аналогичным способом в 2K7neo (Suter et al., см. выше), с получением вектора 2K7neo-CMV-hB2M.
Клеточные культуры, получение рекомбинантных лентивирусных векторов и встраивание генов в клеточную линию SKOV-3: Клеточные линии HEK293T и SKOV-3 получили из АТСС (Американская коллекция типовых культур). Клетки выращивали при 37°C в инкубаторе с влажной атмосферой в присутствии 5% CO2. Полная среда для клеточной линии HEK293T представляла собой среду Игла в модификации Дюльбекко (DMEM) с добавлением 10% эмбриональной бычьей сыворотки (FBS), 1% раствора антибиотика-противогрибкового агента (АА) и 1% раствора заменимых аминокислот в минимальной поддерживающей среде MEM (NEAA). Полная среда для клеточной линии SKOV-3 представляла собой среду МакКоя 5А с добавлением 10% FBS и 1% АА.
Плазмиды 2K7bsd-CMV-hFcRnECD и 2K7neo-CMV-hB2M по отдельности совместно сотрансфицировали с оболочкой VSV-G и пакующей плазмидой gag/pol в клетки HEK293T с использованием трансфекции с хлоридом кальция (Zufferey et al. (1997) Nat Biotechnol 15(9): 871-5; Jakobsson et al. (2006) J Neurosci Res 84: 58-67). Супернатанты культуры HEK293, содержащие сформированные лентивирусные частицы с трансгенами αFcRnECD человека и В2М человека, соответственно, очищали от дебриса путем центрифугирования и фильтрования. Для последовательной трансдукции клеток SKOV-3 использовали два типа лентивирусных частиц. Клетки, успешно интегрировавшие оба гена αFcRnECD и В2М, отбирали путем добавления в культуральную среду бластицидина (Invitrogen) и G418 сульфата (Invitrogen) при пассажах клеток в течение двух недель. Полученную стабильно трансдуцированную клеточную линию SKOV-3 обозначили как SKOV-3 hFcRnECD/hB2M.
Экспрессия рекомбинантного FcRn человека: Клетки SKOV-3, соэкспрессирующие человеческие αFcRnECD и В2М с образованием FcRn человека, размножали и высаживали по 1,5×107 клеток во флаконы HYPERFlask (Corning) в 560 мл полной ростовой среды. Через пять суток, когда клетки оседали и увеличивались в числе, среду заменяли на полную ростовую среду без FBS. Через пять суток культивирование прекращали, а супернатант собирали, пропускали через 45 мкм фильтр и замораживали при -80°C.
Очистка рекомбинантного FcRn человека с помощью хроматографии с применением IgG человека Очистку белка осуществляли в системе  Explorer (GE Healthcare). 1 мл IgG человека (Pharmacia) в 0,2 М NaHCO3, 0,5 М NaCl pH 8,3 в концентрации 10 мг/мл связывали с 1 мл активированной NHS (N-гидроксисукцинимид) HP колонкой HiTrap (GE Healthcare) согласно инструкции изготовителя. Супернатант клеток SKOV-3, содержащую рекомбинантный FcRn человека, размораживали и доводили pH до 5,8 с помощью HCl. Затем супернатант наносили порциями по 100 мл на колонку, предварительно уравновешенную 20 мМ Bis-Tris pH 5,8. Колонку промывали 20 мл 20 мМ Bis-Tris pH 5,8 и элюировали фракциями по 1 мл с помощью 50 мМ Tris, pH 8,1. Замену буфера на PBS (забуференный фосфатом физиологический раствор, 10 мМ фосфат, 137 мМ NaCl, 2,68 мМ KCl, pH 7,4) производили с помощью диализа.
Explorer (GE Healthcare). 1 мл IgG человека (Pharmacia) в 0,2 М NaHCO3, 0,5 М NaCl pH 8,3 в концентрации 10 мг/мл связывали с 1 мл активированной NHS (N-гидроксисукцинимид) HP колонкой HiTrap (GE Healthcare) согласно инструкции изготовителя. Супернатант клеток SKOV-3, содержащую рекомбинантный FcRn человека, размораживали и доводили pH до 5,8 с помощью HCl. Затем супернатант наносили порциями по 100 мл на колонку, предварительно уравновешенную 20 мМ Bis-Tris pH 5,8. Колонку промывали 20 мл 20 мМ Bis-Tris pH 5,8 и элюировали фракциями по 1 мл с помощью 50 мМ Tris, pH 8,1. Замену буфера на PBS (забуференный фосфатом физиологический раствор, 10 мМ фосфат, 137 мМ NaCl, 2,68 мМ KCl, pH 7,4) производили с помощью диализа.
SDS-PAGE и Вестерн-блоттинг: Чистоту элюированных фракций после очистки белка анализировали при помощи SDS-PAGE с использованием реагента для окрашивания GelCode Blue Stain (Pierce) и набора для окрашивания SilverXpress® Silver (Invitrogen). Вестерн-блоттинг проводили с использованием нитроцеллюлозных мембран Amersham Hybond™-C Extra (GE Healthcare). Мембрану блокировали 5% обезжиренным сухим молоком (Semper) в TBS+T (50 мМ основание Trizma, 150 мМ NaCl, 0,05% Tween-20, pH 8) в течение 1 часа, затем зондировали смесью кроличьего поликлонального антитела к FCGRT (Atlas Antibodies) в концентрации 0,15 мкг/мл и кроличьего поликлонального антитела к В2М (Atlas Antibodies) в концентрации 0,23 мкг/мл в TBS+T. Затем мембрану инкубировали со стабилизированным антителом козы против иммуноглобулинов кролика, конъюгированным с пероксидазой хрена (Pierce) в разведении 1:10000 в TBS+T. После добавления субстрата ТМВ (Pierce) получали изображение мембраны на Amersham Hyperfilm ECL (GE Healthcare). Для проявления Hyperfilm использовали проявитель GBX developer и фиксатор GBX fixer (Sigma-Aldrich).
Получение не связанной в комплекс формы В2М человека: В2М человека получали в Е. coli. Экспрессию и очистку выполняли по существу как описано Sandalova et al. (2005) Acta Chryst F61: 1090-1093 и  et al. (2001) J Immunol 166: 7327-7334. Очищенный белок, состоящий из аминокислот 21-119 человеческого В2М, в мочевине подвергали рефолдингу в присутствии аргинина следующим образом: 0,5 мг В2М быстро добавляли к 2 мл буфера для рефолдинга (20 мл 1 М Tris-HCl pH 8,0, 16,87 г L-аргинина (забуференного HCl), 0,8 мл 0,5 М EDTA (этилендиаминтетрауксусная кислота), 61 мг GSSG (окисленный глутатион), 307 мг GSH (восстановленный глутатион) и воды milli-Q до конечного объема 200 мл, pH 8,0 с добавлением ингибитора протеаз (Roche, кат. номер 11873580001)). Процедуру рефолдинга проводили при 4°C в течение 4 часов. У прошедшего рефолдинг белка В2М заменяли буфер на PBS с использованием колонки PD-10 (GE Healthcare).
et al. (2001) J Immunol 166: 7327-7334. Очищенный белок, состоящий из аминокислот 21-119 человеческого В2М, в мочевине подвергали рефолдингу в присутствии аргинина следующим образом: 0,5 мг В2М быстро добавляли к 2 мл буфера для рефолдинга (20 мл 1 М Tris-HCl pH 8,0, 16,87 г L-аргинина (забуференного HCl), 0,8 мл 0,5 М EDTA (этилендиаминтетрауксусная кислота), 61 мг GSSG (окисленный глутатион), 307 мг GSH (восстановленный глутатион) и воды milli-Q до конечного объема 200 мл, pH 8,0 с добавлением ингибитора протеаз (Roche, кат. номер 11873580001)). Процедуру рефолдинга проводили при 4°C в течение 4 часов. У прошедшего рефолдинг белка В2М заменяли буфер на PBS с использованием колонки PD-10 (GE Healthcare).
Результаты
Конструирование плазмид, содержащих гены αFcRn человека и β2-микроглобулина человека для применения при совместной экспрессии: Гены, кодирующие внеклеточный домен α-цепи человеческого FcRn (αFcRnECD) и человеческий В2М, встраивали в лентивирусные плазмиды для переноса 2K7bsd и 2K7neo. соответственно. В обоих случаях встроенный ген находился под контролем промотора CMV. Гены удлиняли таким образом, чтобы полученные белки имели лидерную последовательность каппа цепи Ig на N-конце нацеливания белка для экспорта через эндоплазматический ретикулум в культуральную среду (сигнальная последовательность отщеплялась при секреции). Кроме того, αFcRnECD имел C-концевую спейсерную последовательность, за которой следовала FLAG-метка для возможного выявления. В2М человека имел C-концевую спейсерную последовательность, за которой следовала His6-метка для возможного выявления. Спейсерную последовательность добавляли для увеличения доступности метки. Лентивирусные переносящие плазмиды также содержали гены устойчивости к двум разным антибиотикам для обеспечения отбора клеток, в которые были вставлены обе конструкции.
Экспрессия и очистка рекомбинантного FcRn человека: Гены, кодирующие αFcRnECD и В2М, встраивали в геном SKOV-3 с использованием лентивирусов, и полученный белок FcRn секретировался в культуральную среду. Для захвата только FcRn, у которого сохранилось pH-зависимое связывание IgG, использовали аффинную хроматографию с иммобилизованным IgG, где рецептор захватывали при pH 5,8 и элюировали при pH 8,1. Захваченный белок элюировали в трех фракциях.
SDS-PAGE и Вестерн-блоттинг: Для определения присутствия двух пептидных цепей (αFcRnECD и В2М) полученного белка FcRn и для анализа чистоты элюированного материала элюированные фракции анализировали методом SDS-PAGE. При окрашивании геля красителем GelCode Blue были выявлялены две полосы с молекулярной массой 12 и 36 кДа, соответственно. Это приблизительно соответствует теоретическим молекулярным массам негликозилированных пептидных цепей 12 кДа для В2М и 31 кДа для αFcRnECD-Часть белка, представленная αFcRnECD, содержит один сайт гликозилирования, таким образом, предполагают, что ее молекулярная масса должна превышать 31 кДа. Гель также окрашивали серебром для повышения чувствительности и, возможно, выявления примесей. В первой элюированной фракции выявили полосу приблизительно 66 кДа, которая могла соответствовать BSA (бычий сывороточный альбумин), возникающему в результате прикрепления клетки. Общее количество белка, выделенного во фракциях 2 и 3, соответствовало 1,4 мг/л культуральной среды. Проведенный анализ объединенного вещества методом вестерн-блоттинг продемонстрировал по существу только две основные полосы и очень слабую дополнительную полосу менее 12 кДа, которая могла соответствовать продукту деградации.
Пример 2
Отбор Z вариантов, связывающих FcRn, и связывание в ELISA
В данном Примере FcRn человека использовали в качестве мишени для отбора методом фагового дисплея с использованием фаговой библиотеки Z вариантов. У выбранных клонов секвенировали ДНК, продуцировали в периплазматической фракции Е. coli и анализировали относительно FcRn в ELISA (твердофазный иммуноферментный анализ).
Материалы и методы
Биотинилирование белка-мишени FcRn и В2М: FcRn человека и В2М человека, полученные, как описано в Примере 1, биотинилировали с использованием биотина No-Weigh EZ-Link Sulfo-NHS-LC-Biotin (Pierce, кат. номер 21327) в молярном избытке 31 × (FcRn) и 10 × (В2М), соответственно, согласно рекомендациям производителя. Реакции проводили при комнатной температуре (RT) в течение 30 мин. Последующую замену буфера на PBS осуществляли с помощью кассет для диализа Slide-a-lyzer (Pierce, кат. номер 66380, MWCO (порог отсечения молекулярной массы) 10000 для FcRn, и Pierce, кат. номер 66333, MWCO 3500 для В2М), согласно инструкциям производителя.
Отбор Z вариантов, связывающих FcRn, методом фагового дисплея. Для отбора Z вариантов, связывающих FcRn, использовали библиотеку случайных вариантов белка Z, экспонированную на бактериофаге, сконструированную в фагмиде pAY02592, по существу как описано  et al. (2007) J Biotechnol, 128: 162-183. В данной библиотеке альбумин-связывающий домен (ABD, GA3 белка G штамма Streptococcus G148) использовали в качестве партнера по слиянию с Z вариантами. Библиотека была обозначена Zlib006Naive.ll, а ее размер составил 1,5×1010 элементов библиотеки (Z вариантов). Клетки Е. coli RRIΔM15 (
et al. (2007) J Biotechnol, 128: 162-183. В данной библиотеке альбумин-связывающий домен (ABD, GA3 белка G штамма Streptococcus G148) использовали в качестве партнера по слиянию с Z вариантами. Библиотека была обозначена Zlib006Naive.ll, а ее размер составил 1,5×1010 элементов библиотеки (Z вариантов). Клетки Е. coli RRIΔM15 ( et al., (1982) Nucleic Acids Res 10: 5765-5772), хранившиеся в глицерине, содержащие фагмидную библиотеку Zlib006Naive.ll, инокулировали в 20 л среды определенного состава, не содержащей пролина [дикалия гидрофосфат 7 г/л, тринатрия цитрат дигидрат 1 г/л, урацил 0,02 г/л, YNB (не содержащая аминокислот дрожжевая азотная основа Difco™, Becton Dickinson) 6,7 г/л, глюкозы моногидрат 5,5 г/л, L-аланин 0,3 г/л, L-аргинина моногидрохлорид 0,24 г/л, L-аспарагина моногидрат 0,11 г/л, L-цистеин 0,1 г/л, L-глутаминовая кислота 0,3 г/л, L-глутамин 0,1 г/л, глицин 0,2 г/л, L-гистидин 0,05 г/л, L-изолейцин 0,1 г/л, L-лейцин 0,1 г/л, L-лизина моногидрохлорид 0,25 г/л, L-метионин 0,1 г/л, L-фенилаланин 0,2 г/л, L-серин 0,3 г/л, L-треонин 0,2 г/л, L-триптофан 0,1 г/л, L-тирозин 0,05 г/л, L-валин 0,1 г/л], с добавлением 100 мкг/мл ампициллина. Культивирование осуществляли при 37°C в ферментере (Belach Bioteknik, BR20). Когда клетки достигали оптической плотности 0,75 при 600 нм (OD600), приблизительно 2,6 л культуры инфицировали с использованием хелперного фага M13K07 (New England Biolabs, кат. номер N0315S) в 10 × молярном избытке. Клетки инкубировали в течение 30 минут, после чего ферментер заполняли до 20 л TSB-YE (триптический соевый бульон-дрожжевой экстракт; 30 г/л TSB (триптический соевый бульон), 5 г/л дрожжевого экстракта) с добавлением 100 мкМ изопропил-β-D-1-тиогалактопиранозида (IPTG) для индуцирования экспрессии и 25 мкг/мл канамицина и 12,5 мкг/мл карбенициллина и культивировали при 30°C в течение 22 ч. Клетки в культуре осаждали путем центрифугирования при 15900 g. Фаговые частицы преципитировали из супернатанта двукратно в PEG/NaCl (полиэтиленгликоль/хлорид натрия), фильтровали и растворяли в PBS и глицерине, как описано
et al., (1982) Nucleic Acids Res 10: 5765-5772), хранившиеся в глицерине, содержащие фагмидную библиотеку Zlib006Naive.ll, инокулировали в 20 л среды определенного состава, не содержащей пролина [дикалия гидрофосфат 7 г/л, тринатрия цитрат дигидрат 1 г/л, урацил 0,02 г/л, YNB (не содержащая аминокислот дрожжевая азотная основа Difco™, Becton Dickinson) 6,7 г/л, глюкозы моногидрат 5,5 г/л, L-аланин 0,3 г/л, L-аргинина моногидрохлорид 0,24 г/л, L-аспарагина моногидрат 0,11 г/л, L-цистеин 0,1 г/л, L-глутаминовая кислота 0,3 г/л, L-глутамин 0,1 г/л, глицин 0,2 г/л, L-гистидин 0,05 г/л, L-изолейцин 0,1 г/л, L-лейцин 0,1 г/л, L-лизина моногидрохлорид 0,25 г/л, L-метионин 0,1 г/л, L-фенилаланин 0,2 г/л, L-серин 0,3 г/л, L-треонин 0,2 г/л, L-триптофан 0,1 г/л, L-тирозин 0,05 г/л, L-валин 0,1 г/л], с добавлением 100 мкг/мл ампициллина. Культивирование осуществляли при 37°C в ферментере (Belach Bioteknik, BR20). Когда клетки достигали оптической плотности 0,75 при 600 нм (OD600), приблизительно 2,6 л культуры инфицировали с использованием хелперного фага M13K07 (New England Biolabs, кат. номер N0315S) в 10 × молярном избытке. Клетки инкубировали в течение 30 минут, после чего ферментер заполняли до 20 л TSB-YE (триптический соевый бульон-дрожжевой экстракт; 30 г/л TSB (триптический соевый бульон), 5 г/л дрожжевого экстракта) с добавлением 100 мкМ изопропил-β-D-1-тиогалактопиранозида (IPTG) для индуцирования экспрессии и 25 мкг/мл канамицина и 12,5 мкг/мл карбенициллина и культивировали при 30°C в течение 22 ч. Клетки в культуре осаждали путем центрифугирования при 15900 g. Фаговые частицы преципитировали из супернатанта двукратно в PEG/NaCl (полиэтиленгликоль/хлорид натрия), фильтровали и растворяли в PBS и глицерине, как описано  et al., см. выше. Фаги хранили при -80°C до применения.
et al., см. выше. Фаги хранили при -80°C до применения.
Отбор против биотинилированного FcRn человека осуществляли в четырех циклах, разбитых на две разные цепочки. Приготовление резерва фагов и процедуру отбора осуществляли по существу, как описано в WO 2009/077175 для отбора против другой биотинилированной мишени. Амплификацию фага между циклами отбора осуществляли путем инфицирования фагом Е. coli RRIΔM15, затем культивирования в растворе следующим образом. Элюированный фаг и хелперный фаг M13K07 в 10 × избытке по сравнению с бактериями использовали для одновременного инфицирования бактерий в log фазе при 37°C в течение 30 мин без вращения и затем в течение 30 мин при медленном вращении. Перед инфицированием бактерии выращивали до log фазы в среде определенного состава, не содержащей пролин, описанной выше. Инфицированные бактерии осаждали посредством центрифугирования при 4300 g в течение 10 мин и ресуспендировали в 200 мл среды TSB+YE, дополненной 0,1 мМ IPTG, 25 мкг/мл канамицина и 100 мкг/мл ампициллина и культивировали при 30°C в течение ночи для получения фага.
Буфер для отбора содержал 100 мМ фосфата натрия и 150 мМ хлорида натрия, доведенных до pH 5,5 соляной кислотой, с добавлением 0,1% желатина и 0,1% Tween-20. При отборе в буфер для отбора добавляли человеческий сывороточный альбумин (HSA, Albucult, Novozymes) в конечной концентрации 1,5 мкМ. Для уменьшения количества неспецифически связывающихся вариантов осуществляли предварительный отбор путем инкубации фаговых частиц с покрытыми стрептавидином Dynabeads® М-280 Streptavidin (SA-гранулы, Dynal, кат. номер 112.06) в течение 1 часа при комнатной температуре. Вторую предварительную селекцию осуществляли в течение 30 мин при комнатной температуре против В2М человека, иммобилизованного на иммунопробирках (Nunc, кат. номер 444474). 5 мкг/мл В2М человека в карбонатном буфере (Sigma, кат. номер 068K8214) иммобилизовали на пробирке при 7°C в течение более 1 ч. После двукратного промывания водопроводной водой пробирки блокировали PBS с 0,5% казеина (Sigma, кат. номер С8654) в течение 30 мин при комнатной температуре перед применением. Все пробирки и гранулы, используемые в отборе, предварительно блокировали PBS с 0,1% желатина. Отбор осуществляли в растворе при комнатной температуре, затем осуществляли захват комплексов мишень-фаг на SA-гранулах, где использовали 1 мг гранул на 2,9 мкг биотинилированного FcRn. В 1 цикле отбора использовали 100 нМ биотинилированного FcRn и проводили две отмывки по две минуты каждая с помощью буфера для отбора. В последующих циклах применяли повышенную жесткость с использованием пониженной концентрации мишени и увеличенного числа промывок: 50 нМ/5 отмывок, 25 нМ/8 отмывок и 10 нМ/12 отмывок в циклах 2, 3 и 4, соответственно. После отмывок связанный фаг элюировали, используя две различных процедуры в двух цепочках отбора: 1) 500 мкл 0,1 М глицин-HCl, pH 2,2, с последующей немедленной нейтрализацией 50 мкл 1 М Tris-HCl, pH 8,0, и 450 мкл PBS, или 2) 500 мкл 100 мМ фосфата натрия и 150 мМ хлорида натрия, pH 8,0 с нейтрализацией 500 мкл PBS.
Секвенирование: ПЦР-фрагменты амплифицировали из отдельных колоний, используя стандартную программу ПЦР и праймеры AFFI-21 (5'-tgcttccggctcgtatgttgtgtg (SEQ ID NO: 385)) и AFFI-22 (5'-cggaaccagagccaccaccgg (SEQ ID NO: 386)). Секвенирование амплифицированных фрагментов выполняли с использованием биотинилированного олигонуклеотида AFFI-72 (5'-биотин-cggaaccagagccaccaccgg (SEQ ID NO: 387)) и набора для секвенирования BigDye® Terminator v3.1 Cycle (Applied Biosystems) согласно протоколу производителя. Продукты реакций секвенирования очищали путем связывания с магнитными гранулами, покрытыми стрептавидином (Detach Streptavidin Beads, Nordiag, кат. номер 2012-01), с использованием Magnatrix 8000 (Magnetic Biosolution) и анализировали на генетическом анализаторе ABI PRISM® 3130xl (РЕ Applied Biosystems).
Получение Z вариантов для ELISA: Секвенированные Z варианты получали путем инокуляции отдельных отобранных колоний в 10 мл среды TSB-YE с добавлением 100 мкг/мл ампициллина и 0,1 мМ IPTG и инкубации в течение 24 ч при 37°C. Клетки осаждали путем центрифугирования, ресуспендировали в 2 мл PBST (PBS с добавлением 0,05% Tween-20), замораживали при -80°C и размораживали в водяной бане для высвобождения периплазматической фракции клеток. Процедуру замораживания-оттаивания повторяли семь раз и затем осаждали клетки путем центрифугирования. Супернатант периплазматического экстракта содержал Z варианты в виде слияний с ABD, экспрессированные как AQHDEALE-[Z#####]-VDYV-[ABD]-YVPG ( et al., см. выше). Z##### обозначает отдельные Z варианты длиной 58 аминокислотных остатков.
et al., см. выше). Z##### обозначает отдельные Z варианты длиной 58 аминокислотных остатков.
Анализ KD Z вариантов методом ELISA: Связывание Z вариантов с FcRn анализировали методом ELISA. Половину площади 96-луночных планшетов для ELISA покрывали 2 мкг/мл антитела козы против ABD (собственного производства), разведенного до 2 мкг/мл в буфере для покрытия (50 мМ карбоната натрия, pH 9,6) при 4°C в течение ночи. Раствор антитела сливали и лунки блокировали 100 мкл PBSC (PBS с добавлением 0,5% казеина) в течение 1,5 ч при комнатной температуре. Блокирующий раствор удаляли, а в лунки добавляли 50 мкл периплазматического раствора, разведенного 1:4, и инкубировали в течение 1,5 ч при комнатной температуре при медленном встряхивании. Растворы сливали и лунки промывали четыре раза либо 0,05% буфером РСТ, pH 6,0 (фосфатно-цитратный буфер Мак-Илвейна, pH 6,0, с добавлением 0,05% Tween-20) или 0,05% буфером РСТ, pH 7,4 (фосфатно-цитратный буфер Мак-Илвейна, pH 7,4, с добавлением 0,05% Tween-20). В лунки добавляли белок-мишень, биотинилированный FcRn человека, в серии разведений 1:3 в концентрациях от 2 мкг/мл (45 нМ) до 0,3 нг/мл (6,9 пМ) в буфере РСС с pH 6,0 или pH 7,4, (фосфатно-цитратный буфер Мак-Илвейна, pH 6,0 или pH 7,4, с добавлением 0,5% казеина), соответственно. Планшеты инкубировали в течение 1,5 ч при комнатной температуре с последующими отмывками, как описано выше. Конъюгат стрептавидина и HRP (Thermo Scientific, кат. номер N100) разводили 1:30000 в буфере РСС с pH 6,0 или pH 7,4, соответственно, и добавляли в лунки с последующей инкубацией в течение 45 мин. После отмывки, как описано выше, в лунки добавляли 50 мкл субстрата ImmunoPure ТМВ (Thermo Scientific, кат. номер 34021) и планшеты проявляли согласно рекомендациям производителя. Измеряли поглощение при 450 нм с помощью считывающего устройства для многолуночных планшетов Victor3 (Perkin Elmer). Связывание Z варианта с нерелевантным белком использовали в качестве отрицательного контроля, а пустую пробу создавали, пропуская периплазматическую стадию. Z вариант, который связывался с FcRn в предварительном эксперименте (Z07918, SEQ ID NO: 1), использовали в качестве положительного контроля. Полученные значения анализировали с помощью программы GraphPad Prism 5 (GraphPad Software, Inc.) и нелинейной регрессии для определения аффинности (KD) взаимодействий.
Анализ специфичности Z вариантов методом ELISA: В другом эксперименте ELISA тестировали специфичность Z вариантов в отношении 2 мкг/мл биотинилированных человеческих белков В2М, PSMA (собственного производства) и IgG (поликлональные, Pharmacia) и буфера РСС с pH 6,0 или pH 7,4, соответственно. Исследование проводили при pH 6,0 и pH 7,4, соответственно, как описано выше. На стадии белка-мишени в лунки добавляли биотинилированные белки или буфер вместо FcRn.
Результаты
Отбор Z вариантов, связывающих FcRn, методом фагового дисплея: Индивидуальные клоны получили после четырех циклов отбора посредством фагового дисплея против биотинилированного человеческого FcRn.
Секвенирование: Осуществляли секвенирование клонов, выбранных случайным образом на четвертом раунде отбора. Каждому Z варианту был присвоен уникальный идентификационный номер #####, и отдельные варианты были обозначены как Z#####. Аминокислотные последовательности Z вариантов длиной 58 аминокислотных остатков представлены на Фиг. 1 как SEQ ID NO: 1-16 и SEQ ID NO: 353.
Выведенные FcRn-связывающие мотивы у данных Z вариантов располагаются между остатком 8 и остатком 36 в последовательностях SEQ ID NO: 1-16 и SEQ ID NO: 353 на Фиг. 1. Аминокислотные последовательности полипептидов длиной 49 аминокислотных остатков (BMod), которые, как предсказано, составляют полный трехспиральный пучок в составе каждого из этих Z вариантов, располагаются между остатком 7 и остатком 55.
ELISA с Z вариантами: В Е. coli были получены шестнадцать клонов в виде слитых белков с ABD. Периплазматические фракции использовали в ELISA против серии разведения FcRn человека. Клоны были представлены: Z07909 (SEQ ID NO: 13), Z07918 (SEQ ID NO: 1), Z07930 (SEQ ID NO: 6), Z07960 (SEQ ID NO: 4), Z10109 (SEQ ID NO: 3), Z10111 (SEQ ID NO: 8), Z10127 (SEQ ID NO: 12), Z10129 (SEQ ID NO: 9), Z10140 (SEQ ID NO: 5), Z10141 (SEQ ID NO: 10), Z10145 (SEQ ID NO: 15), Z10152 (SEQ ID NO: 14), Z10156 (SEQ ID NO: 11), Z10161 (SEQ ID NO: 16), Z10183 (SEQ ID NO: 7) и Z10193 (SEQ ID NO: 2). При pH 6,0 определили значения KD для всех вариантов, а при pH 7,4 - для трех вариантов (Таблица 2). У тринадцати вариантов данные для анализа KD при pH 7,4 получены не были. Ни один из шестнадцати вариантов не демонстрировал неспецифического связывания при исследовании против В2М, IgG или PSMA человека.
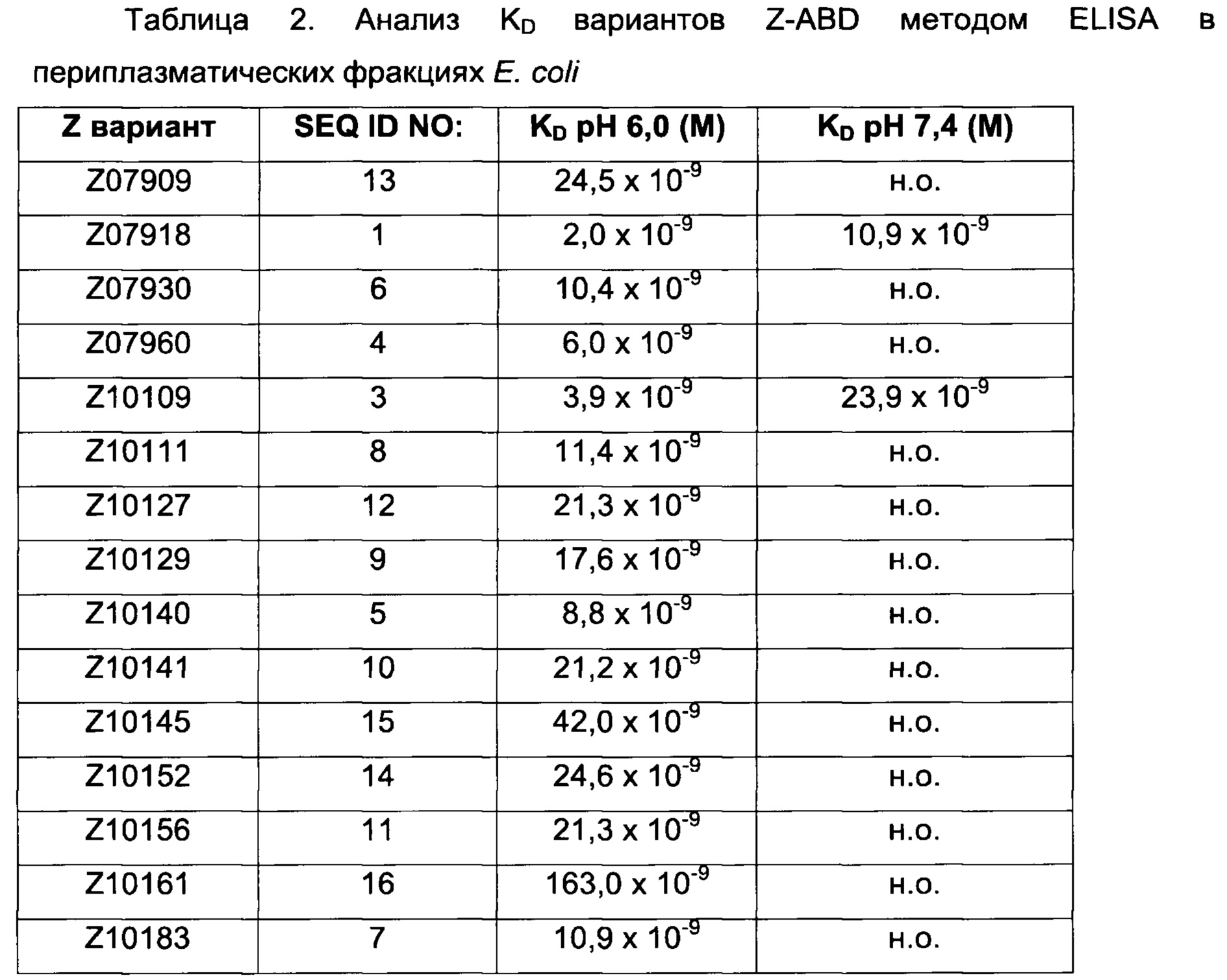

н.о. - не определяется.
Пример 3
Получение и характеризация Z вариантов, связывающих FcRn
В данном Примере в E. coli получили семнадцать Z вариантов, очистили и исследовали против человеческого FcRn в Biacore. Подгруппу указанных вариантов также исследовали против мышиного FcRn. Для исследования вторичной структуры в подгруппе Z вариантов использовали спектроскопию кругового дихроизма (CD).
Материалы и методы
Субклонирование Z вариантов: ДНК семнадцати Z вариантов, связывающих FcRn (SEQ ID NO: 1-16 и SEQ ID NO: 353), амплифицировали из вектора библиотеки pAY02592. Для конструирования мономерных молекул Z вариантов с N-концевой His6-меткой применяли стратегию субклонирования с использованием стандартных методов молекулярной биологии (по существу как подробно описано в WO 2009/077175 для Z вариантов, связывающих другую мишень). Фрагменты Z генов субклонировали в экспрессирующий вектор pAY01448, что приводило к кодируемой последовательности MGSSHHHHHHLQ-[Z#####]-VD.
Кроме того, вариант Z07918 (SEQ ID NO: 707), связывающийся с FcRn, но начинающийся с аминокислот АЕ вместо VD и обозначенный как Z11948 (SEQ ID NO: 1060), клонировали в виде гомодимерных конструкций с двумя разными линкерами между Z вариантами и с последующей C-концевой His6-меткой. Для этого использовали стандартные методы молекулярной биологии, включая амплификацию ДНК, рестрикцию соответствующими рестрикционными ферментами и лигирование ДНК. Два линкера получили от Thermo Fisher Scientific. Фрагменты Z генов субклонировали в экспрессирующий вектор (рЕТ-26 origin, Novagen), что приводило к кодируемой последовательности [Z#####]-GT-(G4S)-PR-[Z#####]-LEHHHHHH и [Z#####]-GT-(G4S)3-[Z#####]-LEHHHHHH, соответственно.
Культивирование и очистка: Клетки E. coli BL21(DE3) (Novagen) трансформировали плазмидами, содержащими фрагмент гена каждого соответствующего Z варианта, связывающего FcRn, и культивировали при 37°C в 800 или 1000 мл среды TSB-YE с добавлением 50 мкг/мл канамицина. При OD600, равной 2 для индуцирования экспрессии добавляли IPTG в конечной концентрации 0,17 или 0,2 мМ и инкубировали культуру при 37°C еще в течение 5 ч. Клетки собирали посредством центрифугирования.
Приблизительно 2-5 г каждого клеточного осадка ресуспендировали в 10-25 мл буфера для связывания (20 мМ фосфат натрия, 0,5 М NaCl, 20 мМ имидазол, pH 7,4) с добавлением Benzonase® (Merck, кат. номер 1.01654.0001) до концентрации 15 U/мл и лизоцима (Sigma, кат. номер L-7651) до концентрации 0,5 мг/мл. После разрушения клеток посредством трех циклов замораживания-оттаивания или воздействия ультразвуком удаляли клеточный дебрис путем центрифугирования и каждый супернатант наносили на колонку His GraviTrap IMAC объемом 1 мл (GE Healthcare, кат. номер 11-0033-99). Примеси удаляли путем промывания отмывочным буфером (20 мМ фосфат натрия, 0,5 М NaCl, 20 или 60 мМ имидазол, pH 7,4), и затем элюировали Z варианты, связывающие FcRn, элюирующим буфером 1 (20 мМ фосфат натрия, 0,5 М хлорид натрия, 250 мМ имидазол, pH 7,4) или элюирующим буфером 2 (0,1 М уксусная кислота, 0,5 М хлорид натрия, pH 4,5). У выделенных Z вариантов заменяли буфер на PBS с использованием колонок PD-10 (GE Healthcare) согласно протоколу производителя. Концентрации белка определяли путем измерения поглощения при 280 нм с помощью спектрофотометра NanoDrop® ND-1000 и с использованием коэффициента экстинкции соответствующего белка. Чистоту Z вариантов, связывающих FcRn, анализировали с помощью SDS-PAGE с окрашиванием Кумасси синим. Идентичность каждого выделенного Z варианта, связывающего FcRn, подтверждали с помощью LC/MS (жидкостная хроматография с масс-спектрометрией).
Анализ CD: Очищенные Z варианты, несущие His6-метку, разводили до 0,5 мг/мл в PBS. Для каждого разведенного Z варианта получали спектр CD при 250-295 нм или 250-190 нм при 20°C. Кроме того, проводили измерение при переменной температуре (VTM) для определения температуры плавления (Tm). При VTM измеряли поглощение при 221 нм при повышении температуры с 20 до 90°C, с увеличением температуры на 5°C в минуту. После процедуры нагревания получали новый спектр CD при 20°C для изучения способности Z вариантов к рефолдингу. Измерения CD проводили с помощью спектрополяриметра Jasco J-810 (Jasco Scandinavia АВ) в ячейке с длиной оптического пути 1 мм.
Анализ связывания и кинетики посредством Biacore: Взаимодействие FcRn-связывающих Z вариантов, несущих His6-метку, с FcRn человека анализировали с использованием прибора Biacore 2000 (GE Healthcare). FcRn человека иммобилизовали в проточной ячейке на слое карбоксилированного декстрана на поверхности чипа СМ5 (GE Healthcare). Иммобилизацию осуществляли посредством химии аминного связывания согласно протоколу производителя и используя в качестве подвижного буфера HBS-EP (GE Healthcare). Поверхность одной проточной ячейки чипа активировали и дезактивировали, чтобы использовать в качестве пустой пробы при инъекциях аналитов. В двух экспериментах по связыванию, представленных ниже, в качестве подвижного буфера использовали фосфатно-цитратный буфер Мак-Илвейна, pH 6,0, с добавлением 0,005% Tween-20 (0,005% РСТ). Во всех экспериментах использовали скорость тока 50 мкл/мин.
В одном эксперименте сравнивали диссоциацию при pH 6,0 и диссоциацию при pH 7,4. Z варианты, несущие His6-метку, и моноклональный IgG1 человека разводили в проточном буфере до конечной концентрации 250 нМ или 2,5 нМ, соответственно, и пропускали через чип с FcRn в течение 1 минуты, используя процедуру соинъекции. Вторая инъекция в процедуре соинъекции, представляющая фазу диссоциации взаимодействий, включала введение либо подвижного буфера (pH 6,0), либо 0,005% РСТ с pH 7,4. Z вариантам позволяли диссоциировать в течение 1 минуты, за исключением Z07918 и Z10193, которым позволяли диссоциировать в течение 4 минут, после чего по поверхности пропускали подвижный буфер в течение 5 минут для уравновешивания. IgG позволяли диссоциировать в течение 4 минут перед уравновешиванием. Инъекции буфера выполняли аналогичным образом; соинъекция буфера с pH 6,0 и затем с pH 6,0, либо соинъекция буфера буфера с pH 6,0 и затем с pH 7,4. Результаты анализировали в программе BiaEvaluation 4.1 (GE Healthcare). Кривые, полученные на поверхности без лиганда, вычитали из кривых, полученных на поверхности с лигандом. Кроме того, кривые инъекций буфера вычитали из кривых Z вариантов и из кривых IgG для корректировки эффектов буфера.
В другом эксперименте определяли приблизительные кинетические константы (kon и koff) и аффинности (KD) для подгруппы Z вариантов, несущих His6-метку. Выполняли инъекции Z вариантов в трех концентрациях в течение 1 минуты с последующей диссоциацией в подвижном буфере в течение 1 минуты. Перед началом следующего цикла поверхности уравновешивали подвижным буфером в течение 7,5 минут. Инъецированные концентрации составляли либо 675 нМ, 225 нМ и 75 нМ (Z10140, Z10156 и Z10183), либо 225 нМ, 75 нМ и 25 нМ (Z07918 и Z10193). Кинетические константы рассчитывали на основании сенсограмм с использованием модели Лангмюра 1:1 программы BiaEvaluation 4.1 (GE Healthcare).
В отдельном эксперименте определяли аффинность взаимодействий Z вариантов с hFcRn (SEQ ID NO: 379) и mFcRn (SEQ ID NO: 384), соответственно, как при pH 6,0 так и pH 7,4 на приборе Biacore 3000 (GE Healthcare). hFcRn и mFcRn получали по существу, как описано в Примере 1, но для получения mFcRn вместо человеческих клеток SKOV-3 использовали мышиные клетки 3Т3, и иммобилизовали их в отдельных проточных ячейках чипа СМ5 в ацетатном буфере с pH 4,65. Уровень иммобилизации составлял приблизительно 1000 RU (резонансные единицы) для обоих рецепторов. Эталонную проточную ячейку создавали путем активации и дезактивации. В качестве подвижного буфера и для разведения аналитов использовали 0,005% РСТ с pH 6,0 или 7,4. Все анализы выполняли при 25°C. Константы аффинности для Z вариантов, несущих His6-метку Z07918 (SEQ ID NO: 1), Z07960 (SEQ ID NO: 4) и Z10193 (SEQ ID NO: 2), определяли путем инъецирования серии разведений от 1024 нМ до 0,5 нМ (pH 6,0) или от 10240 нМ до 5 нМ (pH 7,4). Аффинности рассчитывали с помощью программы GraphPad Prism 5, используя модель насыщения при связывании в одном сайте.
Анализ блокирования посредством AlphaLISA: Способность Z вариантов ингибировать связывание IgG с FcRn анализировали в анализе AlphaLISA с помощью мультипланшетного считывающего устройства EnSpire 2300 (Perkin Elmer). IgG человека (Roactemra) иммобилизовали на гранулах-акцепторах AlphaLISA (Perkin Elmer, кат. номер 6772002) согласно рекомендациям производителя. В 384-луночном планшете (Perkin Elmer, кат. № G6005350) делали ступенчатые серийные разведения 1:3 Z вариантов, меченных His, до конечных концентраций от 250 нМ до 38 пМ и инкубировали в течение 45 мин с 10 нМ биотинилированным FcRn человека (Biorbyt, кат. номер orb84388; биотинилированного по существу, как описано в Примере 2) в буфере AlphaLISA (Perkin Elmer, кат. номер AL000F), pH доводили до 6,0 с помощью HCl. Покрытые IgG гранулы-акцепторы добавляли в конечной концентрации 10 мкМ и инкубировали в течение 45 мин. В конце добавляли покрытые стрептавидином гранулы-доноры (Perkin Elmer, кат. номер 6772002) до конечной концентрации 40 мкг/мл и инкубировали в течение 30 мин. Все инкубации проводили при комнатной температуре в темноте. Планшеты анализировали с помощью инструмента EnSpire, а значения IC50 рассчитывали с помощью GraphPad Prism 5.
Результаты
Культивирование и очистка: В Е. coli получили семнадцать Z вариантов, связывающих FcRn (SEQ ID NO: 1-16 и SEQ ID NO: 353), сконструированных с N-концевой His6 меткой. Количество белка, очищенного с помощью IMAC из приблизительно 2-5 г бактериального осадка, которое определяли спектрофотометрически путем измерения поглощения при 280 нм, у различных Z вариантов, связывающих FcRn, варьировало от приблизительно 10 мг до 20 мг. Анализ каждого полученного препарата белка с помощью SDS-PAGE показал, что они преимущественно содержали FcRn-связывающий Z вариант. Правильную идентичность и молекулярную массу каждого Z варианта, связывающего FcRn, подтверждали с помощью HPLC-MS анализа.
Анализ CD: Спектры CD, определенные для шести Z вариантов, показали, что каждый из них имел α-спиральную структуру при 20°C. Этот результат был также подтвержден с помощью измерения при переменной температуре, где определяли температуры плавления (Tm) (Таблица 3). При наложении спектров, полученных до и после нагревания до 90°C, у шести Z вариантов наблюдался обратимый фолдинг.
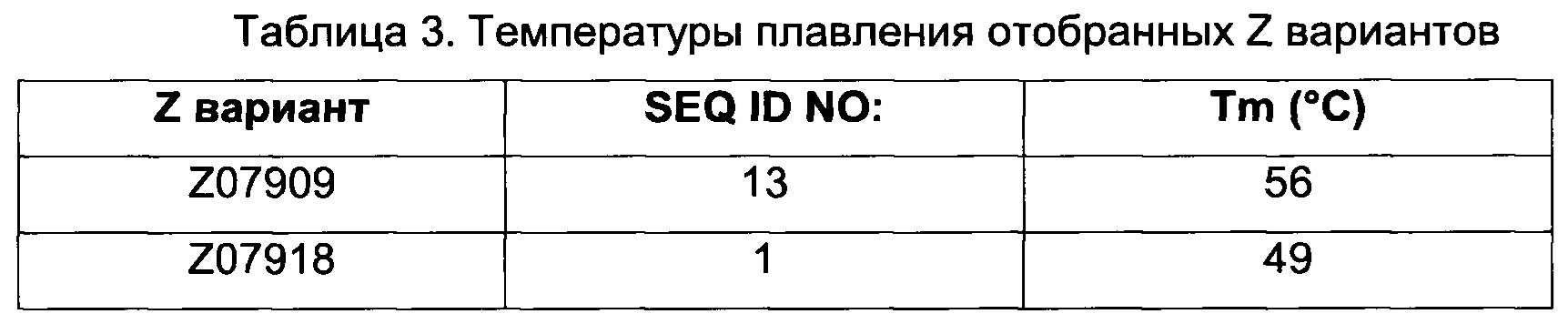
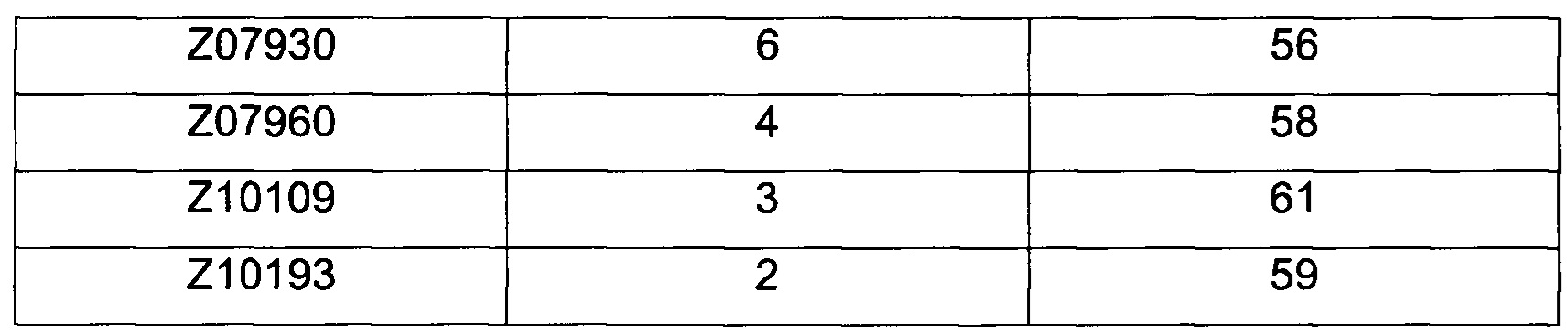
Анализ связывания и кинетики посредством Biacore: Связывание семнадцати Z вариантов, несущих His6-метку, с FcRn человека и диссоциацию при различных значениях pH исследовали на приборе Biacore посредством последовательного инъецирования по поверхности чипа с иммобилизованным FcRn каждого из Z вариантов при pH 6,0 и буфера с pH 6,0 или pH 7,4. FcRn человека иммобилизовали на поверхности в качестве лиганда до уровня 1668 RU. Семнадцать Z вариантов демонстрировали связывание с FcRn при pH 6,0,, и для всех вариантов наблюдали более быстрые скорости диссоциации при pH 7,4 по сравнению с pH 6,0. Аналогичные результаты были получены с IgG, скорость диссоциации была быстрее при pH 7,4. Варианты Z07918 и Z10193 демонстрировали кривые, характеризующие наименьшую скорость диссоциации. Сенсограммы для подгруппы вариантов и IgG приведены на Фиг. 2А-Д.
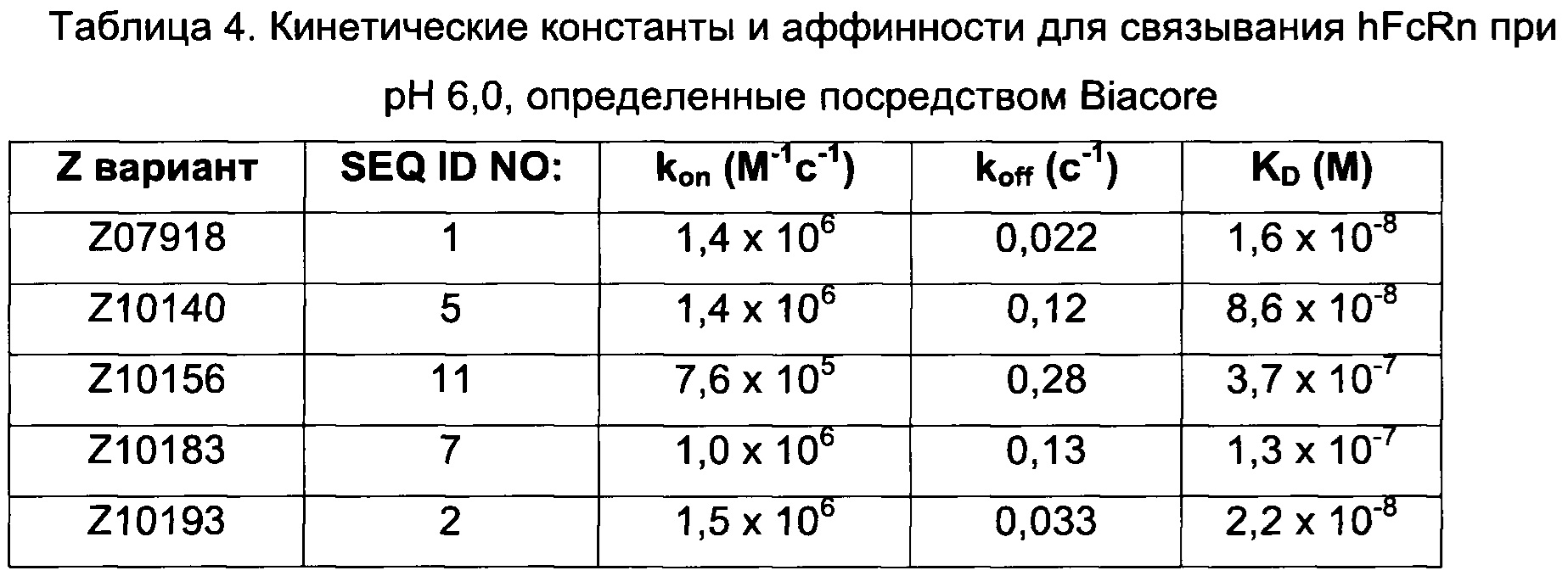
Определили кинетические константы у пяти Z вариантов, взаимодействующих с FcRn при pH 6,0 (см. Таблицу 4). FcRn человека иммобилизовали на поверхности до уровня 2015 RU. Для каждого Z варианта рассчитывали кинетические константы с использованием набора кривых для трех инъецированных концентраций.
Для Z вариантов, несущих His6-метку, Z07918 (SEQ ID NO: 1), Z07960 (SEQ ID NO: 4) и Z10193 (SEQ ID NO: 2), также определяли константы аффинности (KD) при взаимодействии с человеческим и мышиным FcRn при pH 6,0 и pH 7,4 (Таблица 5). У всех трех вариантов значения KD при pH 6,0 были ниже, чем при pH 7,4.

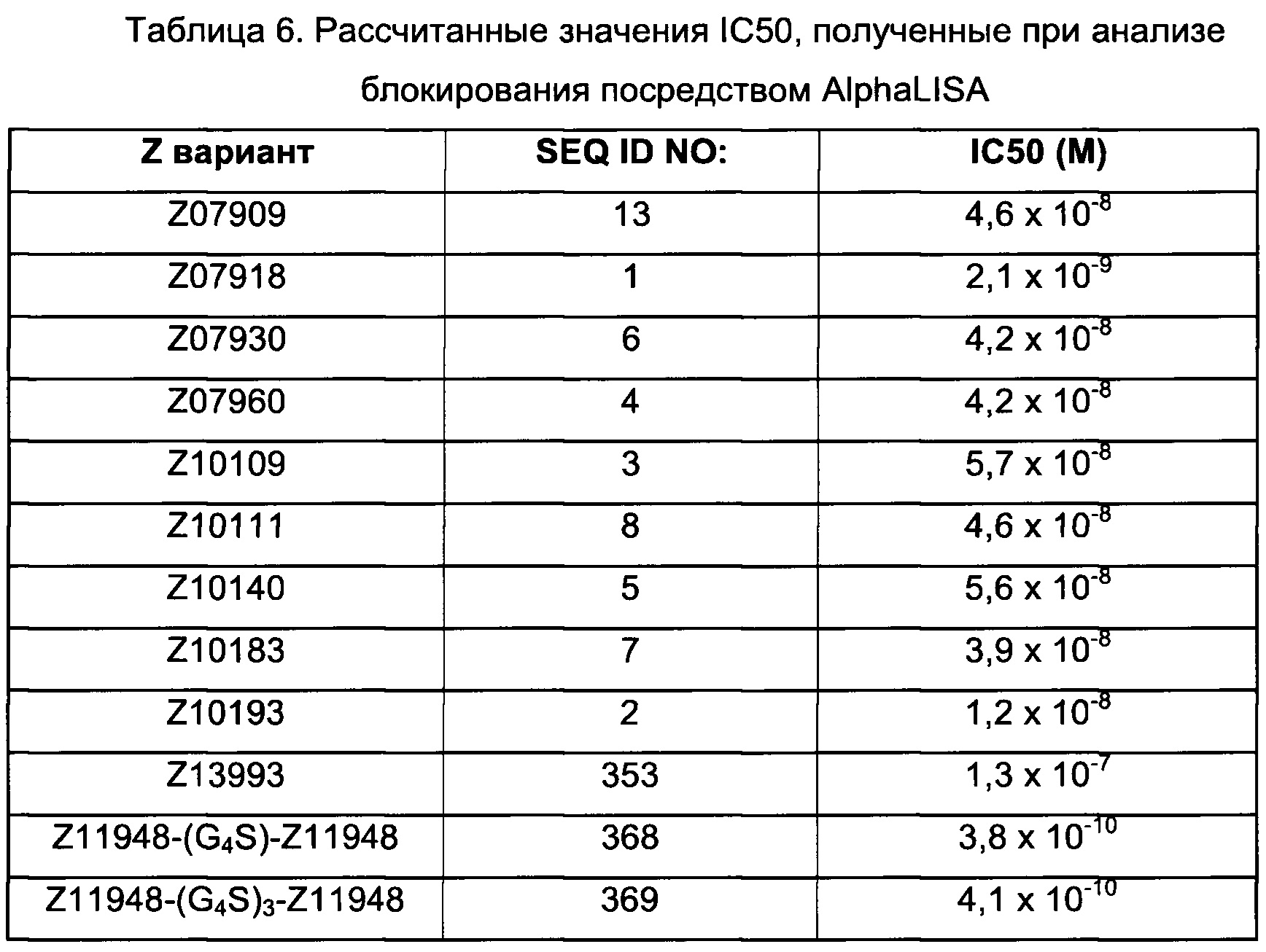
Анализ блокирования посредством AlphaLISA: Способность семнадцати мономерных Z вариантов, несущих His6-метку (SEQ ID NO: 1-16 и SEQ ID NO: 353), и двух димерных вариантов, Z11948-G4S-Z11948 и Z11948-(G4S)3-Z11948, ингибировать связывание IgG с FcRn тестировали с помощью анализа блокирования AlphaLISA. Серийные разведения вариантов Z инкубировали с биотинилированым FcRn человека, и блокирующую способность каждого соответствующего варианта измеяли после добавления гранул-акцепторов, покрытых IgG, и затем гранул-доноров, покрытых стрептавидином. Ингибирование можно было измерить как снижение сигнала AlphaLISA для положительных Z вариантов. Значения IC50, рассчитанные для десяти мономерных вариантов и двух димерных вариантов, продемонстрировавших блокирование связывание IgG с FcRn в данном исследовании, приведены в Таблице 6.
Пример 4
Связывание Z вариантов, связывающих FcRn, с клетками HeLa, трансфицированными человеческим или мышиным FcRn/eGFP
В данном Примере исследовали связывающую способность Z вариантов, связывающих FcRn. Описано получение клеток HeLa, экспрессирующих трансгенный ген человеческого или мышиного FcRn-eGFP, и использование этих клеток в анализе посредством проточной цитометрии с Z вариантами, меченными Alexa Fluor® 647.
Материалы и методы
Клонирование вирусных векторов, несущих FcRn-eGFP и В2М Гены, кодирующие FcRn мыши (mFcRn, Genbank ВС003786.1, OpenBiosystems) и В2М мыши (mB2M, Genbank ВС085164.1, OpenBiosystems) амплифицировали аналогично генам FcRn человека и В2М человека, как описано в Примере 1. Гены FcRn и В2М человека и мыши амплифицировали следующим образом: в случае hFcRn амплифицировали последовательность, кодирующую аминокислоты 1-365 (SEQ ID NO: 382); в случае hB2M амплифицировали последовательность, кодирующую аминокислоты 21-119 (SEQ ID NO: 380); в случае mFcRn амплифицировали последовательность, кодирующую аминокислоты 1-369 (SEQ ID NO: 383); а в случае mB2M амплифицировали последовательность, кодирующую аминокислоты 21-119 (SEQ ID NO: 381). Вектор pHR-cPPT-CMV-EGFP (Jakobsson et al. (2003) J Neurosci Res 73:876-85) и ПЦР-ампликоны FcRn (человеческого и мышиного) разрезали с помощью рестрикционных ферментов BamHI (человеческий) или BclI (мышиный) и MluI (New England Biolabs, кат. номера R0136M, R0160L и R0198L, соответственно) и лигировали с помощью ДНК-лигазы Т4 (New England Biolabs, кат. номер М0202М). Лигирующей смесью химически трансформировали Е. coli RRIΔM15 и производили посев на чашках с ампициллином. Выбирали колонии и производили скрининг с помощью подходящих пар праймеров. Конструкции, кодирующие исходный сигнальный пептид, FcRn человека или мыши и eGFP в цитоплазматическом «хвосте», подтверждали с помощью секвенирования и обозначали как pHR-cPPT-CMV-hFcRn-eGFP и pHR-cPPT-CMV-mFcRn-eGFP, соответственно.
ПЦР-ампликоны В2М человека и мыши встраивали в плазмиду pDONOR221 (Invitrogen, кат. номер 12536-017) при помощи рекомбинации с использованием системы Gateway (Invitrogen, кат. номер 11789020, смесь ферментов Gateway® BP Clonase® II) согласно рекомендациям производителя. После подтверждения правильных последовательностей В2М человека или мыши встраивали в p2k7_gtc (Suter et al., см. выше), многосайтовой системы клонирования Gateway (Invitrogen, кат. номер 11791020, смесь ферментов Gateway® LR Clonase® II) вместе с содержащей промотор плазмидой pENTR-CMV (Tai et al., см. выше), с получением векторов 2k7neo-CMV-hB2M и 2k7neo-CMV-mB2M, соответственно.
Лентивирусная трансдукция клеток HeLa: Осуществляли сотрансфекцию клеток HEK293T парами векторов 2k7neo-CMV-hB2M и pHR-cPPT-CMV-hFcRn-eGFP или 2k7neo-CMV-mB2M и pHR-cPPT-CMV-mFcRn-eGFP вместе с оболочкой VSV-G и пакующей плазмидой gag/pol, с использованием трансфекции с хлоридом кальция (Zufferey et al., см. выше; Jakobsson et al. (2006, см. выше). Супернатанты культуры HEK293T, содержащие сформированные лентивирусные частицы лентивирусов с трансгенами FcRn и В2М, соответственно, использовали для последовательной трансдукции клеток аденокарциномы шейки матки HeLa (Cell Line Service) при малом числе пассажей. Полученные две стабильно трансдуцированные клеточные линии HeLa далее обозначены как hFcRn-eGFP (трансдуцированы генами человеческих FcRn-eGFP и hB2M) и mFcRn-eGFP (трансдуцированы генами мышиных FcRn-eGFP и mB2M).
Мечение Z вариантов, связывающих FcRn, посредством Alexa Fluor® 647 Три Z варианта, несущие His6-метку, Z07918, Z07930 и Z07960, метили сложным сукцинимидиловым эфиром карбоновой кислоты Alexa Fluor® 647 (Invitrogen, кат. номер А20106). Перед мечением производили замену буфера на 0,2 М карбонатный буфер, pH 8,3, используя центрифужные фильтрующие элементы Vivaspin500 (MWCO 10 кДа, Vivaproducts, кат. номер 512-2838) и центрифугируя при 10000 g. Мечение осуществляли в Vivaspin500 и добавляли 1 мкл красителя - сложного сукцинимидилового эфира Alexa647 (40 мкг/мкл в DMSO (диметилсульфоксид), что соответствовало 1,3 × молярному избытку) к 200 мкг/25 мкл Z варианта. Смеси инкубировали при комнатной температуре в темноте в течение 40 минут в покачивающем ротационном смесителе. Затем реакционные смеси помещали на лед на 3,5 часа и удаляли свободный краситель путем промывания 15×100 мкл PBS в Vivaspin500.
Иммунофлуоресцентное окрашивание клеток HeLa, трансфицированных человеческим и мышиным FcRn-eGFP, FcRn-связывающими Z вариантами: клетки HeLa, трансфицированные hFcRn-eGFP и mFcRn-eGFP, собирали посредством трипсинизации и перед подсчетом дважды отмывали PBS с pH 6,0. Переносили пипеткой по 100000 клеток на лунку 96-луночного планшета с v-образной формой дна лунок (Nunc, кат. номер 277143) и затем осаждали клетки в планшете при 1700 об/мин в течение 4 мин при 4°C. Супернатанты удаляли и фиксировали клетки 50 мкл 2% формальдегида (Sigma Aldrich, кат. номер F8775) в PBS при pH 6,0 в течение 10 мин при комнатной температуре. Затем клетки промывали 2×100 мкл PBS, pH 6,0, насыщенным казеином (PBSC), и ресуспендировали в PBSC с добавлением 0,1% сапонина (AppliChem, кат. номер А4518.0100), содержащем 620 нМ меченных Alexa Fluor® 647 Z вариантов, несущих His6-метку, Z07960, Z07930 и Z07918. Трансдуцированные клетки HeLa, которые инкубировали только с буфером, использовали в качестве контроля. Клетки инкубировали в течение 1 ч при 8°C на шейкере в темноте, промывали 2×100 мкл PBSC и ресуспендировали в 180 мкл PBS, pH 6,0, содержащем 1% BSA (фракция V, Merck, кат. номер 1.12018.0100). 10000 клеток/лунку анализировали на проточном цитометре Gallios (Beckman Coulter) и анализировали данные с помощью программы Kaluza (Beckman Coulter).
Результаты
Анализ посредством проточной цитометрии использовали для определения того, могли ли Z варианты, связывающиеся с FcRn, связываться с человеческим и/или мышиным FcRn на клетках HeLa, трансдуцированных человеческим или мышиным FcRn/eGFP. Эксперимент проводили при pH 6,0 с Z07960, Z07930 и Z07918 (SEQ ID NO: 4, 6 и 1, соответственно), меченными Alexa Fluor® 647. Анализ точечной диаграммы (ось у: Alexa Fluor® 647, ось х: eGFP) показал, что популяцию трансдуцированных клеток можно было разделить на FcRn-eGFP-отрицательную и положительную популяции (Фиг. 3, гейты Н и I, соответственно), что указывало на гетерогенную экспрессию слитого белка FcRn-eGFP клетками HeLa (Фиг. 3). Соответственно, из значений средней интенсивности флуоресценции (MFI) для Alexa Fluor® 647 в гейте I вычитали фоновые значения MFI Alexa Fluor® 647 в гейте Н. Рассчитанные значения MFI представлены на Фиг. 4. Результаты показывают, что Z07960, Z07930 и Z07918 способны связывать клетки HeLa, экспонирующие человеческий (Фиг. 4А) или мышиный (Фиг. 4Б) FcRn-eGFP.
Пример 5
Блокирование связывания IgG с FcRn с помощью FcRn-связывающего Z варианта Z07918
В данном Примере в исследовании на клетках изучали возможную конкуренцию за связывание FcRn между FcRn-связывающими Z вариантами и IgG. Такое связывание приведет к блокированию взаимодействия IgG-FcRn.
Материалы и методы
Блокирование иммунофлуоресцентного окрашивания IgG-FcRn: Трансдуцированные человеческим или мышиным FcRn-eGFP клетки HeLa получали, как описано в Примере 4. Фиксированные клетки ресуспендировали в 50 мкл смеси 100 нМ конъюгированных с Alexa Fluor® 647 человеческих или мышиных IgG (Jackson laboratories, кат. номер 009-600-003 и 015-600-003, соответственно) и 1000, 100, 10, 1 или 0 (контроль - буфер) нМ Z07918 с His6-меткой, разведенного в PBS с казеином, pH 6,0, содержащем 0,1% сапонин (AppliChem). Клетки инкубировали в течение 1 ч при 37°C на шейкере в темноте, отмывали 2×100 мкл PBS с казеином, pH 6,0 и ресуспендировали в 180 мкл PBS, pH 6,0, содержащем 1% BSA. Данные для 10000 клеток/лунку (за исключением несколько меньшего числа клеток в случае 100 нМ мышиного mIgG-Alexa Fluor® 647) получали на проточном цитометре Gallios (Beckman Coulter) и анализировали данные с помощью программы Kaluza (Beckman Coulter).
Результаты
Эксперимент проводили для определения того, блокирует ли FcRn-связывающий Z вариант Z07918 (SEQ ID NO: 1) взаимодействие IgG-FcRn. Трансдуцированные человеческим или мышиным FcRn-eGFP клетки HeLa инкубировали с IgG человека или мыши, конъюгированными с Alexa Fluor® 647. Связывание блокировали немеченным Z07918 в различных концентрациях. Вследствие гетерогенной экспрессии FcRn трансдуцированными клетками HeLa (описано в Примере 4), из значений MFI Alexa Fluor® 647 в гейте N каждого образца вычитали соответствующие значения MFI в гейте М (Фиг. 5). Процент связывания IgG Alexa Fluor® 647 рассчитывали путем деления разных значений MFI на MFI в пустом контроле. Результаты показали, что Z07918 эффективно блокировал связывание hIgG с hFcRn (Фиг. 6А) дозозависимым образом. Кроме того, Z07918 также блокировал связывание mIgG с mFcRn (Фиг. 6Б), хотя и менее эффективно по сравнению со связыванием hIgG.
Пример 6
Фармакокинетическое исследование трех Z вариантов, связывающих FcRn
В данном Примере исследовали способность Z вариантов, связывающих FcRn, пролонгировать период полувыведения в сыворотке неспецифических Z вариантов в фармакокинетическом исследовании, проведенном на мышах. Материалы и методы
Субклонирование Z вариантов: Подгруппу Z вариантов (Z07918, Z07960 и Z10193) подвергали второму субклонированию. В качестве матрицы использовали ДНК субклонированных в Примере 3 вариантов, несущих His6-метку. Вначале выполняли ПЦР-амплификацию с использованием соответствующих пар праймеров для создания генов, кодирующих Z варианты, начинающиеся с аминокислот АЕ вместо VD. Мутированные Z варианты представлены на Фиг. 1 и обозначены Z11948 (SEQ ID NO: 354), Z11946 (SEQ ID NO: 355) и Z11947 (SEQ ID NO: 356), что соответствует мутированным Z07918, Z07960 и Z10193, соответственно. Гены, кодирующие новые Z варианты, рестрикционно разрезали и лигировали в вектор, несущий гены, кодирующие альбумин-связывающий вариант РР013 (SEQ ID NO: 377) и Z03638 (SEQ ID NO: 378) со спейсерными последовательностями, в результате получая слияние генов, кодирующее [Z#####]-GAP(G4S)4TS-[PP013]-GT(G4S)4PR-[Z03638] (обозначаемое также "Z#####-PP013-Z03638" или "Z вариант в слиянии с PP013-Z03638"). Молекулу отрицательного контроля [Z03638]-GAP(G4S)4TS-[РР013] субклонировали аналогичным образом путем лигирования Z03638 в вектор, содержащий линкер (G4S)4 и последовательность РР013. Последующие стадии трансформации Е. coli вектором осуществляли, как описано в Примере 3.
Культивирование и очистка: Z варианты в слиянии с PP013-Z03638, получали в Е. coli, как описано в Примере 3. Приблизительно 3 г каждого клеточного осадка ресуспендировали в 30 мл буфера TST (25 мМ Tris-HCl, 1 мМ EDTA, 200 мМ NaCl, 0,05% Tween 20, pH 8,0) с добавлением Benzonase® (Merck). После разрушения клеток ультразвуком и просветления путем центрифугирования каждый супернатант наносили на колонку объемом 5 мл, заполненную агарозой с иммобилизованным анти-ABD лигандом, для гравитационного элюирования (см. WO 2014/064237). После промывания буфером TST и 5 мМ буфером NH4Ac, pH 5,5, Z варианты элюировали 0,1 М НАс. К элюированным фракциям после стадии очистки при помощи аффинной хроматографии на анти-ABD агарозе добавляли ацетонитрил (ACN) в конечной концентрации 10% и загружали образцы на колонку Resource 15RPC (GE Healthcare) объемом 3 мл, предварительно уравновешенную растворителем RPC А (0,1% трифторуксусная кислота (TFA), 10% ACN, 90% вода). После промывания колонки растворителем RPC А элюировали связанный белок линейным градиентом 0-50% растворителя RPC В (0,1% TFA, 80% ACN, 20% вода) в объеме 60 мл. Фракции, содержащие очищенный Z вариант, идентифицировали при помощи анализа SDS-PAGE и объединяли. После очистки с помощью RPC заменяли буфер в объединенных фракциях на PBS, используя обессоливающую колонку HiPrep 26/10 (GE Healthcare). Наконец, очищали Z варианты на колонках EndoTrap red объемом 1 мл (Hyglos, кат. номер 321063) для обеспечения низкого содержания эндотоксина.
Концентрацию белка, чистоту и идентичность каждого очищенного Z варианта анализировали, как описано в Примере 3.
Анализ Biacore: Экспрессированные и очищенные Z варианты, слитые с PP013-Z03638, исследовали против FcRn человека при pH 6,0 по существу, как описано для кинетического анализа в Примере 3. Z варианты и отрицательный контроль Z03638-PP013 инъецировали в концентрациях 40 нМ, 160 нМ и 640 нМ в течение 1 минуты с последующей диссоциацией в течение 2,5 минут и уравновешиванием в течение 1 минуты. Кинетические константы и аффинности для Z вариантов определяли с использованием программы BiaEvaluation.
Фармакокинетическое исследование: Z11947, Z11946 и Z11948, слитые с PP013-Z03638, вводили внутривенно (в/в) самцам мышей NMRI (Charles River, Germany) в дозе 92 нмоль/кг массы тела. Сыворотку у групп из трех мышей получали через 0,08, 6, 18, 78, 120, 168 и 240 часов. Концентрацию соответствующего Z варианта определяли при помощи ELISA.
ELISA: Половину площади 96-луночных планшетов для ELISA покрывали 50 мкл/лунку Z-специфического антитела козы (собственного производства), разведенного до 4 мкг/мл в буфере для покрытия (50 мМ карбоната натрия, pH 9,6) при 4°C в течение ночи. Раствор антитела сливали и блокировали лунки 100 мкл PBSC в течение 1,5 ч при комнатной температуре. Сыворотку разводили в PBSC, содержащем 1% мышиную сыворотку (матрикс), от 1:100 до 1:51 200 в серии двукратных разведений в планшете для разведения. В каждый планшет вносили раститрованные стандартным образом соответствующие Z варианты и четыре контроля качества (очень низкий, низкий, средний и высокий контроли), разведенные в матриксе. В каждую лунку переносили по 50 мкл разведения и инкубировали планшеты для ELISA в течение 1,5 ч при комнатной температуре. Планшеты промывали четыре раза PBST. Связанные Z варианты выявляли, добавляя 50 мкл/лунку кроличьих Ig против РР013 (собственного производства), разведенных до 4 мкг/мл в PBSC. Затем планшеты инкубировали в течение 1,5 ч при комнатной температуре с последующими отмывками, как описано выше. Добавляли конъюгированное с HRP противокроличье антитело осла, приобретенное у Jackson laboratories (кат. номер 711-035-152), разведенные 1:20000 в PBSC и планшеты инкубировали в течение 1 ч. После отмывки, как описано выше, в лунки добавляли 50 мкл субстрата ImmunoPure ТМВ и планшеты проявляли согласно рекомендациям производителя. После развития окраски в течение 15 минут измеряли поглощение при 450 нм с помощью считывающего устройства для многолуночных планшетов (Victor3). Значения поглощения анализировали с помощью программы GraphPad Prism 5 для определения концентраций (аппроксимация кривой посредством кубического сплайна) и площади под кривой (AUC). Затем концентрации откладывали на графике в виде их натуральных логарифмов относительно времени. Полученные кривые соответствовали двухкомпартментной модели, и рассчитывали терминальный период полувыведения как In2, разделенный на наклон, на основе последних трех моментов времени.
Результаты
Культивирование и выделение: В Е. coli получили три FcRn-связывающих Z варианта, Z11947, Z11946 и Z11948 (SEQ ID NO: 356, 355 и 354), сконструированные как Z#####-PP013-Z03638, и отрицательный контроль Z03638-PP013. Количество очищенного белка из приблизительно 3 г бактериальных осадков, определенное спектрофотометрически посредством измерения поглощения при 280 нм, у различных Z вариантов, связывающих FcRn, варьировало приблизительно от 10 мг до 25 мг. Анализ каждого полученного препарата белка с помощью SDS-PAGE показал, что они преимущественно содержали соответствующий FcRn-связывающий Z вариант. Точную молекулярную массу каждого Z варианта, связывающего FcRn, подтверждали с помощью LC/MS анализа.
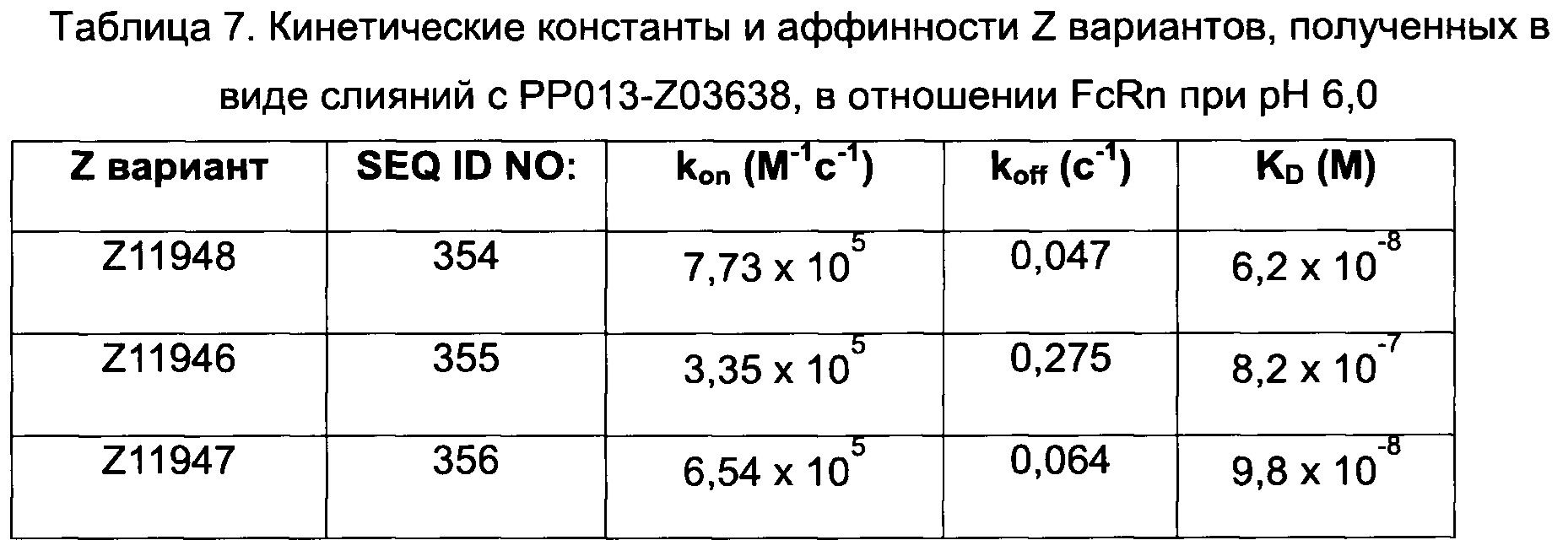
Анализ Biacore: Связывание с FcRn анализировали для трех Z вариантов, слитых с PP013-Z03638. FcRn человека иммобилизовали на поверхности до уровня 548 RU. Полученные приблизительные кинетические константы и аффинности связывания с мишенью при pH 6,0 приведены в Таблице 7. Аппроксимированные кривые приведены на Фиг. 7А-В. Отрицательный контроль Z03638-PP013 был отрицательным в отношении FcRn.
Фармакокинетическое исследование: В фармакокинетическом исследовании на мышах сравнивали фармакокинетические профили указанных выше конструкций Z вариантов, слитых с PP013-Z03638, и отрицательного контроля Z03638-PP013. В предыдущей работе, например как описано в заявке РСТ WO 2009/016043, было показано, что слитые с ABD белки имеют длительный период полувыведения в сыворотке благодаря связыванию ABD с сывороточным альбумином. Конечный период полувыведения молекулы Z варианта, слитого с ABD (Z03638-PP013), составлял приблизительно 43 часа, что сопоставимо с периодом полувыведения мышиного альбумина (35 часов), что согласовывалось полученными ранее результатами. Конечные периоды полувыведения конструкций, содержащих, помимо ABD, молекулу FcRn-связывающего Z варианта, были в два-три раза дольше (Фиг. 8). Рассчитанные конечные периоды полувыведения составили 99 часов (Z11947), 69 часов (Z11946) и 58 часов (Z11948), что позволило предположить, что связывание Z вариантов с FcRn способствовало увеличению периодов полувыведения.
Пример 7
Дизайн и конструирование библиотеки созревания FcRn-связывающих Z вариантов
В данном Примере сконструировали библиотеку созревания. Библиотеку использовали для отбора FcRn-связывающих Z вариантов. Полагают, что отбор из библиотек созревания обычно приводит к связывающим агентам с увеличенной аффинностью (Orlova et al., (2006) Cancer Res 66(8): 4339-48). В данном исследовании получали рандомизированные одноцепочечные линкеры посредством синтеза по принципу смешивания и разделения, позволяющего включить в синтез определенные кодоны в нужных положениях.
Материалы и методы
Дизайн библиотеки: Основу библиотеки составили шестнадцать последовательностей FcRn-связывающих Z вариантов, приведенных в Таблице 1 и дополнительно описанных в Примерах 2-6. В новой библиотеке 13 вариабельных положений в каркасе Z молекулы изменяли в направлении определенных аминокислотных остатков согласно стратегии, основанной, главным образом, на мотивах связывания Z вариантов, представленных SEQ ID NO: 1-16. ДНК-линкер, созданный при помощи синтеза по принципу смешивания и разделения, содержал 147 п.о. (пар оснований) частично рандомизированных спиралей 1 и 2 с последовательностью: 

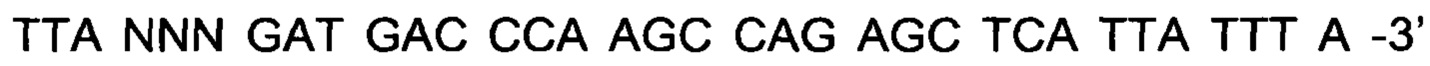 (SEQ ID NO: 388; рандомизированные кодоны представлены NNN), фланкированной рестрикционными сайтами XhoI и SacI, и был заказан в компании DNA 2.0 (Menlo Park, СА, USA). Теоретическое распределение аминокислотных остатков в новой библиотеке, включая восемь вариабельных аминокислотных положений (9, 10, 11, 13, 14, 24, 32 и 35) и пять константных аминокислотных положений (17, 18, 25, 27 и 28) в каркасе Z молекулы, приведено в Таблице 8. Теоретический размер полученной библиотеки составил 5,3×108 вариантов.
(SEQ ID NO: 388; рандомизированные кодоны представлены NNN), фланкированной рестрикционными сайтами XhoI и SacI, и был заказан в компании DNA 2.0 (Menlo Park, СА, USA). Теоретическое распределение аминокислотных остатков в новой библиотеке, включая восемь вариабельных аминокислотных положений (9, 10, 11, 13, 14, 24, 32 и 35) и пять константных аминокислотных положений (17, 18, 25, 27 и 28) в каркасе Z молекулы, приведено в Таблице 8. Теоретический размер полученной библиотеки составил 5,3×108 вариантов.
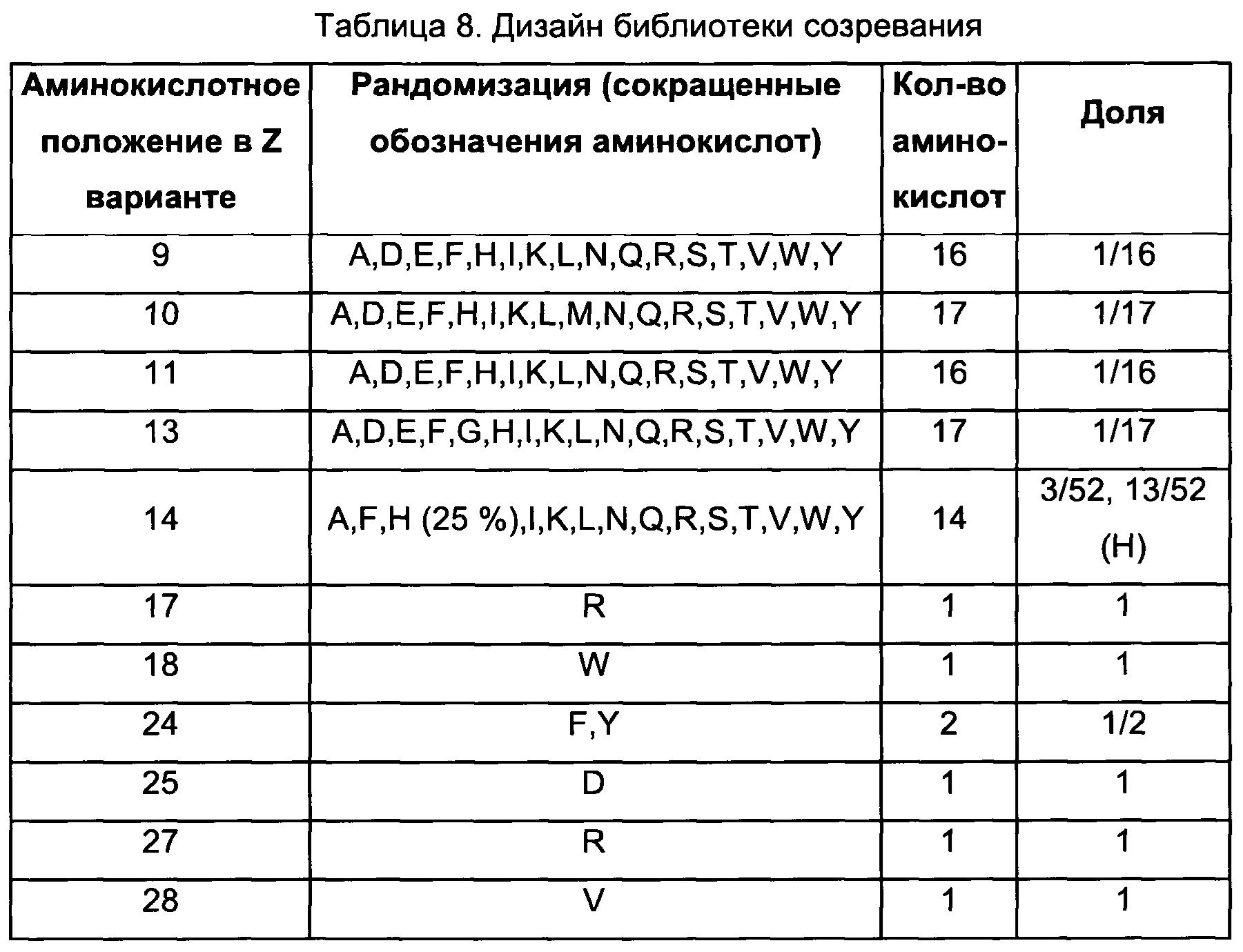
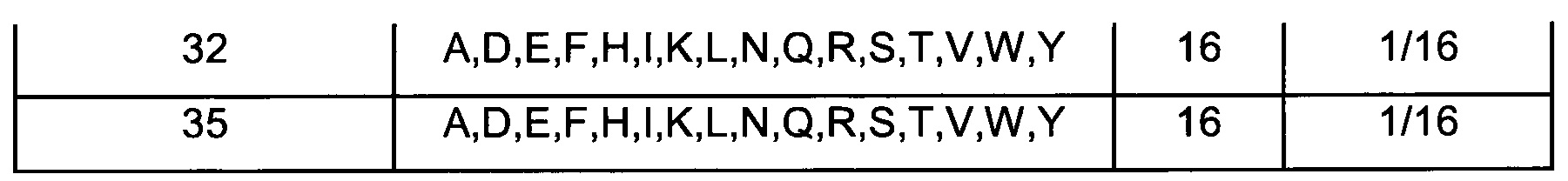
Конструирование библиотеки: Библиотеку амплифицировали с использованием полимеразы AmpliTaq Gold (Applied Biosystems, кат. номер 4311816) на протяжении 12 циклов ПЦР и объединенные продукты ПЦР очищали с помощью набора QIAquick PCR Purification Kit (QIAGEN, кат. номер 28106) согласно рекомендациям поставщика. Пул очищенных рандомизированных фрагментов библиотеки разрезали рестрикционными ферментами XhoI и SacI-HF (New England Biolabs, кат. номер R0146L, и кат. номер R3156M) и концентрировали с помощью набора для очистки продуктов ПЦР. Затем выполняли препаративный электрофорез продукта в 2,5% агарозном геле (Nuisieve GTC agarose, Cambrex, Invitrogen) и очищали с использованием набора для экстракции из гела QIAGEN (QIAGEN, кат. номер 28706) согласно рекомендациям поставщика.
Фагмидный вектор pAY02592 (по существу как pAffi1, описанный  et al., см. выше) разрезали теми же ферментами, очищали посредством фенол-хлороформной экстракции и преципитации этанолом. Полученные в результате рестрикции фрагменты и вектор лигировали в молярном отношении 5:1 с помощью ДНК-лигазы Т4 (Fermentas, кат. номер EL0011) в течение 2 ч при комнатной температуре с последующей инкубацией при 4°C в течение ночи. Лигированную ДНК выделяли посредством фенол-хлороформной экстракции и преципитации этанолом с последующим растворением в 10 мМ Tris-HCl, pH 8,5. Полученная таким образом библиотека в векторе pAY02592 кодировала Z варианты, каждый из которых был слит с альбумин-связывающим доменом (ABD), происходящим из белка G стрептококка.
et al., см. выше) разрезали теми же ферментами, очищали посредством фенол-хлороформной экстракции и преципитации этанолом. Полученные в результате рестрикции фрагменты и вектор лигировали в молярном отношении 5:1 с помощью ДНК-лигазы Т4 (Fermentas, кат. номер EL0011) в течение 2 ч при комнатной температуре с последующей инкубацией при 4°C в течение ночи. Лигированную ДНК выделяли посредством фенол-хлороформной экстракции и преципитации этанолом с последующим растворением в 10 мМ Tris-HCl, pH 8,5. Полученная таким образом библиотека в векторе pAY02592 кодировала Z варианты, каждый из которых был слит с альбумин-связывающим доменом (ABD), происходящим из белка G стрептококка.
Продукты реакции лигирования (приблизительно 160 нг ДНК/трансформацию) использовали для электропорации электрокомпетентных клеток Е. coli ER2738 (50 мкл, Lucigen, Middleton, WI, USA). Немедленно после электропорации добавляли приблизительно 1 мл среды для выделения (поставляемой с клетками ER2738). Трансформированные клетки инкубировали при 37°C в течение 60 мин. Отбирали образцы для титрования и определения количества трансформантов. Затем клетки объединяли и культивировали в течение ночи при 37°C в 1 л среды TSB-YE с добавлением 2% глюкозы, 10 мкг/мл тетрациклина и 100 мкг/мл ампициллина. Клетки осаждали в течение 7 мин при 4000 g и ресуспендировали в растворе PBS/глицерин (приблизительно 40% глицерина). Клетки аликвотировали и хранили при -80°C. Клоны из библиотеки Z вариантов секвенировали для того, чтобы подтвердить содержание, и для оценки результатов, полученных с использованием сконструированной библиотеки, в зависимости от дизайна библиотеки. Секвенирование осуществляли, как описано в Примере 1, и подтвердили распределение аминокислот.
Получение резерва фагов: Резерв фагов, содержащих фагмидную библиотеку, получали в ферментере объемом 20 л (Belach Bioteknik). Клетки из резерва в глицерине, содержащие фагмидную библиотеку, инокулировали в 10 л TSB-YE (триптический соевый бульон-дрожжевой экстракт; 30 г/л TSB, 5 г/л дрожжевого экстракта) с добавлением 1 г/л глюкозы, 100 мг/л ампициллина и 10 мг/л тетрациклина. Когда клетки достигали оптической плотности 0,6 при 600 нм (OD600), приблизительно 1,5 л культуры инфицировали хелперным фагом M13K07 в 5 × молярном избытке. Клетки инкубировали в течение 30 мин, после чего ферментер заполняли до 10 л комплексной средой для ферментации [2,5 г/л (NH4)2SO4; 5,0 г/л дрожжевого экстракта; 30 г/л триптона, 2 г/л K2HPO4; 3 г/л KH2PO4, 1,25 г/л Na3C6H5O7⋅2H2O; пеногаситель Breox FMT30 0,1 мл/л]. Добавляли следующие компоненты: 10 мл карбенициллина 25 мг/мл; 5 мл канамицина 50 мг/мл; 1 мл 1 М изопропил-β-D-1-тиогалактопиранозида (IPTG); 17,5 мл/л 300 г/л MgSO4 и 5 мл раствора следовых элементов [35 г/л FeCl3⋅6H2O; 10,56 г/л ZnSO4⋅7H2O; 2,64 г/л CuSO4⋅5H2O; 13,2 г/л MnSO4⋅H2O; 13,84 г/л CaCl2⋅2H2O, растворенные в 1,2 М HCl]. Лимитированное по глюкозе культивирование с подпиткой начинали при подаче в реактор раствора глюкозы 600 г/л (3,5 г/ч на старте, 37,5 г/ч через 20 ч и до окончания культивирования). Поддерживали pH на уровне pH 7 посредством автоматического добавления 25% NH4OH, обеспечивали приток воздуха (5 л/мин) и устанавливали скорость перемешивания 500 об/мин. Через 24 ч культивирования с подпиткой OD600 составляла 33,2. Клетки в культуре осаждали путем центрифугирования при 15900 g. Фаговые частицы осаждали из надосадочной жидкости дважды в PEG/NaCl, фильтровали и растворяли PBS и глицерине, как в Примере 2. Резерв фагов хранили при -80°C до применения в отборе.
Результаты
Конструирование библиотеки: В основу новой библиотеки легла группа 16 FcRn-связывающих Z вариантов с подтвержденными свойствами связывания (Примеры 2-6). Теоретический размер разработанной библиотеки составлял 5,3×108 Z вариантов. Фактический размер библиотеки, установленный путем титрования после трансформации клеток Е. coli ER2738, составил 4,5×109 трансформантов.
Качество библиотеки проверяли путем секвенирования 96 трансформантов и сравнения их фактических последовательностей с теоретическими. Содержимое фактической библиотеки по сравнению с теоретической библиотекой оказалось удовлетворительным. Таким образом, была успешно сконструирована библиотека зрелых потенциальных агентов, связывающихся с FcRn.
Пример 8
Отбор и скрининг из библиотеки зрелых Z вариантов
Материалы и методы
Отбор зрелых Z вариантов, связывающих FcRn, посредством фагового дисплея: Белки-мишени - человеческий FcRn (Biorbyt, кат. номер orb84388) и мышиный FcRn (Biorbyt, кат. номер orb99076), биотинилировали по существу, как описано в Примере 2, используя биотин в 10 × молярном избытке. Четыре цикла отбора методом фагового дисплея с использованием новой библиотеки молекул Z вариантов, описанной в Примере 7, осуществляли против человеческого FcRn или мышиного FcRn по существу, как описано в Примере 2, за следующими исключениями. Буферы для отбора представляли собой 0,1% буфер PCTG, pH 5,5 (фосфатно-цитратный буфер Мак-Илвейна, pH 5,5, с добавлением 0,1% Tween-20 и 0,1% желатина) или 0,1% буфер PCTG, pH 7,4 (фосфатно-цитратный буфер Мак-Илвейна, pH 7,4, с добавлением 0,1% Tween-20 и 0,1% желатина), соответственно. Перед отбором в буферы добавляли HSA в конечной концентрации 1,5 мкМ. Все пробирки и гранулы, которые использовали в отборе, предварительно блокировали любым из двух разных буферов для отбора. В цикле 1 осуществляли стадию предварительного отбора посредством инкубации фаговых частиц с SA-гранулами в течение 45 минут.
Для захвата комплексов фаг-мишень использовали 1 мг гранулы на 1,1 мкг биотинилированного FcRn человека или 1,6 мкг биотинилированного FcRn мыши. Осуществляли отмывку 0,1% буфером РСТ pH 5,5 или pH 7,4, за исключением цепочек 2-1-2-1 и 2-1-2-2, где использовали 0,1% РСТ с добавлением 25 нМ IgG (Herceptin®) или 10 нМ IgG, соответственно, как показано в Таблице 9.
Пять цепочек (1-5) в цикле 1 разделяли во втором-четвертом циклах, в результате чего получилось семь цепочек (от 1-1 до 5-1) в цикле 2, одиннадцать цепочек (от 1-1-1 до 5-1-1) в цикле 3 и четырнадцать цепочек (от 1-1-1-1 до 5-1-1-1) в цикле 4. Связанные фаговые частицы элюировали, как описано в Примере 2.
Обзор стратегии отбора, описывающий повышенную жесткость в последующих циклах с использованием пониженной концентрации мишени и увеличенного числа промывок, приведен в Таблице 9.
Амплификация фаговых частиц: Амплификацию фаговых частиц между 1 и 2 циклами отбора осуществляли по существу, как описано в Примере 2, за следующими исключениями. Для амплификации фага использовали Е. coli ER2738 и хелперный фаг M13K07 в 5 × избытке. Амплификацию фаговых частиц между 2 и 4 циклами отбора осуществляли посредством инфицирования бактерий в растворе следующим способом. После инфицирования E. coli ER2738 в log фазе фаговыми частицами добавляли TSB с добавлением 2% глюкозы, 10 мкг/мл тетрациклина и 100 мкг/мл ампициллина с последующей инкубацией при вращении в течение 30 мин при 37°C. Затем бактерии инфицировали хелперным фагом M13K07 в 5 × избытке. Инфицированные бактерии осаждали центрифугированием, ресуспендировали в среде TSB-YE с добавлением 100 мкМ IPTG, 25 мкг/мл канамицина и 100 мкг/мл ампициллина и выращивали в течение ночи при 30°C. Выращенные в течение ночи осаждали центрифугированием и дважды преципитировали фаговые частицы в супернатанте буфером PEG/NaCl. Наконец, перед проведением следующего цикла отбора фаговые частицы ресуспендировали в буфере для отбора.
В последнем цикле отбора бактерии в log фазе инфицировали элюатом и разводили перед распределением на чашки с ТВАВ (30 г/л основы триптозного кровяного агара, Oxoid, кат. номер СМO233В) с добавлением 0,2 г/л ампициллина для образования одиночных колоний для использования в скрининге посредством ELISA.
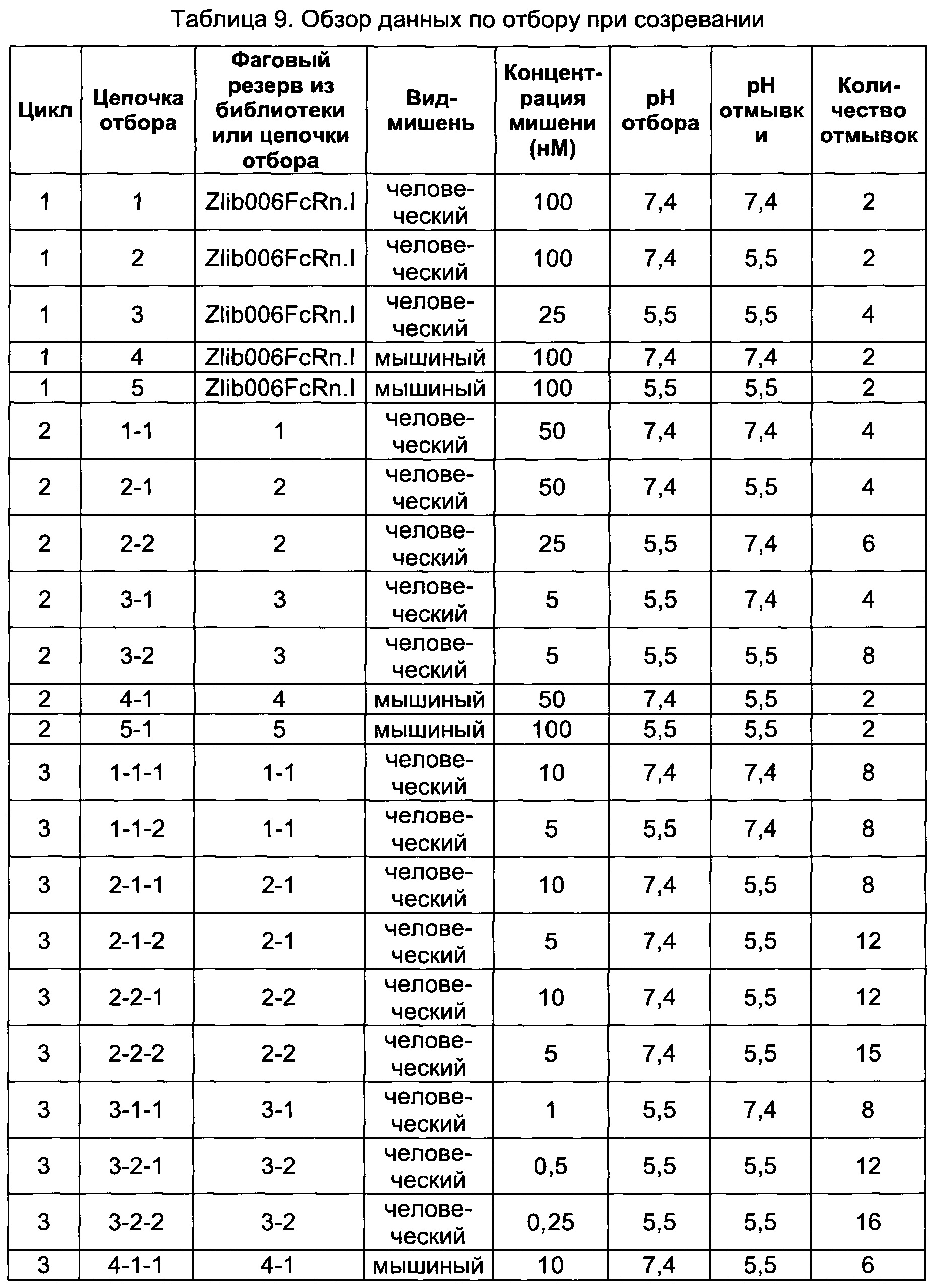

Секвенирование потенциальных связывающих агентов: Для секвенирования отбирали отдельные клоны из различных цепочек отбора. Секвенировали все клоны, участвовавшие в скрининге посредством ELISA. Амплификацию фрагментов генов и секвенирование фрагментов генов осуществляли по существу, как описано в Примере 2.
Скрининг Z вариантов посредством ELISA: Отдельные колонии, содержащие Z варианты (экспрессируемые как Z варианты, слитые с ABD, как описано в Примере 2), выбирали случайным образом из клонов, отобранных из библиотеки зрелой аффинности к FcRn, и культивировали в объеме 1 мл по существу, как описано в Примере 2. Супернатанты периплазматической фракции получали, как описано в Примере 2, в восьми циклах замораживания-оттаивания и использовали периплазматические фракции в скрининге посредством ELISA без разведения. Скрининг посредством ELISA осуществляли как при pH 6,0, так и pH 7,4 по существу, как описано в Примере 2, используя биотинилированный FcRn человека в концентрации 2 нМ на каждую лунку. Периплазматические фракции первичного агента, связывающегося с FcRn, Z10193 (SEQ ID NO: 2; исследованного в предыдущих экспериментах) использовали в качестве положительного контроля. Периплазму, содержащую только ABD-группировку, использовали в качестве отрицательного контроля.
Анализ KD FcRn-связывающих Z вариантов посредством ELISA: Анализировали ответ отобранных агентов, связывающихся с FcRn против серии разведений биотинилированного FcRn человека при помощи ELISA при pH 6,0 и pH 7,4, как описано выше. Биотинилированный FcRn человека добавляли в концентрации 30 нМ и ступенчато разводили 1:3 до 14 пМ. В качестве фонового контроля все Z варианты также исследовали без добавления белка-мишени. Образцы периплазмы, содержащие первичный агент, связывающийся с FcRn, Z07918 (SEQ ID. NO: 1), включали и анализировали в качестве положительного контроля. Периплазму, содержащую только ABD-группировку, использовали в качестве отрицательного контроля. Данные анализировали с использованием GraphPad Prism 5 и нелинейной регрессии и рассчитывали значения KD (полумаксимальная эффективная концентрация).
Результаты
Отбор зрелых Z вариантов, связывающих FcRn, посредством фагового дисплея. Отбор осуществляли в 14 параллельных цепочках, каждая из которых включала четыре цикла. Разные цепочки отбора отличались по концентрации мишени, типу мишени (FcRn человека или FcRn мыши), времени отбора и условиям отмывания.
Секвенирование потенциальных связывающих агентов: Секвенировали случайным образом выбранные клоны. Каждый отдельный Z вариант получал идентификационный номер Z#####, как описано в Примере 2. Всего идентифицировали 445 новых уникальных молекул Z вариантов.
Аминокислотные последовательности подгруппы Z вариантов длиной 58 аминокислотных остатков представлены на Фиг. 1 и в перечне последовательностей как SEQ ID NO: 17-352. Предсказанные мотивы связывания FcRn у данных Z вариантов располагаются между остатком 8 и остатком 36 в последовательностях SEQ ID NO: 17-352. Аминокислотные последовательности полипептидов длиной 49 аминокислотных остатков (BMod), которые, как предсказано, составляют полный трехспиральный пучок в составе каждого из этих Z вариантов, располагаются между остатком 7 и остатком 55.
Скрининг Z вариантов посредством ELISA: Клоны, полученные после четырех циклов отбора, получали в 96-луночных планшетах и проводили скрининг FcRn-связывающей активности с помощью ELISA. Анализировали случайным образом выбранные клоны. Обнаружили, что при pH 6,0 333 из 445 уникальных вариантов Z дают ответ 0,3 AU (единица абсорбции) или выше (соответствующий по меньшей мере 3х негативного контроля) против человеческого FcRn в концентрации 2 нМ. Обнаружили, что при pH 7,4 278 из 445 уникальных вариантов Z дают ответ 0,3 AU или выше (соответствующий по меньшей мере 3х негативного контроля) против человеческого FcRn в концентрации 2 нМ. Клоны, давшие положительный сигнал против FcRn человека, были обнаружены во всех цепочках (включая и те, где белок-мишень был мышиным), за исключением 1-1-1-1. У отрицательных контролей поглощение составило 0,070-0,096 AU (pH 6,0) и 0,060-0,112 AU (pH 7,4), соответственно. Средний ответ пустых контролей составлял 0,070 AU (pH 6,0) и 0,062 (pH 7,4).
Анализ KD FcRn-связывающих Z вариантов посредством ELISA: По результатам эксперимента ELISA, описанного выше, отбирали подгруппу Z вариантов (с наибольшим значением ELISA при pH 6,0 и/или 7,4) и подвергали титрованию с мишенью в формате ELISA. Образцы периплазм инкубировали с серией разведений биотинилированного FcRn человека. В качестве положительного контроля также исследовали образец периплазмы с первичным связывающим агентом - Z07918 (SEQ ID NO: 1). Анализировали полученные значения и рассчитывали их соответствующие значения KD (Таблица 10).
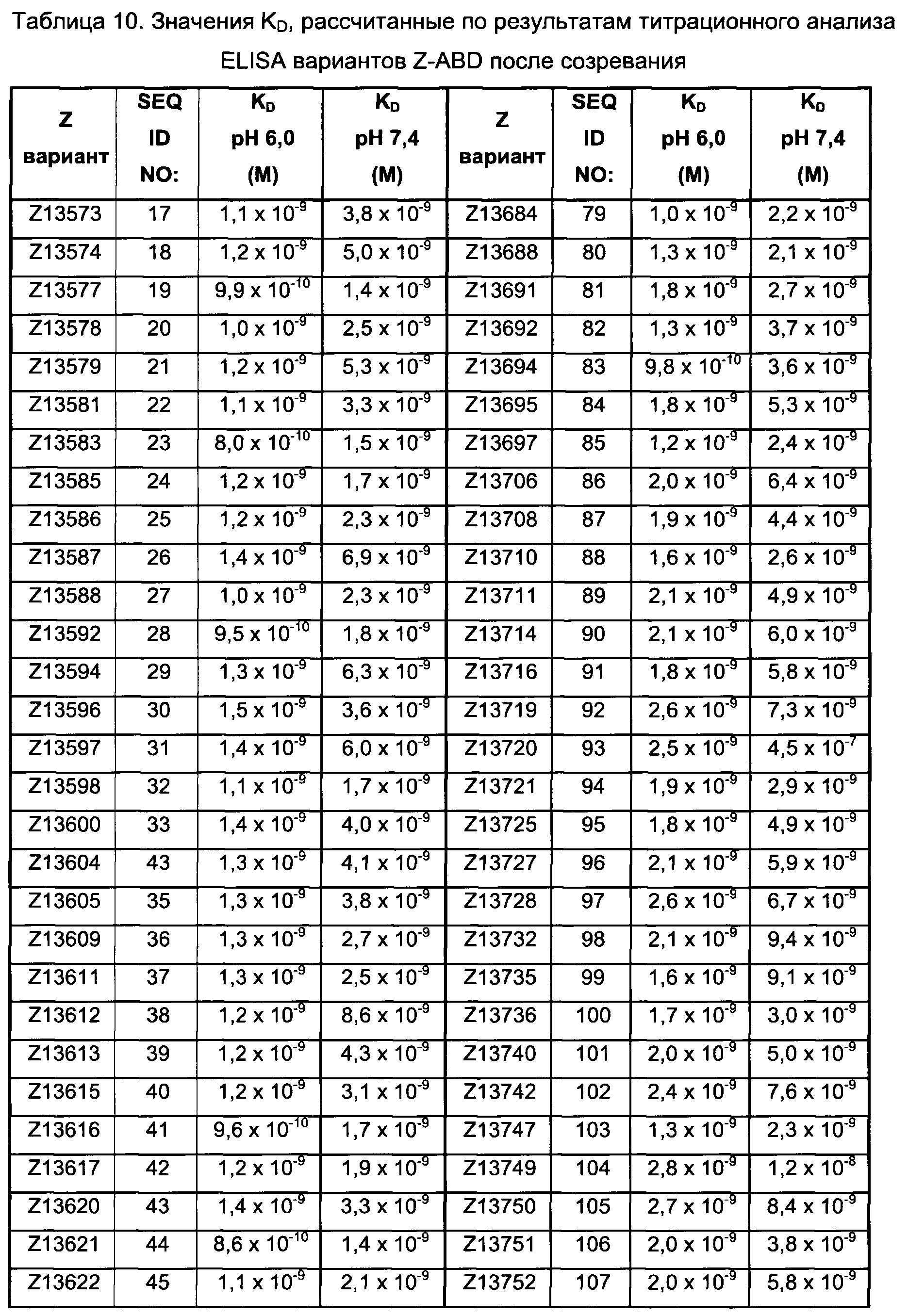
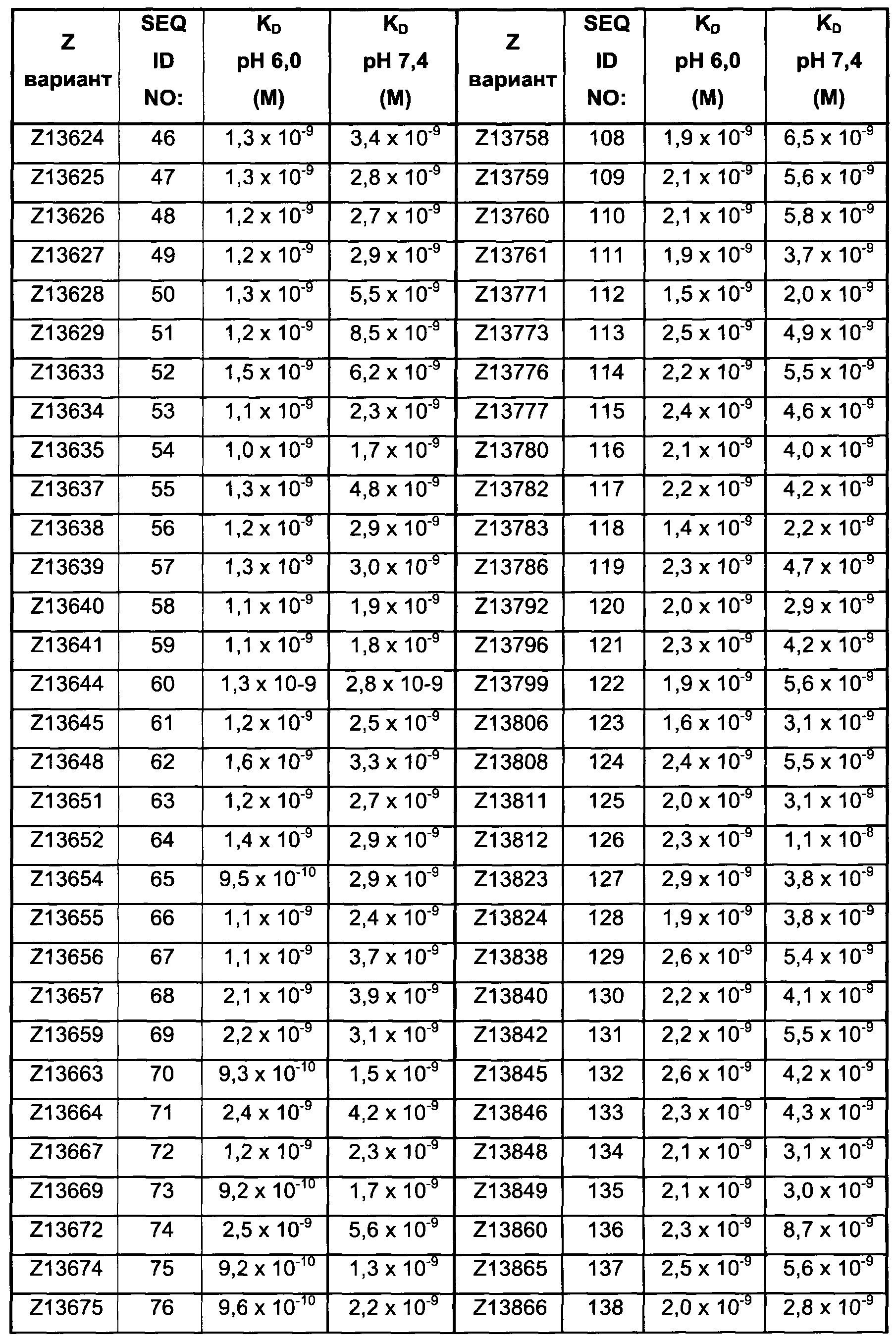
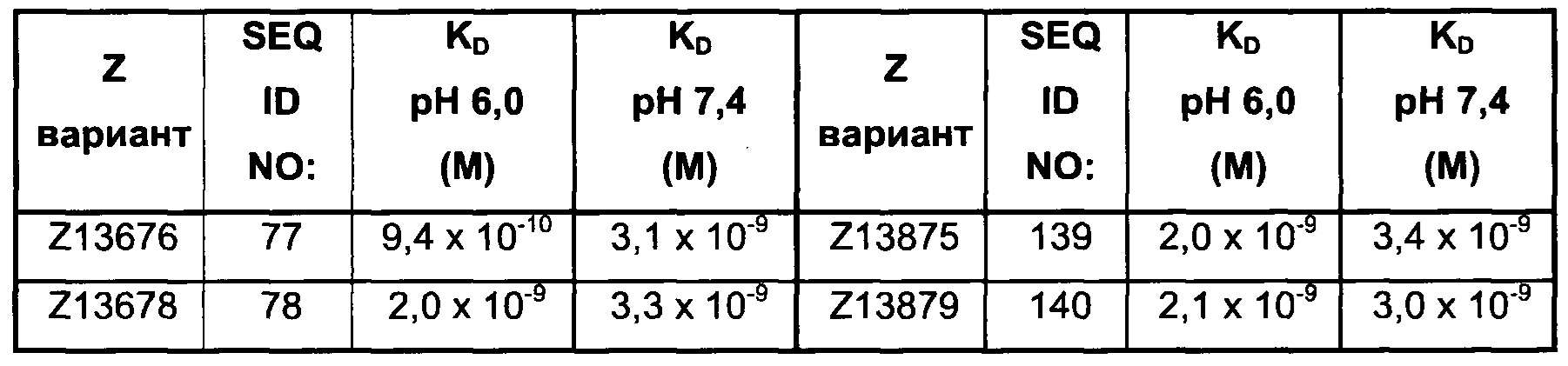
Пример 9
Получение и характеризация Z вариантов из созревшей библиотеки
В данном Примере в Е. coli получили двенадцать Z вариантов, выделили и исследовали их стабильность, связывание с FcRn, а также ингибирование связывания IgG с FcRn.
Материалы и методы
Субклонирование Z вариантов в экспрессирующие векторы: ДНК двенадцати FcRn-связывающих Z вариантов (Z13577 (SEQ ID NO: 19), Z13578 (SEQ ID NO: 20), Z13583 (SEQ ID NO: 23), Z13592 (SEQ ID NO: 28), Z13616 (SEQ ID NO: 41), Z13621 (SEQ ID NO: 44), Z13654 (SEQ ID NO: 65), Z13663 (SEQ ID NO: 70), Z13669 (SEQ ID NO: 73), Z13674 (SEQ ID NO: 75), Z13675 (SEQ ID NO: 76) и Z13676 (SEQ ID NO: 77)) амплифицировали из вектора библиотеки pAY02592. Субклонирование осуществляли, как описано в Примере 3. Фрагменты Z генов субклонировали в экспрессирующий вектор pAY01448 что приводило к кодируемой последовательности 
Получение Z вариантов: Культивирование и очистку Z вариантов, несущих His6 метку, осуществляли по существу, как описано в Примере 3. Для достижения большей чистоты после IMAC очистки второй партии двенадцати зрелых вариантов и первичного Z варианта Z07918 добавили стадию обращенно-фазовой хроматографии (RPC). Образцы из этой партии использовали, где указано.
Анализ CD: Для определения температуры плавления (Tm) и оценки вторичной структуры Z вариантов (партия, очищенная посредством RPC), выполняли анализ CD, как описано в Примере 3.
Анализы связывания и кинетики посредством Biacore: Взаимодействие FcRn-связывающих Z вариантов, несущих His6-метку, с FcRn человека анализировали на приборе Biacore 2000 по существу, как описано в Примере 3. В качестве белка-мишени использовали FcRn человека (hFcRn) или FcRn яванского макака (cFcRn), приобретенные у Biorbyt (кат. номера orb84388 и orb99075, соответственно). В первой серии экспериментов 100 нМ Z вариантов инъецировали при pH 6,0 в течение 2 мин со скоростью 30 мкл/мин поверх иммобилизованного hFcRn с последующей диссоциацией в буферах с pH 6,0 или pH 7,4, используя процедуру соинъекции. Фаза диссоциации составляла 4 мин, а время уравновешивания между инъекциями аналитов составляло 30 мин.
Во второй серии экспериментов определяли приблизительные кинетические константы (kon и koff) и аффинности (KD) для подгруппы Z вариантов, которые инъецировали в концентрациях 540 нМ, 180 нМ, 60 нМ, 20 нМ и 6,7 нМ поверх иммобилизованного hFcRn. Как указано выше, аналиты инъецировали в течение 2 мин со скоростью 30 мкл/мин, фаза диссоциации составляла 4 мин, а время уравновешивания между инъекциями аналитов составляло 30 мин.
В третьей серии экспериментов выполняли кинетический анализ двенадцати зрелых Z вариантов и первичного Z варианта Z07918 (SEQ ID NO: 1) (партии, очищенные посредством RPC) с hFcRn и cFcRn при pH 6. Серию концентраций Z вариантов, несущих His6-метку (270, 90, 30 и 10 нМ), пропускали в течение 4 мин со скоростью 30 мкл/мин через различные проточные ячейки чипа СМ5 с иммобилизованными на их поверхности hFcRn и cFcRn. В качестве подвижного буфера и для разведения Z вариантов, несущих His6-метку, использовали 0,005% РСТ с pH 6,0. Перед началом следующего цикла проводили диссоциацию в подвижном буфере в течение 20 мин, затем поверхность регенерировали путем 3×30 секундных пульсовых введений 0,005% РСТ с pH 7,4 и уравновешиванием в течение десяти минут.
Анализ блокирования посредством AlphaLISA: Способность Z вариантов ингибировать связывание IgG с FcRn анализировали в анализе AlphaLISA с, как описано в Примере 3.
Результаты
Получение Z вариантов: Двенадцать Z вариантов, связывающих FcRn, сконструированных с N-концевой His6 меткой, получили в Е. coli. Анализ каждого полученного препарата белка с помощью SDS-PAGE показал, что они преимущественно содержали FcRn-связывающий Z вариант. Правильную идентичность и молекулярную массу каждого Z варианта, связывающего FcRn, подтверждали с помощью HPLC-MS анализа.
Анализ CD: Результаты определения температур плавления показаны в Таблице 11. При наложении спектров, полученных до и после нагревания до 90°C, у всех Z вариантов, связывающих FcRn, наблюдался обратимый фолдинг.
Анализ связывания и кинетики посредством Biacore: В первой серии экспериментов на приборе Biacore исследовали связывание двенадцати Z вариантов с FcRn человека и диссоциацию при различных значениях pH, последовательно инъецируя каждый из Z вариантов при pH 6,0 и буфер с pH 6,0 или pH 7,4 по поверхности чипа, содержаещй FcRn. Лиганд, FcRn человека, иммобилизовали на поверхности до уровня 890 RU. Двенадцать Z вариантов демонстрировали связывание с FcRn при pH 6,0, и у всех вариантов скорость диссоциации при pH 7,4 была быстрее, чем при pH 6,0.
Во второй серии экспериментов определяли кинетические константы у Z вариантов Z13577 (SEQ ID NO: 19) и Z13621 (SEQ ID NO: 44), взаимодействующих с FcRn при pH 6,0 (см. Таблицу 12). Кинетические константы рассчитывали с использованием кривых, полученных для двух или четырех инъецированных концентраций Z13577 и Z13621, соответственно.
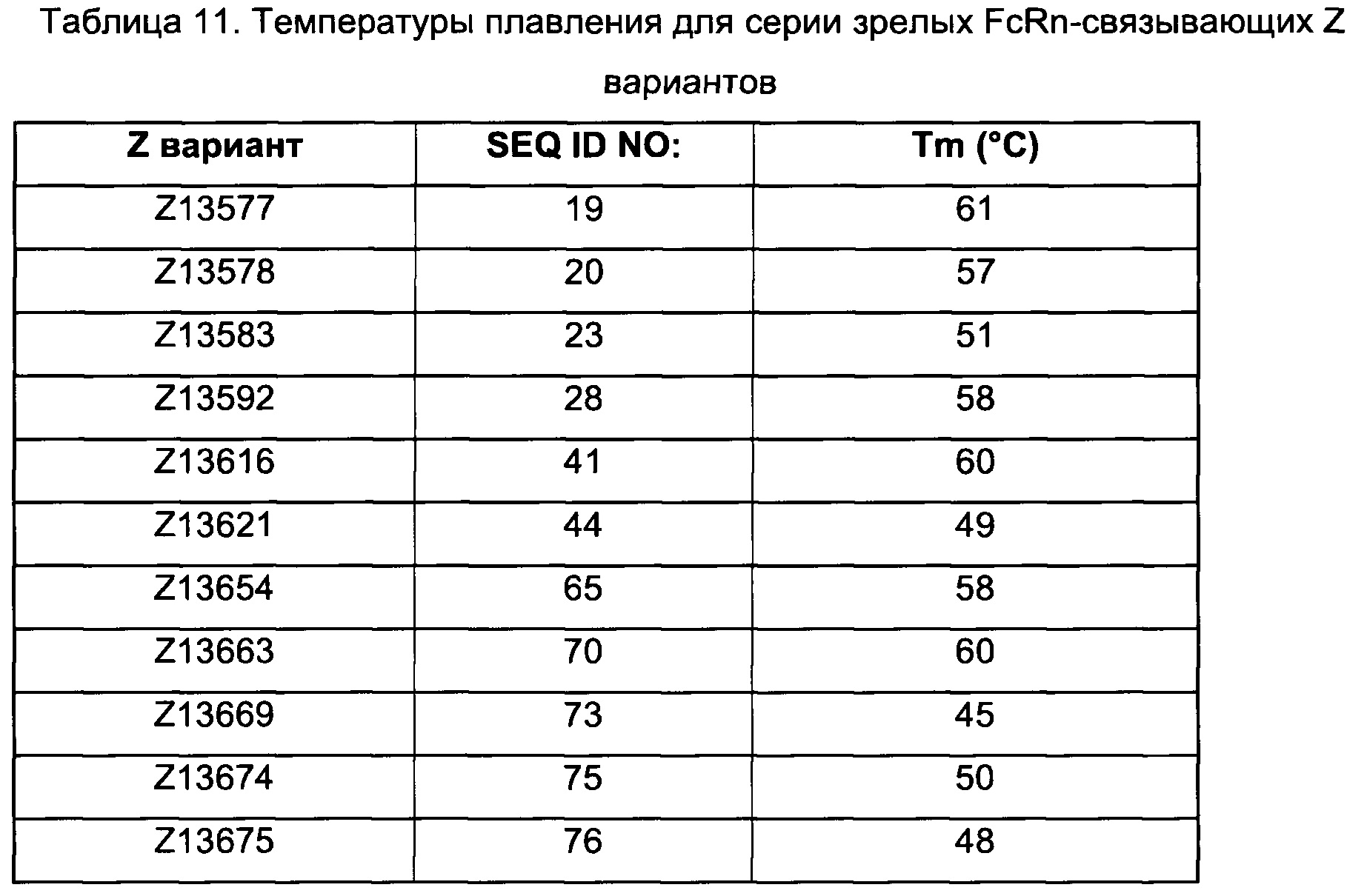
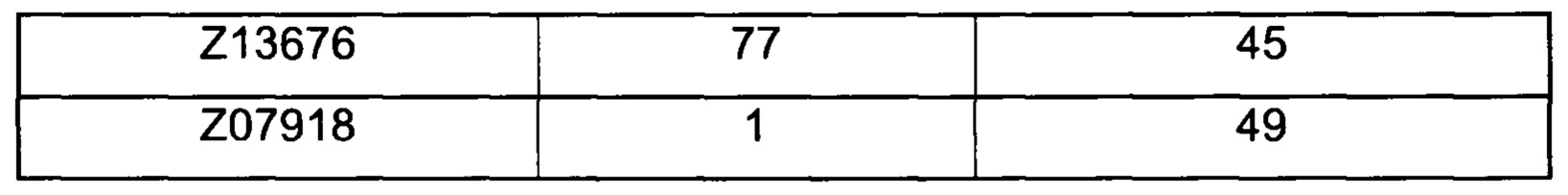

В третьей серии экспериментов определяли кинетические константы тринадцати Z вариантов, несущих His6-метку, взаимодействующих с FcRn человека или яванского макака при pH 6,0 (Таблица 13). FcRn иммобилизовали на поверхности чипа до уровня 1196 RU (FcRn человека) и 788 RU (FcRn яванского макака), соответственно. Для каждого Z варианта рассчитывали кинетические константы с использованием кривых, полученных для четырех инъецированных концентраций.
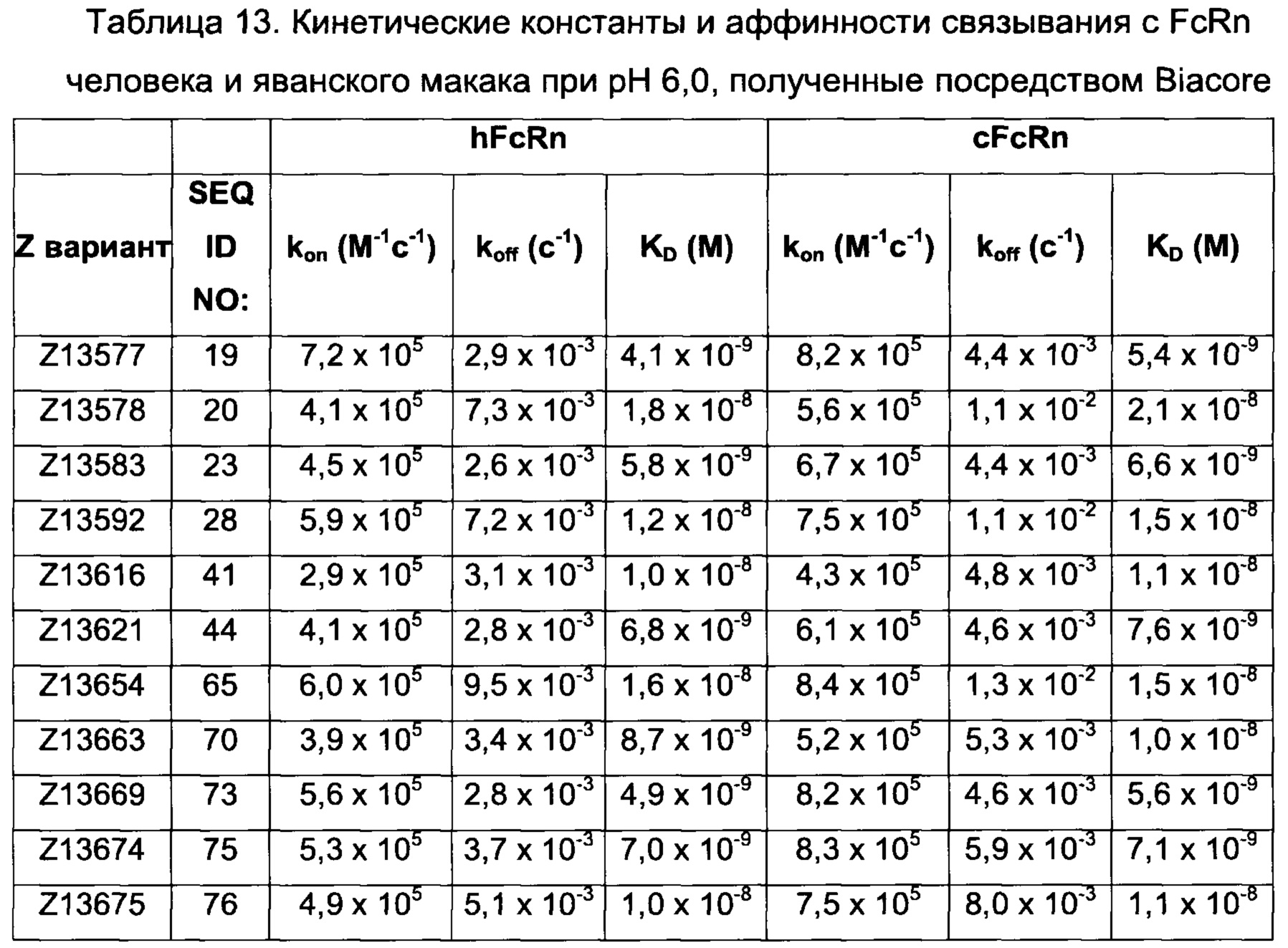

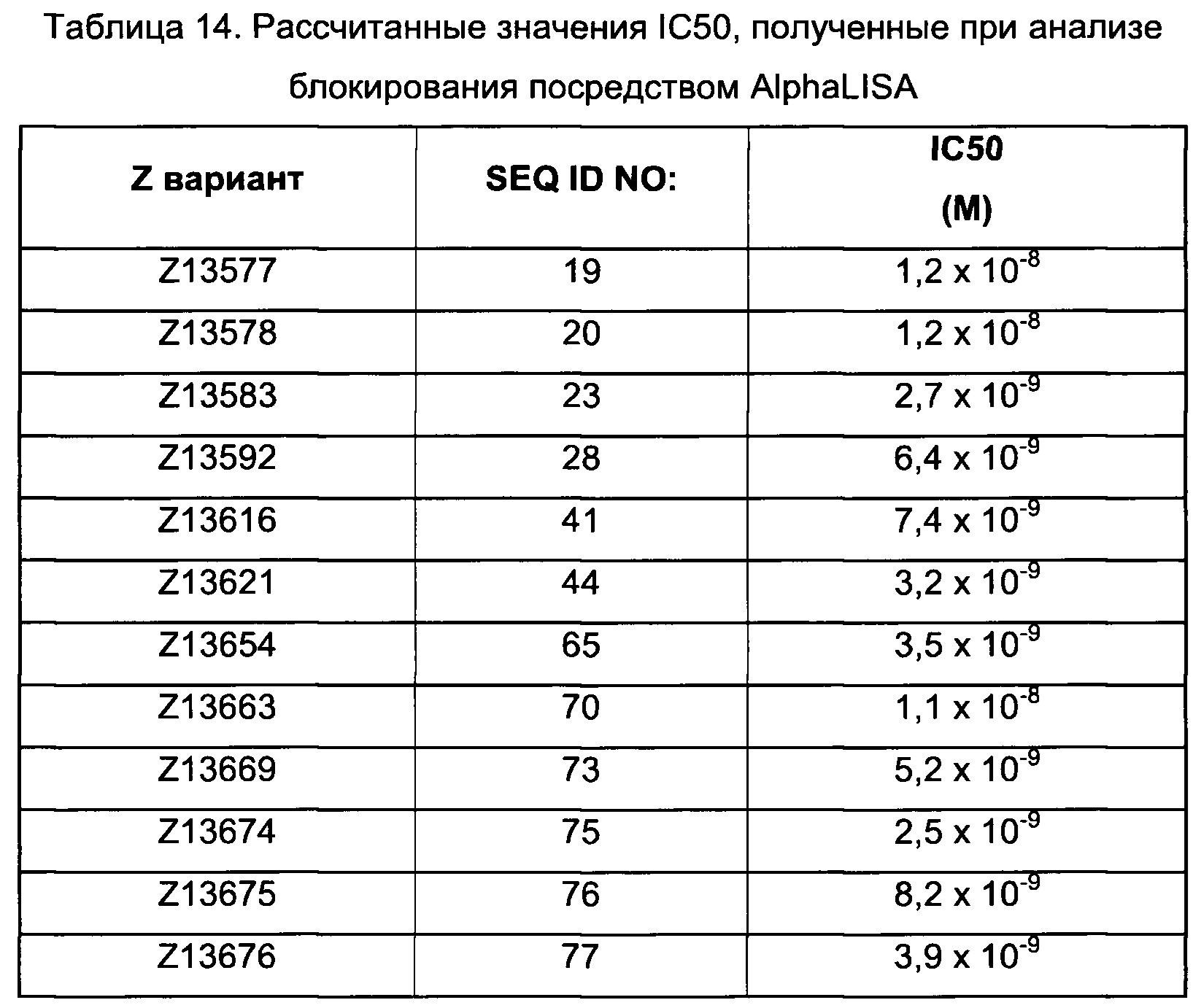
Анализ блокирования посредством AlphaLISA: Способность двенадцати зрелых Z вариантов, несущих His6-метку, ингибировать связывание IgG с FcRn тестировали с помощью анализа блокирования AlphaLISA. Z варианты в серии разведений инкубировали с биотинилированным FcRn человека и определяли блокирующую способность каждого соответствующего варианта после добавления покрытых IgG гранул-акцепторов и затем покрытых стрептавидином гранул-доноров. Ингибирование можно было измерить как снижение сигнала AlphaLISA в случае положительных Z вариантов. Показали, что все двенадцать исследованных Z вариантов блокировали связывание IgG с FcRn, рассчитанные значения IC50 приведены в Таблице 14.
Пример 10
Сравнение способности блокировать связывание IgG с FcRn
В данном примере способность Z варианта - His6-Z07918 (SEQ ID NO: 1) к блокированию связывания IgG с FcRn сравнивали с внутривенным иммуноглобулином (IVIg) и подкожным иммуноглобулином (SCIg), используемыми в настоящее время в лечении некоторых аутоиммунных расстройств.
Материалы и методы
Блокирование иммунофлуоресцентного окрашивания IgG-FcRn: Трансдуцированные человеческим или мышиным FcRn-eGFP клетки HeLa получали, как описано в Примере 4. Фиксированные клетки ресуспендировали в 50 мкл смеси 50 нМ конъюгированных с Alexa Fluor® 647 IgG человека (Jackson laboratories, кат. номер 009-600-003) и Z07918, несущего His6-метку, IVIg (Octagam®, Octapharma) или SCIg (Gammanorm®, Octapharma), соответственно, разведенных в концентрациях 1000, 100, 10, 1, 0,1 или 0 (контроль - буфер) нМ в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG (Life Technologies) и 0,1% сапонин (AppliChem). Клетки инкубировали в течение 1 ч при 37°C в темноте, отмывали 2×100 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG и ресуспендировали в 180 мкл буфера Мак-Илвейна, pH 6,0, содержащего 1% BSA. Получали данные для 10000 GFP/FcRn-положительных клеток с помощью FACS Calibur (Beckman Coulter) и анализировали данные с помощью программы Flowing 2.5.0 (Университет Турку).
Результаты
Эксперимент проводили для определения того, блокирует ли FcRn-связывающий Z вариант His6-Z07918 (SEQ ID NO: 1) взаимодействие IgG-FcRn, и для сравнения блокирующего эффекта с IVIg и SCIg. Трансдуцированные человеческим или мышиным FcRn-eGFP клетки HeLa инкубировали с IgG человека, конъюгированным с Alexa Fluor® 647. Связывание блокировали немеченными His6-Z07918, IVIg и SCIg в различных концентрациях. Результаты показали, что His6-Z07918 эффективно блокировал связывание hIgG с hFcRn в той же степени, что и IVIg и SCIg (Фиг. 9).
Пример 11
Усиление катаболизма IgG у мышей посредством FcRn-связывающих Z вариантов
Способность FcRn-связывающего Z варианта Z07918 блокировать связывание IgG с FcRn in vitro была продемонстрирована в Примере 10. В данном Примере блокирующую способность того же Z варианта оценивали in vivo. Блокирование взаимодействия IgG-FcRn in vivo приведет к усилению катаболизма IgG и последующему снижению уровней IgG (Mezo 2008, см. выше).
Материалы и методы
Исследование на животных: Самцам мышей NMRI (Charles River) вводили FcRn-связывающие Z варианты Z11948 (SEQ ID NO: 354) и Z07918-РР013 (Z07918 (SEQ ID NO: 1) идентичен Z11948, но имеет начальные N-концевые аминокислоты VD вместо АЕ, в слиянии с вариантом РР013 ABD (SEQ ID NO: 377)) в дозе 16,3 мкмоль/кг или носитель (буфер PBS). Мыши получали пять внутривенных инъекций через 0, 24, 48, 72 и 96 ч. Образцы сыворотки брали через 0, 72, 120 и 168 ч (окончание исследования) и хранили при -20°C. Концентрацию мышиных IgG в сыворотке количественно определяли с помощью ELISA.
ELISA мышиного IgG: Концентрацию мышиных IgG в образцах сыворотки мышей анализировали посредством набора ELISA для мышиного IgG (Mabtech 3825-1AD-6), как описано производителем. Концентрацию mIgG рассчитывали из прилагавшейся стандартной кривой и формулы нелинейной регрессии в программе GraphPad Prism 5. Концентрацию IgG у отдельных мышей через 24, 72, 120 и 168 ч соотносили с уровнем в момент времени 0 ч и, таким образом, представляли результаты в процентах от IgG (0 ч).
Результаты
Результаты продемонстрировали снижение концентрации мышиных IgG у мышей, получавших FcRn-специфические Z варианты. И Z11948, и слитый с ABD вариант Z07918-PP013 снижали концентрацию эндогенных IgG у мышей in vivo. Наиболее выраженные эффекты были получены для слитого с ABD варианта через 120 часов. Таким образом, результаты свидетельствуют, что FcRn-специфические Z варианты блокируют рециркуляцию IgG, что приводит к усиленному катаболизму IgG и последующему снижению уровней IgG у мышей.
Пример 12
Трансцитоз FcRn-связывающих Z вариантов in vitro
В данном Примере тестируют способность FcRn-связывающих Z вариантов транспортироваться через эпителиальные или эндотелиальные клетки или рециркулировать посредством FcRn in vitro. Лекарственное средство, содержащее вариант Z, со способностью к трансцитозу будет облегчать поглощение лекарственного средства, например, при пероральном или легочном введении.
Материалы и методы
Клетки, например Т84, MDCK, HeLa, СаСо2, CaLu-1 и/или CaLu-3, с или без эндогенной или рекомбинантной экспрессии FcRn, выращивают в соответствующей питательной среде на мембране в трансвеллах для образования монослоя. Целостность монослоя можно оценить путем измерения электрического сопротивления или добавления зонда, который не способен проникать или активно транспортироваться через клеточный монослой. Определенный монослой клеток метят с апикальной или базолатеральной стороны лигандом, таким как FcRn-связывающие Z варианты, HSA или IgG в буфере, таком как HBSS (сбалансированный солевой раствор Хенкса, SigmaAldrich, кат. номер Н9269) или ростовой среде при подходящем pH и температуре, и затем вытесняют его, добавляя в противоположный отсек буфер, такой как HBSS, или ростовую среду при подходящем pH и температуре.
В варианте данного исследования лиганды можно вытеснять добавлением буферов, таких как HBSS, или ростовой среды при подходящем pH и температуре, с той же стороны, что и введение, чтобы также измерить рециркуляцию лиганда. Это можно выполнять в трансвеллах или в культуральных чашках. Клетки высаживают в трансвеллы или культуральные чашки и добавляют к ним лиганды, такие как FcRn-связывающие Z варианты, HSA или IgG. Поглощенные путем эндоцитоза лиганды будут связываться с FcRn и возвращаться на клеточную поверхность с той же стороны, где их загрузили, или с противоположной стороны. После добавления свободные лиганды удаляют путем промывания клеток холодным буфером. Для вытеснения лигандов к клеткам добавляют теплый буфер или среду, а через промежуток времени в диапазоне от 10 минут до нескольких часов буфер или среду удаляют и подвергают анализу на предмет присутствия лигандов.
В варианте данного исследования такие лиганды, как FcRn-связывающие Z варианты, HSA или IgG, можно использовать для блокирования связывания FcRn с такими лигандами, как другие FcRn-связывающие Z варианты, HSA или IgG, путем введения их к клеткам одновременно или последовательно.
Количество лиганда можно количественно определить такими способами, как ELISA, HPLC-MS, флуоресцентное окрашивание или радиоактивное мечение.
Ожидается, что описанные выше результаты эксперимента демонстрируют, что FcRn-специфические Z варианты могут подвергаться трансцитозу и/или рециркуляции in vitro.
Пример 13
Связывание гомодимерных FcRn-связывающих полипептидов с клетками HeLa, трансфицированными человеческим FcRn/eGFP
В данном Примере исследовали связывающую способность гомодимерных FcRn-связывающих полипептидов и сравнивали со связывающей способностью мономерных первичных и зрелых Z вариантов. Клетки HeLa, экспрессирующие трансгенный ген человеческого FcRn-eGFP, получали, как описано в Примере 4, и описано применение указанных клеток в анализе посредством проточной цитометрии с Z вариантами, меченными Alexa Fluor® 647.
Материалы и методы
Мечение FcRn-связывающих полипептидов Alexa Fluor® 647: Два гомодимерных полипептида, несущие His6-метку, Z11948-(G4S)3-Z11948 (SEQ ID NO: 369) и Z11948-(G4S)-Z11948 (SEQ ID NO: 368), первичный мономерный Z вариант, несущий His6-метку Z07918 (SEQ ID NO: 1) и зрелые мономерные несущие His6-метку Z варианты Z13583 (SEQ ID NO: 23), Z13621 (SEQ ID NO: 44), Z13654 (SEQ ID NO: 65) и Z13674 (SEQ ID NO: 75) метили сукцинимидиловым эфиром карбоновой кислоты Alexa Fluor® 647 (Invitrogen кат. номер А20106). Перед присоединением метки pH суспензий образцов (в PBS pH 7,4) доводили до 8,3 путем добавления к 90 мкл суспензии образца 10 мкл 0,1 М натрий-бикарбонатного буфера, pH 8,3. К 100 мкл суспензии каждого образца добавляли 10 мкл красителя - сложного сукцинимидилового эфира Alexa Fluor® 647 (10 мг/мл в DMSO, что соответствовало 4х молярному избытку). Смеси инкубировали при комнатной температуре в темноте в течение 1 ч в покачивающем ротационном смесителе. Реакционные смеси немедленно переносили в кассеты для диализа (НОММ 3500) (Thermo Scientific, кат. номер 66333) и удаляли свободный краситель посредством диализа в PBS pH 7,4.
Иммунофлуоресцентное окрашивание трансфицированных человеческим hFcRn-eGFP клеток HeLa FcRn-связывающими полипептидами: клетки HeLa, несущие hFcRn-eGFP, собирали посредством трипсинизации и перед подсчетом дважды отмывали буфером Мак-Илвейна с pH 6,0. Переносили пипеткой по 100000 клеток на лунку 96-луночного планшета с v-образной формой дна лунок (Nunc, кат. номер 277143) и затем осаждали клетки в планшете при 1700 об/мин в течение 4 мин при 4°C. Супернатанты удаляли и фиксировали клетки 50 мкл 2% формальдегида (Sigma Aldrich, кат. номер F8775) в буфере Мак-Илвейна в течение 10 мин при комнатной температуре. Затем клетки промывали 2×100 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG (Life Technologies), и ресуспендировали в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG и 0,1% сапонина (AppliChem, кат. номер А4518.0100), с добавлением 640 нМ меченных Alexa Fluor® 647 полипептидов, несущих His6-метку, Z11948-(G4S)3-Z11948 и Z11948-(G4S)-Z11948 и Z07918. Трансдуцированные клетки HeLa, которые инкубировали только с буфером, использовали в качестве контроля. Клетки инкубировали в течение 1 ч при 8°C на шейкере в темноте. Затем клетки промывали в двух разных условиях; 2×150 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG, или 2×150 мкл PBS, pH 7,4, содержащего 2,5% FBS со сверхнизким содержанием IgG, и проводили 20-минутную стадию инкубации в PBS, pH 7,4, содержащем 2,5% FBS со сверхнизким содержанием IgG. После отмывки все образцы ресуспендировали в 180 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG. Получали данные для 10000 GFP/FcRn-положительных клеток с помощью FACS Calibur (Beckman Coulter) и анализировали данные с помощью программы Flowing 2.5.0 (Университет Турку).
Результаты
Для определения способности FcRn-связывающих димеров связываться с человеческим FcRn на клетках HeLa, тансдуцированных человеческим FcRn/eGFP, и сравнения их связывающей способности с мономерными FcRn-связывающими Z вариантами использовали метод проточной цитометрии. Анализ также проводили для определения того, влияет ли димерный формат на pH-зависимое отсоединение от белка FcRn. Эксперимент проводили при pH 6,0 с отмывками при pH 6,0 или pH 7,4 с димерами Z11948-(G4S)3-Z11948 и Z11948-(G4S)-Z11948 и мономерами Z07918, Z13583, Z13621, Z13654 и Z13674, меченными Alexa Fluor® 647. Последовательности Z11948 и Z07918 идентичны, за исключением первых двух аминокислотных остатков (АЕ или VD). Рассчитанные значения MFI представлены на Фиг. 11. Результаты показывают, что димерный формат увеличивает связывающую способность FcRn-связывающих полипептидов по сравнению с соответствующим мономером (Фиг. 11А) и что зрелые Z варианты (Z13583, Z13621, Z13654 и Z13674) обладают более высокой связывающей способностью по сравнению с первичным Z вариантом Z07918 (Фиг. 11Б). Данные демонстрируют, что pH-зависимое отсоединение от FcRn снижается при использовании димерного формата, что позволяет предположить, что FcRn-связывающие димеры могут иметь улучшенный профиль pH-зависимого связывания по сравнению с соответствующими мономерными вариантами.
Пример 14
Сравнение способности блокировать связывание IgG с FcRn
В данном примере способность FcRn-связывающих димеров Z11948-(G4S)3-Z11948 и Z11948-(G4S)-Z11948 к блокированию связывания IgG с FcRn сравнивали с таковой у мономерных FcRn-связывающих Z вариантов, а также внутривенного иммуноглобулина (IVIg) и подкожного иммуноглобулина (SCIg), которые в настоящее время применяют в лечении некоторых аутоиммунных расстройств.
Материалы и методы
Блокирование иммунофлуоресцентного окрашивания IgG-FcRn: Трансдуцированные человеческим FcRn-eGFP клетки HeLa получали, как описано в Примере 4. Фиксированные клетки ресуспендировали в 50 мкл смеси 50 нМ IgG человека (Jackson laboratories, кат. номер 009-600-003), конъюгированных с Alexa Fluor® 647, и несущих His6-метку Z11948-(G4S)3-Z11948 (SEQ ID NO: 369), Z11948-(G4S)-Z11948 (SEQ ID NO: 368), Z07918 (SEQ ID NO: 1), Z13583 (SEQ ID NO: 23), Z13621 (SEQ ID NO: 44); IVIg (Octagam®, Octapharma) или SCIg (Gammanorm®, Octapharma), соответственно, разведенных до концентраций 1000, 100, 10, 1, 0,1 или 0 (контроль - буфер) нМ в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG (Life Technologies) и 0,1% сапонин (AppliChem). Клетки инкубировали в течение 1 ч при 37°C в темноте, отмывали 2×100 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG, и ресуспендировали в 180 мкл буфера Мак-Илвейна, pH 6,0, содержащем 1% BSA. Получали данные для 10000 GFP/FcRn-положительных клеток с помощью FACS Calibur (Beckman Coulter) и анализировали данные с помощью программы Flowing 2.5.0 (Университет Турку).
Результаты
Эксперимент проводили для определения того, блокируют ли FcRn-связывающие димеры Z11948-(G4S)3-Z11948 и Z11948-(G4S)-Z11948 взаимодействие IgG-FcRn, и для сравнения блокирующего эффекта с таковым мономерных FcRn-связывающих Z вариантов Z07918, Z13583 и Z13621, а также IVIg и SCIg. Трансдуцированные человеческим FcRn-eGFP клетки HeLa инкубировали с IgG человека, конъюгированными с Alexa Fluor® 647. Связывание блокировали немеченными Z-вариантами, IVIg и SCIg в различных концентрациях. Результаты показывают, что FcRn-связывающие димеры обладают улучшенным блокирующим эффектом в отношении связывания hIgG с hFcRn по сравнению с мономерным Z вариантом Z07918, IVIg и SCIg (Фиг. 12А). Кроме того, блокирующая способность зрелых мономерных Z вариантов Z13583 и Z13621 улучшалась по сравнению с блокирующей способностью первичного мономерного Z варианта Z07918 (Фиг. 12Б). Рассчитанные значения IC50, полученные при анализе блокирования, представлены в Таблице 15.

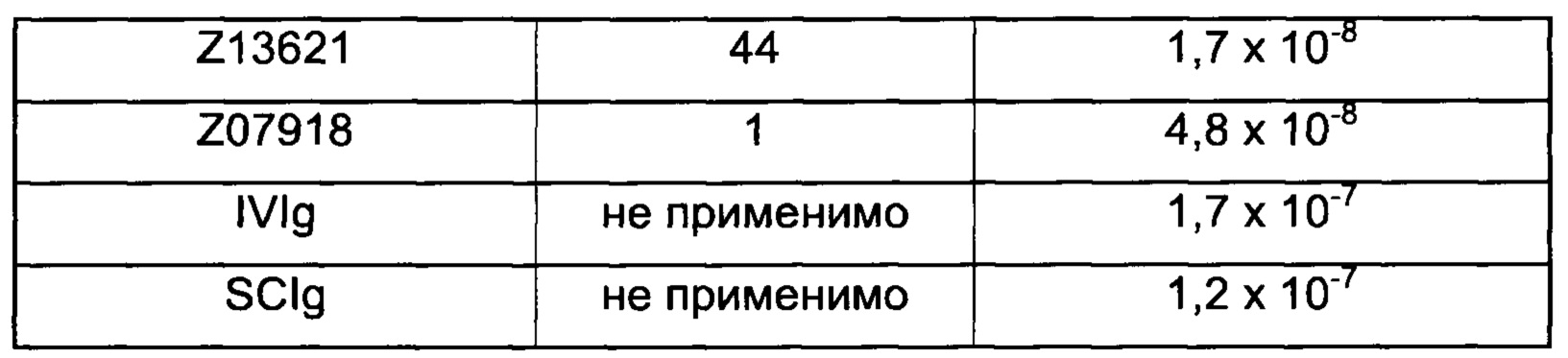
Пример 15
Получение димерных FcRn-связывающих полипептидов
Материалы и методы
Z варианты Z17303 (SEQ ID NO: 357), Z18632 (SEQ ID NO: 365), Z18633 (SEQ ID NO: 366) и Z18634 (SEQ ID NO: 367) конструировали в виде димеров, в слиянии с альбумин-связывающим вариантом РР013 (SEQ ID NO: 377) в общем формате [Z#####]-ASGS-PP013-GT-(G4S)-[Z#####]. Полученные полипептиды обозначали как ZAZ3824 (SEQ ID NO: 373), ZAZ3869 (SEQ ID NO: 374), ZAZ3870 (SEQ ID NO: 375) и ZAZ3871 (SEQ ID NO: 376), соответственно. Димерные полипептиды Z варианта Z17303 в слиянии с РР013 также конструировали с разными линкерами или C-концевым слиянием РР013, что приводило к полипептидам Z17303-GAP(G4S)3TS-PP013-GT(G4S)3PR-Z17303 и Z17303-GAP(G4S)3TS-Z17303-GT(G4S)3PR-PP013, обозначенных как ZAZ3715 (SEQ ID NO: 371) и ZZA3716 (SEQ ID NO: 372), соответственно. Кроме того, Z17303 сконструировали в виде димера без РР013, но с N-концевой His6-меткой, что приводило к полипептиду GSS-His6-LQ-Z17303-GT(G4S)3-Z17303, обозначенного как ZZ3556 (SEQ ID NO: 370).
Культивирование осуществляли, как описано в Примере 3. Очистку полипептидов, содержащих РР013, осуществляли с использованием анти-ABD аффинной хроматографии и RPC, как описано в Примере 6, тогда как очистку ZZ3556 с His6-меткой осуществляли с помощью IMAC, как описано в Примере 3.
Результаты
Семь FcRn-связывающих димерных полипептидов, сконструированных либо с His6-меткой, либо с группировкой ABD, получали в Е. coli. Количество белка, очищенного посредством аффинной хроматографии, которое определяли спектрофотометрически путем измерения поглощения при 280 нм, варьировало приблизительно от 2 до 18 мг на г бактериального осадка. Анализ каждого полученного препарата белка с помощью SDS-PAGE показал, что они преимущественно содержали FcRn-связывающий полипептид. Правильную идентичность и молекулярную массу каждого FcRn-связывающего полипептида подтверждали посредством HPLC-MS анализа.
Пример 16
Связывание гомо- и/или гетеродимерных FcRn-связывающих полипептидов с клетками HeLa, трансфицированными человеческим FcRn/eGFP
В данном Примере исследуют связывающую способность гомо- и гетеродимерных FcRn-связывающих полипептидов, содержащих зрелые Z варианты. Клетки HeLa, экспрессирующие трансгенный ген человеческого FcRn-eGFP, полученные, как описано в Примере 4, используют в анализе посредством проточной цитометрии с Z вариантами, меченными Alexa Fluor® 647.
Материалы и методы
Мечение Z вариантов, связывающих FcRn, посредством Alexa Fluor® 647 Гомо- и/или гетеродимерные FcRn-связывающие полипептиды метят сложным сукцинимидиловым эфиром карбоновой кислоты Alexa Fluor® 647 (Invitrogen, кат. номер А20106), как описано в Примере 13.
Иммунофлуоресцентное окрашивание клеток HeLa, трансфицированных FcRn-eGFP, FcRn-связывающими полипептидами: клетки HeLa, несущие hFcRn-eGFP, собирают посредством трипсинизации и перед подсчетом дважды отмывают буфером Мак-Илвейна с pH 6,0. Переносят пипеткой по 100000 клеток на лунку 96-луночного планшета с v-образной формой дна лунок (Nunc, кат. номер 277143) и затем осаждают клетки в планшете при 1700 об/мин в течение 4 мин при 4°C. Супернатанты удаляют и фиксируют клетки 50 мкл 2% формальдегида (Sigma Aldrich, кат. номер F8775) в буфере Мак-Илвейна в течение 10 мин при комнатной температуре. Затем клетки промывают 2×100 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG (Life Technologies), и ресуспендируют в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG и 0,1% сапонина (AppliChem, кат. номер А4518.0100), с добавлением 640 нМ меченных Alexa Fluor® 647 гомо- и/или гетеродимерных FcRn-связывающих полипептидов, несущих His6-метку, и соответствующего мономерного Z варианта.
Примеры форматов гомо- и/или гетеродимеров включают Z#####-(G4S)3-Z##### и Z#####-(G4S)-Z#####, где Z####, например, выбран из Z13583 (SEQ ID NO: 23), Z13621 (SEQ ID NO: 44), Z13654 (SEQ ID NO: 65) или Z13674 (SEQ ID NO: 75), или тех же Z вариантов, начинающихся аминокислотными остатками АЕ вместо VD, например как в Z17303 (SEQ ID NO: 357), который идентичен Z13621 (SEQ ID NO: 44) за исключением N-концевых АЕ. Клонирование можно возможно осуществлять с C-концевой His6-меткой, как описано в Примере 3 или с N-концевой His6-меткой, как в SEQ ID NO: 362.
Трансдуцированные клетки HeLa, инкубированные только с буфером, используют в качестве контроля. Клетки инкубируют в течение 1 ч при 8°C на шейкере в темноте. Затем клетки промывают в двух разных условиях; 2×150 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG, или 2×150 мкл PBS, pH 7,4, содержащего 2,5% FBS со сверхнизким содержанием IgG, и проводили 20-минутную стадию инкубации в PBS, pH 7,4, содержащем 2,5% FBS со сверхнизким содержанием IgG. После отмывки все образцы ресуспендируют в 180 мкл буфера Мак-Илвейна, pH 6,0, содержащего 2,5% FBS со сверхнизким содержанием IgG. Получают данные для 10000 GFP/FcRn-положительных клеток с помощью FACS Calibur (Beckman Coulter) и анализируют данные с помощью программы Flowing 2.5.0 (Университет Турку).
Результаты
Для определения способности гомо- и/или гетеродимерных FcRn-связывающих полипептидов, содержащих зрелые Z варианты, связываться с человеческим FcRn на клетках HeLa, трансдуцированных человеческим FcRn/eGFP, и сравнения их связывающей способности с мономерными FcRn-связывающими Z вариантами или димерными вариантами, содержащими первичные Z варианты, используют метод проточной цитометрии. Анализ также проводят для определения того, влияет ли димерный формат на pH-зависимое отсоединение от белка FcRn. Эксперименты проводят при pH 6,0 с отмывками при pH 6,0 или pH 7,4 с FcRn-связывающими Z вариантами, меченными Alexa Fluor® 647. Ожидается, что результаты эксперимента демонстрируют, что гомо- и/или гетеродимерный формат, так же как и включение зрелых Z вариантов с улучшенной аффинностью к FcRn, повышают связывающую способность FcRn-связывающих полипептидов, и что pH-зависимое отсоединение указанных полипептидов от FcRn снижается.
Пример 17
pH-зависимое связывание димерных полипептидов с FcRn человека
В данном Примере способность димерных полипептидов связываться с FcRn при различных значениях pH исследовали посредством ELISA и сравнивали со связывающей способностью мономерного Z варианта.
Материалы и методы
Способность димерных полипептидов ZAZ3824 (SEQ ID NO: 373), ZAZ3869 (SEQ ID NO: 374), ZAZ3870 (SEQ ID NO: 375) и ZAZ3871 (SEQ ID NO: 376), а также мономерного Z варианта Z13621 (SEQ ID NO: 44) связываться с FcRn человека при различных значениях pH исследовали посредством ELISA, где все этапы связывания и отмывания проводили либо при pH 6,0, либо при pH 7,4. Половину площади 96-луночных планшетов для ELISA покрывали 4 мкг/мл hFcRn (Biorbyt, кат. номер orb84388), разведенным в PBS при 4°C в течение ночи. Планшеты дважды промывали водопроводной водой и блокировали лунки 100 мкл PBSC (PBS, pH 7,4, с добавлением 1% казеина) в течение 1,5 ч при комнатной температуре. Блокирующий раствор сливали и лунки, на которые воздействовали pH 6,0, промывали однократно фосфатно-цитратным буфером Мак-Илвейна с pH 6,0. Различные FcRn-связывающие полипептиды добавляли в концентрации 100 нМ и ступенчато разводили разводили 1:10 до 0,1 пМ либо в РСС (фосфатно-цитратном буфере Мак-Илвейна, pH 6,0, с добавлением 1% казеина), либо в PBSC. В каждую лунку переносили по 50 мкл разведения и инкубировали планшеты для ELISA в течение 1,5 ч при комнатной температуре. Планшеты промывали четыре раза либо РСТ (фосфатно-цитратным буфером Мак-Илвейна, pH 6,0, с добавлением 0,05% Tween-20), либо PBST (PBS, pH 7,4, с добавлением 0,05% Tween-20). Связанные полипептиды выявляли с помощью 50 мкл/лунку Z-специфического мышиного антитела (собственного производства), разведенного до 2 мкг/мл в РСС или PBSC. Затем планшеты инкубировали в течение 1,5 ч при комнатной температуре с последующими отмывками, как описано выше. Добавляли конъюгированное с HRP козье антитело к Ig мыши, приобретенное от DAKO (Р0447), разведенное 1:5000 в РСС или PBSC, и инкубировали планшеты при комнатной температуре в течение 1 ч. После отмывки (как описано выше) в каждую лунку добавляли 50 мкл субстрата ImmunoPure ТМВ и проявляли планшеты согласно рекомендациям производителя. После развития окраски в течение 30 минут измеряли поглощение при 450 нм с помощью считывающего устройства для многолуночных планшетов (Victor3) и рассчитывали значения ЕС50 с использованием GraphPad Prism 5.
Результаты
Анализ проводили для сравнения связывающего потенциала мономерного и димерного формата при различных pH, а также для определения того, влияют ли внедренные в каркас мутации (Y5F, N52S и D53E; см. далее Примеры 24 и 25) на pH-зависимое связывание с FcRn. Эксперимент проводили в формате ELISA при pH 6,0 или pH 7,4. Результаты демонстрировали, что димерный формат превосходил мономерный формат в связывании FcRn независимо от pH (улучшение до 10х и 85х при pH 6,0 и pH 7,4, соответственно). Наиболее сильное связывание, как при pH 6,0, так и при pH 7,4 наблюдалось у димерного полипептида ZAZ3824, который также демонстрировал аналогичную связывающую способность (значение ЕС50) при pH 6,0 и pH 7,4. Кривые титрования, полученные в ELISA, показаны на Фиг. 13, а рассчитанные значения ЕС50 представлены в Таблице 16.
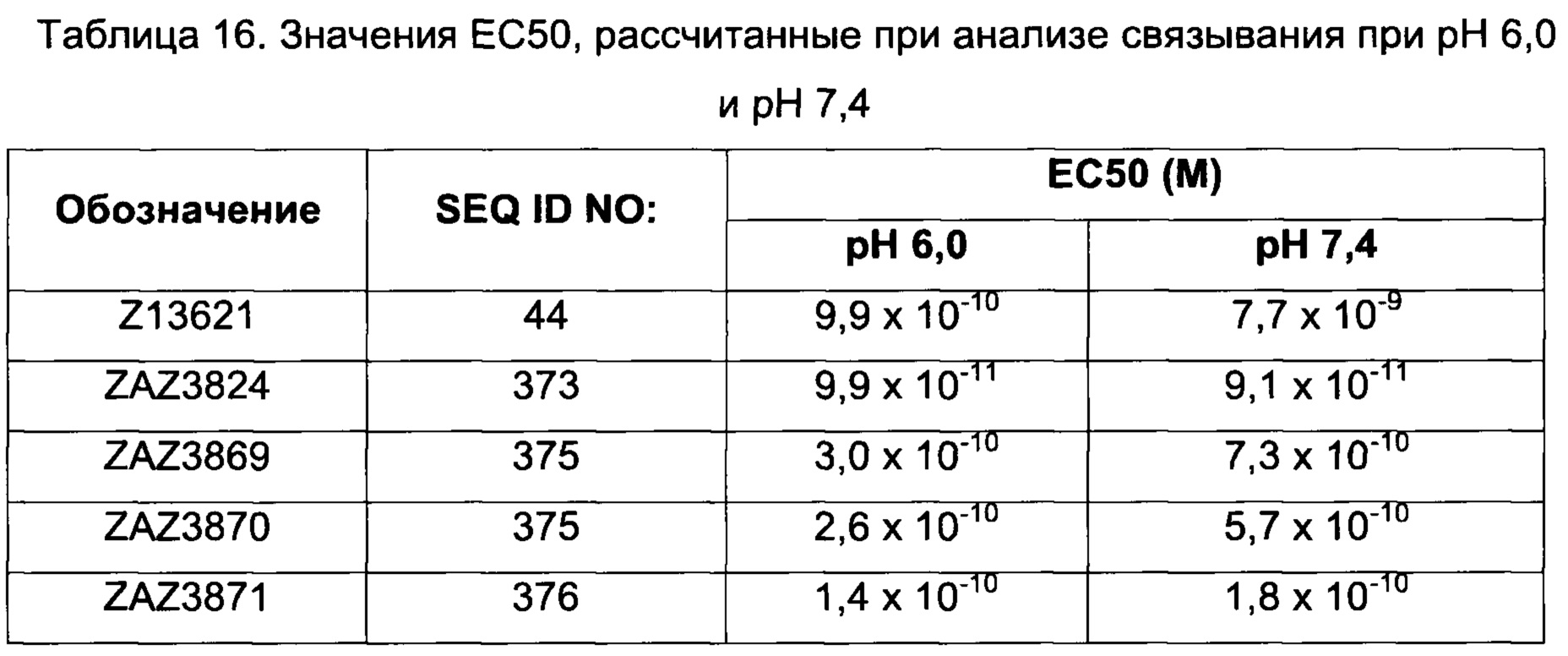
Пример 18
Сравнение способности блокировать связывание IgG с FcRn
В данном Примере способность полипептидов ингибировать связывание IgG с FcRn анализировали in vitro двумя различными способами: AlphaLISA и в анализе на клетках.
Материалы и методы
Анализ блокирования посредством AlphaLISA: Способность димерных FcRn-связывающих полипептидов ZZ3556 (SEQ ID NO: 370), ZAZ3715 (SEQ ID NO: 371) и ZZA3716 (SEQ ID NO: 372), а также мономерного Z13621 (SEQ ID NO: 44) блокировать взаимодействие IgG-FcRn анализировали посредством AlphaLISA. IgG человека (Roactemra) иммобилизовали на гранулах-акцепторах AlphaLISA (Perkin Elmer, кат. номер 6772002) согласно рекомендациям производителя. Человеческий FcRn (Biorbyt, кат. номер orb84388) биотинилировали по существу, как описано в Примере 2. Полипептиды серийно разводили 1:3 в буфере AlphaLISA (Perkin Elmer, кат. номер AL000F) pH 6,0 (доведен с помощью HCl) до конечных концентраций от 250 нМ до 13 пМ в 384-луночном планшете (Perkin Elmer, кат. номер G6005350) и инкубировали в течение 45 мин с 10 нМ биотинилированного hFcRn. Покрытые IgG гранулы-акцепторы добавляли в конечной концентрации 10 мкг/мл и инкубировали в течение 45 мин. В конце добавляли покрытые стрептавидином гранулы-доноры (Perkin Elmer, кат. номер 6760002) до конечной концентрации 40 мкг/мл и инкубировали в течение 30 мин. Все инкубации проводили при комнатной температуре в темноте. Планшеты анализировали с использованием мультипланшетного считывающего устройства EnSpire 2300 (Perkin Elmer), а значения IC50 рассчитывали с помощью GraphPad Prism 5.
Анализ блокирования IgG-FcRn с использованием клеток HeLa: Трансдуцированные FcRn-eGFP клетки HeLa получали, как описано в Примере 4. Каждый из полипептидов ZZ3556 (SEQ ID NO: 370), ZAZ3715 (SEQ ID NO: 371), ZZA3716 (SEQ ID NO: 372), ZAZ3824 (SEQ ID NO: 373), ZAZ3869 (SEQ ID NO: 374), ZAZ3870 (SEQ ID NO: 375), ZAZ3871 (SEQ ID NO: 376), Z13621 (SEQ ID NO: 44), Z18632 (SEQ ID NO: 365), Z18633 (SEQ ID NO: 366) и Z18634 (SEQ ID NO: 367), а также IVIg (Octagam®, Octapharma) или SCIg (Gammanorm®, Octapharma) разводили до концентраций 1000, 100, 10, 1, 0,1 или 0 (контроль - буфер) нМ в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG (Life Technologies) и 0,1% сапонин (AppliChem) и 50 нМ IgG человека, конъюгированных с Alexa Fluor® 647 (Jackson laboratories, кат. номер 009-600-003). Фиксированные клетки ресуспендировали в 100 мкл смеси и инкубировали в течение 1 ч при 37°C в темноте. Клетки отмывали и ресуспендировали в буфере Мак-Илвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG. Получали данные для 10000 GFP/FcRn-положительных клеток с помощью FACS Calibur (Beckman Coulter) и анализировали данные с помощью программы Flowing 2.5.0 (Университет Турку), а значения IC50 рассчитывали с помощью GraphPad Prism 5.
Результаты
AlphaLISA: Способность одного мономерного и трех димерных полипептидов ингибировать связывание IgG с FcRn тестировали в анализе блокирования AlphaLISA. Результаты показывают, что димерные полипептиды обладали лучшей блокирующей IgG способностью по сравнению с мономерным форматом. Два слитых с ABD димерных полипептида, ZAZ3715 и ZZA3716, имели значения IC50, очень близкие к таковым для ZZ3556, которое не содержит группировки ABD. Рассчитанные значения IC50, полученные при анализе блокирования методом AlphaLISA, представлены в Таблице 17.
Анализ блокирования IgG с использованием клеток HeLa: Трансдуцированные человеческим FcRn-eGFP клетки HeLa инкубировали с IgG человека, конъюгированными с Alexa Fluor® 647, и отобранными полипептидами для оценки способности полипептидов блокировать взаимодействие IgG-FcRn. В тест также включали внутривенный иммуноглобулин (IVIg) и подкожный иммуноглобулин (SCIg), которые в настоящее время применяют в лечении некоторых аутоиммунных расстройств. Эксперимент проводили при pH 6,0. Результаты показали, что димерный формат придавал лучшую блокирующую IgG способность по сравнению с мономерным форматом. ZAZ3715 (более длинные линкеры) сравнивали с ZAZ3824 (более короткие линкеры), и две конструкции показали схожие значения IC50. Между значениями IC50, полученными для ZAZ3715 и ZZA3716, то есть димерными конструкциями с ABD в разных положениях, различий не наблюдалось. Все исследованные полипептиды демонстрировали блокирующий IgG эффект, превосходящий таковой для IVIg и SCIg. Рассчитанные значения IC50, полученные при анализе блокирования с использованием клеток, представлены в Таблице 18.


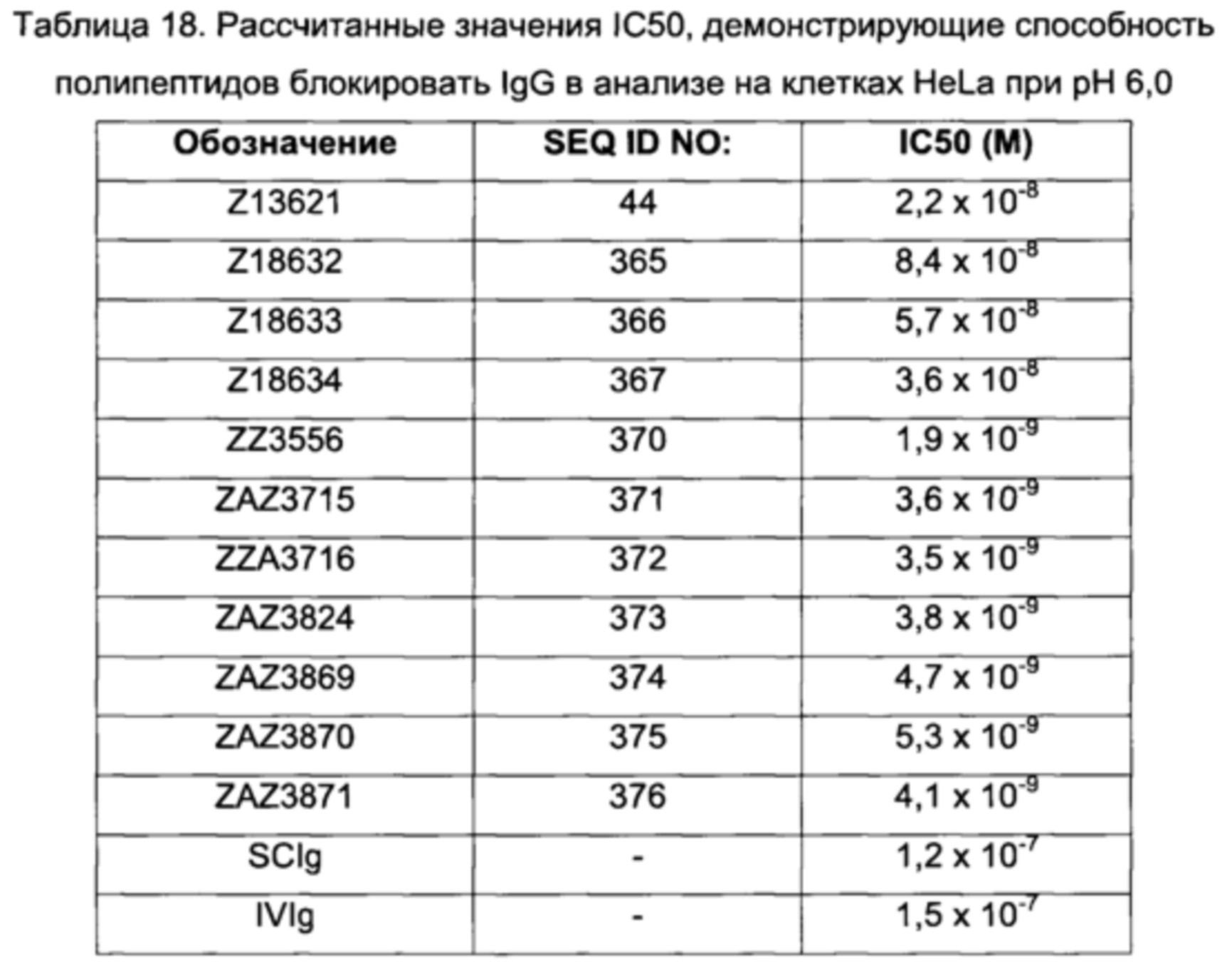
Пример 19
Сравнение способности блокировать рециркуляцию IgG
В данном Примере исследуют эффект гомо- и гетеродимерных FcRn-связывающих полипептидов на рециркуляцию IgG в клетках MDCK.2, трансдуцированных FcRn.
Материалы и методы
Лентивирусная трансдукция клеток MDCK.2: Осуществляют сотрансфекцию клеток HEK293T парами векторов 2k7neo-CMV-hB2M и pHR-cPPT-CMV-hFcRn-eGFP вместе с оболочкой VSV-G и пакующей плазмидой gag/pol, с использованием трансфекции с хлоридом кальция (Zufferey ef al., см. выше; Jakobsson et al. (2006, см. выше). Супернатанты культуры HEK293T, содержащие сформированные лентивирусные частицы лентивирусов с трансгенами FcRn и В2М, соответственно, используют для последовательной трансдукции клеток клеток MDCK.2 (АТСС, кат. номер CRL-2936) при малом числе пассажей. Полученные стабильно трансдуцированные клеточные линии MDCK.2 обозначают как hFcRn-eGFP (трансдуцированы генами человеческих FcRn-eGFP и hB2M).
Способность блокировать рециркуляцию IgG: Клетки MDCK.2, трансдуцированные человеческим FcRn-eGFP, высаживают в 96-луночные планшеты по 25000 клеток/лунку и инкубировали в течение ночи при 37°C, 5% CO2. Клетки инкубируют с гомо- и/или гетеродимерными FcRn-связывающими полипептидами в концентрациях, варьирующих от 200 до 0,01 нМ в буфере МакИлвейна, pH 6,0, содержащем 2,5% FBS со сверхнизким содержанием IgG, перед добавлением 500 нг/мл IgG человека, конъюгированных с Alexa Fluor® 647 (Jackson laboratories, кат. номер 009-600-003), и инкубируют дополнительно один час. Клетки промывают буфером Мак-Илвейна, pH 6,0, содержащим 2,5% FBS со сверхнизким содержанием IgG, и затем инкубируют в PBS, pH 7,4, содержащем 2,5% FBS со сверхнизким содержанием IgG в течение 2 ч при 37°C, 5% СО2. Затем супернатанты анализируют на предмет присутствия IgG человека, конъюгированных с Alexa Fluor® 647, с использованием мультипланшетного считывающего устройства EnSpire (Perkin Elmer). Для определения значений IC50 анализируют кривые ингибирования с помощью нелинейной регрессии с использованием программы GraphPad Prism 5.
Результаты
Ожидается, что результаты данного эксперимента демонстрируют дозозависимое снижение рециркуляции IgG под воздействием димерных FcRn-связывающих полипептидов.
Пример 20
Усиление катаболизма IgG под влиянием FcRn-связывающих полипептидов у мышей, трансгенных по FcRn
В данном Примере исследовали эффект гомодимерных FcRn-связывающих полипептидов на катаболизм IgG у мышей, трансгенных по FcRn человека, в двух различных вариантах.
Материалы и методы
Исследования на животных: В первом исследовании гомодимерный FcRn-связывающий полипептид ZAZ3715 (SEQ ID NO: 371) или ZZA3716 (SEQ ID NO: 372), или носитель (буфер PBS), вводили в момент времени 0 часов путем в/в инъекции самцам мышей B6.Cg-Fcgrttm1Dcr Tg(FCGRT)32Dcr/DcrJ (Jackson Laboratory, номенклатурный номер 14565) в дозе 6 мг/кг. За 24 часа до введения полипептида вводили 500 мг/кг hIgG (Kiovig, Baxter) путем в/в инъекции. Образцы сыворотки брали в -168, 0, 24, 72, 120 и 144 ч (окончание исследования) и хранили при -20°C.
Во втором исследовании гомодимерный FcRn-связывающий полипептид ZAZ3869 (SEQ ID NO: 374), ZAZ3870 (SEQ ID NO: 375) или ZAZ3871 (SEQ ID NO: 376), или носитель (буфер PBS), вводили в момент времени 0 часов путем в/в инъекции самцам мышей B6.Cg-Fcgrttm1Dcr Tg(FCGRT)32Dcr/DcrJ (Jackson Laboratory, номенклатурный номер 14565) в дозе 6 мг/кг. За 24 часа до введения полипептида вводили 500 мг/кг hIgG (Kiovig, Baxter) путем в/в инъекции. Образцы сыворотки брали в -168, 0, 24, 72, 120 и 144 ч (окончание исследования) и хранили при -20°C.
ELISA IgG человека: Концентрацию IgG человека в образцах сыворотки мышей, собранных в двух исследованиях, анализировали с помощью набора AlphaLISA для IgG человека (Perkin Elmer, кат. номер AL205C), как описано производителем. Концентрацию hIgG рассчитывали из прилагавшейся стандартной кривой и формулы нелинейной регрессии в программе GraphPad Prism 5.
Результаты
Результаты данных экспериментов демонстрируют снижение концентраций IgG человека с течением времени под воздействием FcRn-связывающих полипептидов. Снижение уровней IgG в двух группах, получавших полипептиды ZAZ3715 и ZZA3716, различалось незначительно или не различалось (Фиг. 14А). Данные результаты указывают, что FcRn-связывающие полипептиды, в которых ABD размещен в центре или на C-конце, одинаково эффективны в усилении катаболизма IgG. Полипептиды ZAZ3869, ZAZ3870 и ZAZ3871, соответственно, снижали уровни hIgG в одинаковой степени (Фиг. 14Б). Результаты второго эксперимента находились в том же диапазоне, что и результаты, полученные в первом эксперименте.
Таким образом, результаты двух экспериментов указывают, что FcRn-специфические полипептиды блокировали рециркуляцию IgG, что приводит к усиленному катаболизму IgG и последующему снижению уровней IgG у мышей, трансгенных по FcRn человека.
Пример 21
Усиление катаболизма IgG посредством димерных FcRn-связывающих полипептидов у мышей NMRI
В данном Примере эффект гомодимерных FcRn-связывающих полипептидов на катаболизм IgG исследовали на мышах NMRI.
Материалы и методы
Исследование на животных: Гомодимерный FcRn-связывающий полипептид ZAZ3715 (SEQ ID NO: 371) или ZZA3824 (SEQ ID NO: 372), или носитель (буфер PBS), вводили в момент времени 0 ч путем в/в инъекции самкам мышей NMRI в дозе 0,6 или 1,7 мкмоль/кг. Образцы сыворотки брали в 0, 24, 48 и 72 ч (окончание исследования) и хранили при -20°C.
ELISA IgG мыши: Концентрацию мышиных IgG в образцах сыворотки мышей анализировали с использованием набора ELISA для IgG мыши (Mabtech, кат. номер 3825-1AD-6), как описано производителем. Концентрацию mIgG рассчитывали с использованием стандартной кривой и формулы нелинейной регрессии в программе GraphPad Prism 5. Концентрацию IgG у отдельных мышей через 24, 48 и 72 ч соотносили с уровнем в момент времени 0 ч и, таким образом, представляли результаты в процентах от IgG (0 ч).
ELISA для исследования фармакокинетики (РК): Концентрации FcRn-связывающих полипептидов в образцах сыворотки мышей определяли посредством ELISA. В данном исследовании половинну площади 96-луночных планшетов покрывали мышиным поликлональным антителом против Z (собственного производства) в концентрации 4 мкг/мл в PBS (50 мкл/лунку) и инкубировали в течение ночи при 4°C. Затем планшеты двукратно промывали водопроводной водой и блокировали блокирующим буфером в течение 1 ч. Стандарт Z варианта собственного производства раститровывали в серии 3-кратных разведений (0,003-300 нг/мл), добавляли разведенные образцы сыворотки (50 мкл/лунку) в покрытые планшеты для ELISA и инкубировали в течение 1,5 ч при комнатной температуре. Планшеты промывали 4 раза в автоматическом промывающем устройстве и добавляли 4 мкг/мл (50 мкл/лунку) козьего поликлонального антитела против ABD (собственного производства). После инкубации в течение 1 ч планшеты промывали и в каждую лунку добавляли по 50 мкл противокозьего IgG-HRP (Jackson, кат. номер 711-035-152) в концентрации 20 нг/мл. После 1 часа дополнительной инкубации и последующей отмывки планшеты проявляли с помощью 50 мкл ТМВ на лунку и останавливали реакцию 50 мкл 2М H2SO4. Измеряли поглощение при 450 нм с использованием считывающего устройства для 96-луночных планшетов (Victor3).
Результаты
Катаболизм IgG: Результаты продемонстрировали снижение концентрации мышиных IgG у мышей, получавших FcRn-специфические полипептиды ZAZ3715 и ZAZ3824, соответственно (Фиг. 15). Результат, полученный с полипептидом ZAZ3824, показывает, что снижение уровней эндогенных IgG является дозозависимым, и наиболее выраженный эффект наблюдали через 72 ч. Следует отметить, что снижение уровней IgG, полученное с применением димеров, было больше, чем снижение, полученное с мономерным Z07918 в Примере 11, даже несмотря на то, что мономер вводили повторно и в более высокой дозе, чем димеры (сравнение Фиг. 15 и 10 через 72 ч). Таким образом, результаты показывают, что FcRn-специфические полипептиды, описанные здесь, могут блокировать рециркуляцию IgG, что приводит к усиленному катаболизму IgG и последующему снижению уровней IgG у мышей.
Фармакокинетический анализ: Фармакокинетические профили ZAZ3715 и ZAZ3824 показаны на Фиг. 16. Период полувыведения ZAZ3824 составил приблизительно 62 ч, что согласуется с периодом полувыведения, полученным в фармакокинетическом исследовании, представленном в Примере 6, демонстрируя, что помимо увеличения периода полувыведения в результате связывания альбумина, фармакокинетические свойства полипептидов дополнительно улучшаются за счет связывания с FcRn.
Пример 22
Усиление катаболизма IgG у яванских макаков посредством FcRn-связывающих димеров
В данном Примере эффект гомо- или гетеродимерных FcRn-связывающих полипептидов на катаболизм IgG исследуют на яванских макаках.
Материалы и методы
Исследование на животных: Получают гомо- и/или гетеродимерные FcRn-связывающие полипептиды, рекомбинантно слитые с РР013 (SEQ ID NO: 377). Примеры таких FcRn-связывающих FcRn включают SEQ ID NO: 371-376, раскрытые здесь.
Затем FcRn-связывающие димеры, включающие альбумин-связывающий домен, вводят яванским макакам в дозах 2, 0,4 и 0,1 мкмоль/кг. Димерные полипептиды инъецируют внутривенно в 0 ч. У обезьян берут кровь за 1 час до первого введения и ежедневно вплоть до суток 21, а затем раз в неделю вплоть до суток 50. Сыворотки получают и хранят при -20°C. Концентрацию IgG яванского макака определяют посредством ELISA.
Гомо- и/или гетеродимерные FcRn-связывающие полипептиды без альбумин-связывающего домена вводят яванским макакам в дозах 2, 0,4 и 0,1 мкмоль/кг. Димерные полипептиды вводят три раза в неделю в течение двух недель. У обезьян берут кровь за 1 час до первого введения и ежедневно вплоть до суток 21, а затем раз в неделю вплоть до суток 50. Сыворотки получают и хранят при -20°C. Концентрацию IgG яванского макака определяют посредством ELISA.
ELISA IgG яванского макака: Концентрацию IgG яванского макака в образцах сыворотки определяют с использованием набора для ELISA IgG человека/яванского макака (например, Mabtech 3850-1AD-6, который перекрестно реагирует с IgG человека и яванского макака), согласно описанию производителя.
Результаты
Ожидается, что результаты данного эксперимента демонстрируют дозозависимое снижение концентраций IgG яванского макака под воздействием FcRn-связывающих димеров, причем эффект более выражен у димеров, которые также содержат альбумин-связывающий домен.
Пример 23
Фармакокинетическое исследование димерных FcRn-связывающих полипептидов
В данном Примере исследуют период полувыведения в сыворотке гомо- и/или гетеродимерных FcRn-связывающих полипептидов в фармакокинетическом исследовании на мышах.
Материалы и методы
Фармакокинетическое исследование: FcRn-связывающие полипептиды, в виде гомо- и/или гетеродимеров, как таковые или рекомбинантно слитые с РР013 (SEQ ID NO: 377), вводят внутривенно (в/в) самцам мышей NMRI (Charles River, Germany) в дозе 92 нмоль/. Сыворотки у групп из трех мышей получают через 0,08, 6, 18, 78, 120, 168 и 240 часов. Концентрацию соответствующих полипептидов определяют посредством ELISA.
ELISA: Половину площади 96-луночных планшетов для ELISA покрывают при 4°C в течение ночи 50 мкг/мл козьего антитела (собственного производства), специфического в отношении Z вариантов в целом, разведенного до 4 мкг/мл в буфере для покрытия (50 мМ карбоната натрия, pH 9,6). Раствор антитела сливают и блокируют лунки 100 мкл PBSC в течение 1,5 ч при комнатной температуре. Сыворотки разводят в PBSC, содержащем 1% мышиную сыворотку (матрикс), от 1:100 до 1:51200 в серии двукратных разведений в планшете для разведения. В каждый планшет вносят раститрованные стандартным образом соответствующие полипептиды Z вариантов и четыре контроля качества (очень низкий, низкий, средний и высокий контроли), разведенные в матриксе. В каждую лунку переносят по 50 мкл разведения и инкубируют планшеты для ELISA в течение 1,5 ч при комнатной температуре. Планшеты промывают четыре раза PBST. Связанные полипептиды Z вариантов выявляют с помощью 50 мкл/лунку кроличьих Ig против РР013 (собственного производства) или 50 мкл/лунку мышиного mAb (моноклональное антитело) против Z (собственного производства), разведенных до 4 мкг/мл в PBSC. Затем планшеты инкубируют в течение 1,5 ч при комнатной температуре с последующими отмывками, как описано выше. Добавляют конъюгированное с HRP противокроличье антитело осла (Jackson laboratories, кат. номер 711-035-152), разведенное 1:20000 в PBSC, и планшеты инкубируют в течение 1 ч. После отмывки, как описано выше, в лунки добавляют 50 мкл субстрата ImmunoPure ТМВ и планшеты проявляют согласно рекомендациям производителя. После развития окраски в течение 15 минут измеряют поглощение при 450 нм с помощью считывающего устройства для многолуночных планшетов (Victor3). Значения поглощения анализируют с помощью программы GraphPad Prism 5 для определения концентраций (аппроксимация кривой посредством кубического сплайна) и площади под кривой (AUC). Затем концентрации откладывают на графике в виде их натуральных логарифмов относительно времени. Ожидается, что полученные кривые соответствуют двухкомпартментной модели, и рассчитывают терминальный период полувыведения как In2, разделенный на наклон, на основе последних трех моментов времени.
Результаты
Ожидается, что результаты описанного здесь эксперимента демонстрируют двухкомпартментную фазу элиминации с терминальным периодом полувыведения, составляющим приблизительно 60 мин для полипептидов без ABD и приблизительно 90 часов для полипептидов, содержащих ABD.
Пример 24
Создание, исследование стабильности и оценка связывания FcRn-связывающих полипептидов с модифицированным каркасом
В следующем Примере описаны FcRn-связывающие Z варианты с модифицированным каркасом, демонстрирующие улучшенную стабильность при повышенных температурах. Z варианты Z17347 (SEQ ID NO: 358) с аминокислотными заменами N52S и D53E, а также Z17348 (SEQ ID NO: 359) с аминокислотными заменами D36R, D37Q, S39E, N52S и D53E сравнивают с их родительской молекулой Z11948 (SEQ ID NO: 354) по стабильности и способности связывать FcRn.
Материалы и методы
Создание полипептидов с модифицированным каркасом: Z17347 (SEQ ID NO: 358), Z17348 (SEQ ID NO: 359) и Z11948 (SEQ ID NO: 354) клонировали с N-концевой гексагистидиновой меткой (His6) и полученные конструкции кодировали полипептиды в формате MGSSHHHHHHLQ-[Z#####]. Мутации вводили в плазмиды, кодирующие модифицированные Z варианты, используя пары перекрывающихся олигонуклеотидных праймеров, кодирующих необходимые аминокислотные замены, и применяя стандартные методы молекулярной биологии. Правильные последовательности плазмид подтверждали путем секвенирования ДНК.
Клетки Е. coli (штамм Т7Е2) (GeneBridge) трансформировали плазмидами, содержащими фрагменты генов, кодирующих исходные Z варианты и Z варианты с модифицированным каркасом. Клетки культивировали при 37°C в среде TSB-YE с добавлением 50 мкг/мл канамицина и затем индуцировали экспрессию белка путем добавления IPTG. Осажденные клетки разрушали с использованием гомогенизатора FastPrep®-24 (Nordic Biolabs) и удаляли клеточный дебрис путем центрифугирования. Каждый супернатант, содержащий Z вариант в виде белка с His6-меткой, очищали посредством металл-аффинной хроматографии (IMAC) с использованием колонок His GraviTrap™ (GE Healthcare) согласно инструкциям производителя. У очищенных Z вариантов заменяли буфер на забуференный фосфатом физиологический раствор (PBS; 1,47 мМ KH2PO4, 8,1 мМ Na2HPO4, 137 мМ NaCl, 2,68 мМ KCl, pH 7,4) с использованием обессоливающих колонок PD-10 (GE Healthcare). Правильную идентичность каждого полипептида подтверждали с помощью SDS-PAGE и HPLC-MS.
Спектроскопия кругового дихроизма: Для определения температур плавления (Tm) и оценки возможных изменений во вторичной структуре полипептидов по изобретению в результате аминокислотных замен анализировали спектры кругового дихроизма (CD), как описано в Примере 3.
Сравнительное исследование стабильности: Z-варианты, несущие His6-метку, находившиеся в PBS с pH 7,4, разводили до 1 мг/мл и аликвоты объемом 200 мкл инкубировали при 37°C в течение 2 недель. Образцы, взятые до и после теста на стабильность, анализировали посредством SDS-PAGE, используя 10% гели Bis-Tris NuPAGE (Invitrogen) и загружая в каждую лунку по 5 мкг белка. Стабильность оценивали по внешнему виду новых вариантов после инкубации при повышенной температуре и сравнивали мутированные варианты с исходным полипептидом.
Исследование связывания полипептидов с модифицированным каркасом: Z-варианты, несущие His6-метку, дополнительно оценивали на предмет сохранения способности связывать FcRn после внесения изменений в каркас, а также после подвергания тесту на стабильность, то есть инкубации при 37°C в течение 2 недель. Сравнительне кинетические константы (kon и koff) и аффинности (KD) определяли с помощью прибора Biacore 2000. Белок-мишень, FcRn человека (Biorbyt, кат. номер orb 84388), иммобилизовали на слое карбоксилированного декстрана поверхности чипа СМ5 (GE Healthcare), посредством химии аминного связывания согласно протоколу производителя и используя HBS-EP в качестве подвижного буфера. Поверхность одной проточной ячейки чипа активировали и дезактивировали, чтобы использовать в качестве пустой пробы при инъекциях аналитов. FcRn человека иммобилизовали на поверхности приблизительно до уровня 750 RU. Z варианты разводили в подвижном буфере до конечных концентраций 3,33, 10 и 30 нМ и инъецировали в течение 3 мин с последующей диссоциацией в подвижном буфере в течение 15 мин. После каждой инъекции аналита выполняли регенерацию тремя пульсовыми введениями HBS-EP с последующим уравновешиванием подвижным буфером в течение 10 мин. Кинетические константы рассчитывали на основании сенсограмм в программе BiaEvaluation 4.1 (GE Healthcare), используя модель 1:1 Лангмюра. Кривые, полученные на поверхности без лиганда, вычитали из кривых, полученных на поверхности с лигандом, а данные, полученные в циклах с введением буфера, вычитали из данных, полученных в циклах с введением исследуемого образца, для поправки на возможный дрейф сигнала.
Результаты
Анализ спектроскопии кругового дихроизма: Tm каждого соответствующего Z варианта, которую определяли в точке перегиба графика зависимости сигнала CD от температуры, приведены в Таблице 19. Мутированные Z варианты демонстрировали сохраненную альфа-спиральную структуру и обратимый рефолдинг после нагревания до 90°C.
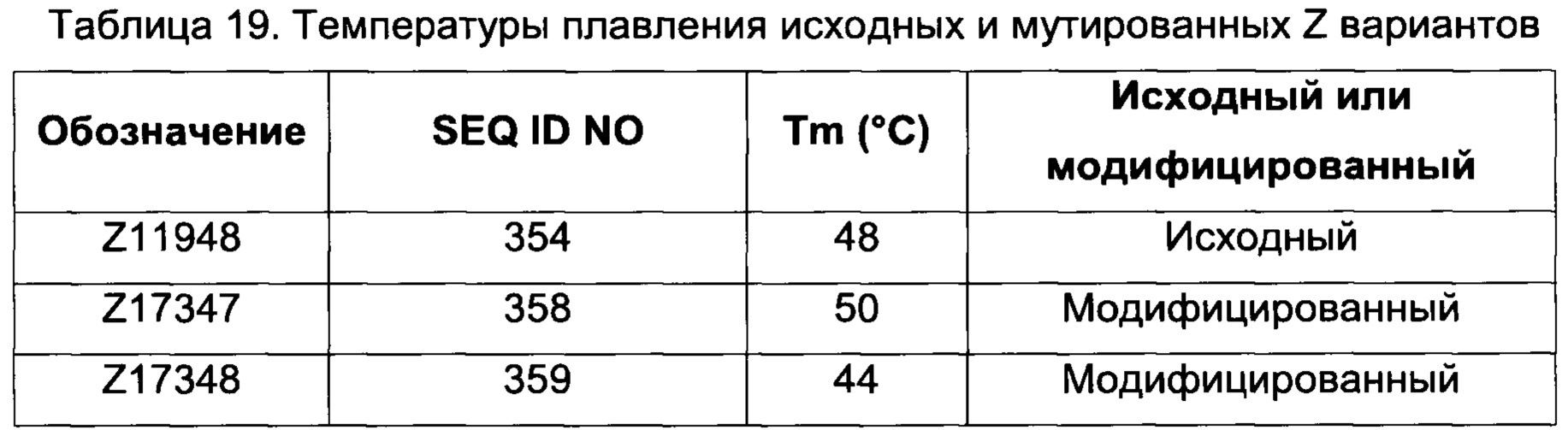
Сравнительное исследование стабильности: Z варианты с модифицированным каркасом, Z17347 и Z17348, демонстрировали улучшенную стабильность по сравнению с исходным полипептидом Z11948. Вторая полоса, втдная на геле непосредственно над основной полосой для Z11948, не была видна в образцах Z17347 и Z17348 (Фиг. 17), то есть мутации каркаса предотвращают образование альтернативных видов, наблюдаемых в образце полипептида с исходным каркасом.
Оценка связывания полипептидов с модифицированным каркасом: Сравнительные кинетические константы для FcRn-связывающих Z вариантов приведены в Таблице 20. На аффинность незначительно влияли аминокислотные замены ND на SE в положении 52-53, как у Z17347 (SEQ ID NO: 358), а также аминокислотные замены ND на SE в положении 52-53 в сочетании с D36R, D37Q и S39E, как у Z17348 (SEQ ID NO: 359), и были получены функциональные связывающие агенты с KD около 10-9 М. У исследованных вариантов связывающая способность также сохранялась через 2 недели инкубации при 37°C.
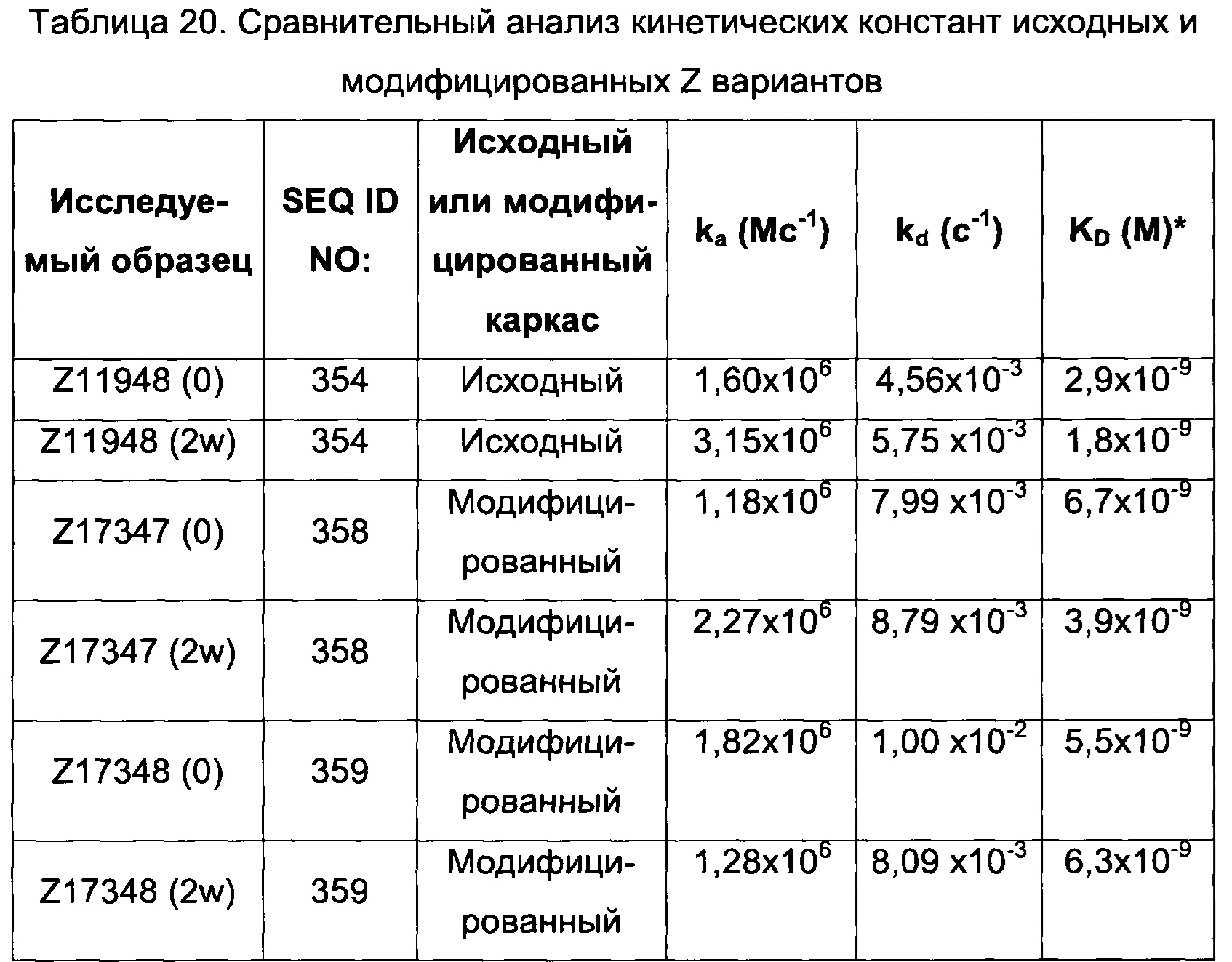
* Значения KD не следует считать абсолютными, поскольку их определяли в целях сравнения и использовали только ограниченное число концентраций образцов.
Пример 25
Создание и оценка дополнительных FcRn-связывающих полипептидов с модифицированным каркасом
В данном Примере анализировали стабильность и связывание с FcRn у дополнительных вариантов с заменами каркасных аминокислот N52S и D53E, а некоторые также с аминокислотной заменой Y5F, по сравнению с их соответствующим родительским FcRn-связывающим Z вариантом. Результаты показывают, что у мутированных вариантов сохраняются структура, стабильность и способность связывать FcRn.
Материалы и методы
Создание полипептидов с модифицированным каркасом: Аминокислотные замены N52S и D53E вводили в плазмиды, кодирующие Z13578 (SEQ ID NO: 20), Z13583 (SEQ ID NO: 23), Z13616 (SEQ ID NO: 41), Z13621 (SEQ ID NO: 44) и Z13674 (SEQ ID NO: 75), несущие His6-метку, с помощью стандартных методик молекулярной биологии с получением соответствующих Z вариантов Z18614 (SEQ ID NO: 360), Z18615 (SEQ ID NO: 361), Z18616 (SEQ ID NO: 362), Z18617 (SEQ ID NO: 363) и Z18618 (SEQ ID NO: 364). В результате дополнительных модификаций в положении 5, где остаток тирозина был заменен на фенилаланин, а также N-концевых модификаций, благодаря которым начальными аминокислотными остатками становились АЕ вместо VD, были получены Z варианты Z18632 (SEQ ID NO: 365), Z18633 (SEQ ID NO: 366) и Z18634 (SEQ ID NO: 367), имеющие связывающие мотивы (ВМ), идентичные Z13578, Z13616 и Z13621, соответственно. Культивирование и очистку осуществляли по существу, как описано в Примере 3 и Примере 9. Пять Z вариантов, Z18614-Z18618, очищали только с помощью IMAC, тогда как три Z варианта, Z18632-Z18634, дополнительно очищали посредством RPC.
Анализ CD: Для определения температур плавления (Tm) и оценки возможных изменений во вторичной структуре мутированных Z вариантов по сравнению с их родительским Z вариантом выполняли анализ CD, как описано в Примере 3.
Анализ связывания Biacore: Анализ связывания при pH 6,0 с использованием прибора Biacore осуществляли по существу, как описано в Примере 3. Серию концентраций (270, 90, 30, 10 и 3,3 нМ) Z вариантов, несущих His6-метку, Z18632, Z18633 и Z18634, и их соответствующих родительских Z вариантов, Z13578, Z13616 и Z13621, инъецировали в течение 4 мин со скоростью 30 мкл/мин поверх hFcRn (Biorbyt, кат. номер orb84388) и cFcRn (Biorbyt, кат. номер orb99075), иммобилизованных в различных проточных ячейках поверхности чипа СМ5. В качестве подвижного буфера и для разведения Z вариантов, несущих His6-метку, использовали 0,005% РСТ с pH 6,0. Перед началом следующего цикла проводили диссоциацию в подвижном буфере в течение 20 мин, с последующей регенерацией поверхности путем 3×30 сек пульсовых введений 0,005% РСТ с pH 7,4 и уравновешиванием в течение 15 минут.
Результаты
Культивирование и выделение: FcRn-связывающие Z варианты сконструировали с N-концевой His6-меткой и получили в Е. coli. Анализ каждого полученного препарата белка с помощью SDS-PAGE показал, что они преимущественно содержали Z вариант. Правильную идентичность и молекулярную массу каждого FcRn-связывающего Z варианта подтверждали с помощью HPLC-MS анализа.
Анализ CD: Tm мутированных Z вариантов, связывающих FcRn, были идентичны или почти идентичны Tm соответствующего родительского Z варианта (Таблица 21). Кроме того, у всех семи Z вариантов наблюдался обратимый фолдинг посредством наложения спектров, полученных до и после нагревания до 90°C.
Анализ связывания Biacore: У трех мутированных Z вариантов и их соответствующих родительских Z вариантов, Z18632/Z13578 (SEQ ID NO: 365/20), Z18633/Z13616 (SEQ ID NO: 366/41) и Z18634/Z13621 (SEQ ID NO: 367/44), попарно сравнивали профили связывания, характеризующие взаимодействия с FcRn при pH 6,0. Наложения сенсограмм, полученных при инъекциях 90 нМ Z вариантов над поверхностями с hFcRn и cFcRn, демонстрируют, что мутированные Z варианты сохраняли способность связываться с FcRn человека и яванского макака (Фиг. 18 А-В). FcRn иммобилизовали на поверхности чипа до уровня 1577 RU в случае FcRn человека и 1098 RU в случае FcRn яванского макака.
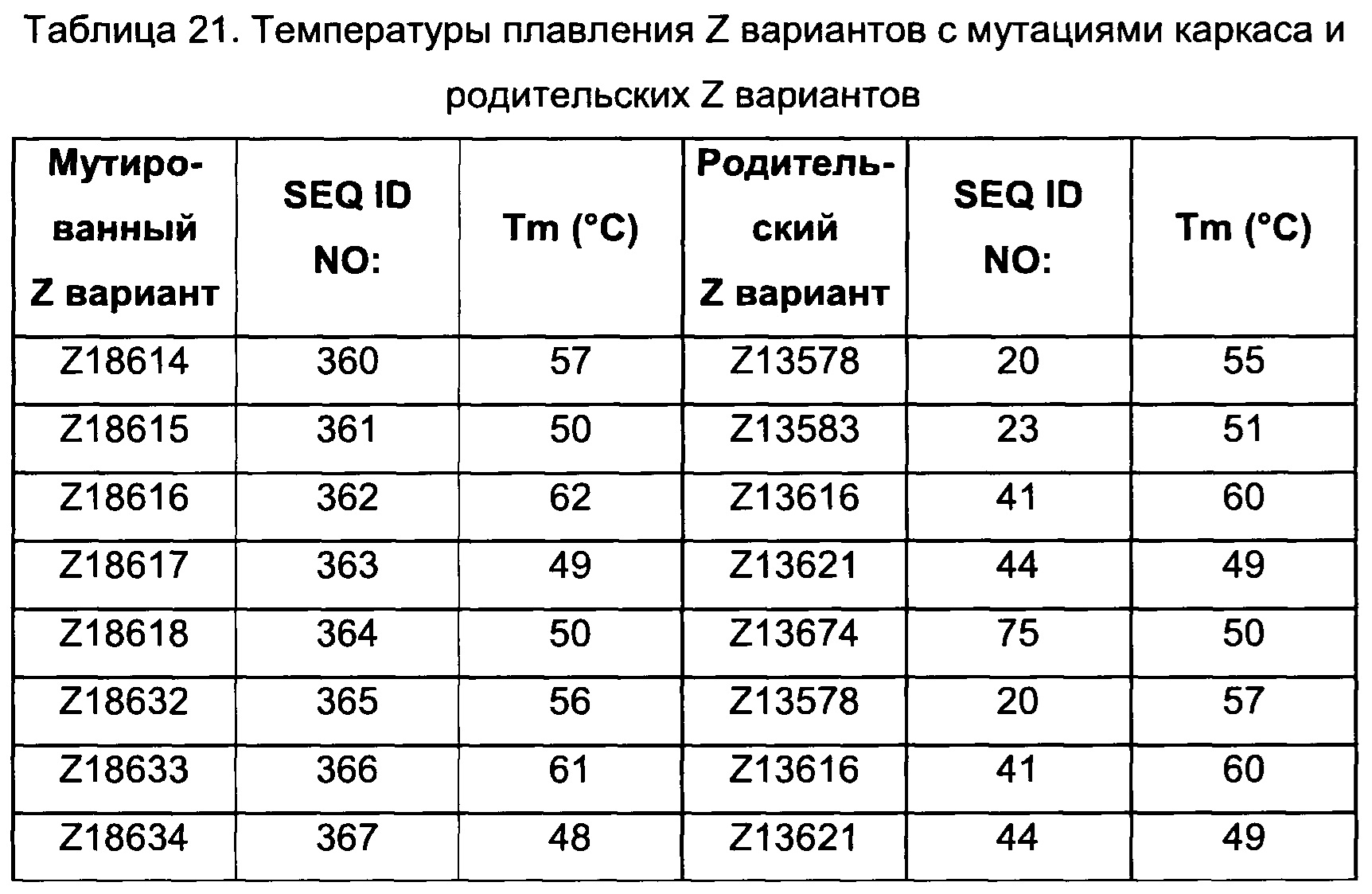
ПРОНУМЕРОВАННЫЙ ПЕРЕЧЕНЬ ВОПЛОЩЕНИЙ
1. FcRn-связывающий димер, содержащий первую мономерную единицу, вторую мономерную единицу и аминокислотный линкер, где каждая из указанных первой и второй мономерных единиц содержит FcRn-связывающий мотив (ВМ), который состоит из аминокислотной последовательности
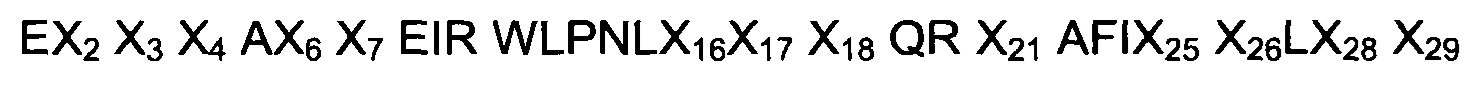
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, G, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из А, Е, F, G, Н, I, K, Q, R, S и V;
Х7 выбран из A, F, Н, K, N, Q, R, S и V;
X16 выбран из N и Т;
Х17 выбран из F, W и Y;
X18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из D, Е, G, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х26 выбран из K и S;
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y; и
X29 выбран из D и R,
и где указанный FcRn-связывающий димер связывает FcRn с большей связывающей способностью по сравнению с указанной первой мономерной единицей или указанной второй мономерной единицей в отдельности.
2. FcRn-связывающий димер по п. 1, где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, F, Н, I, K, L, М, N, Q, R, S, Т, V, W и Y;
Х4 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х6 выбран из А, Е, F, G, Н, I, K, Q, R и S;
Х7 выбран из A, F, Н, K, N, Q, R, S и V;
X16 выбран из N и Т;
Х17 выбран из F и Y;
X18 представляет собой D;
Х21 представляет собой V;
Х25 выбран из D, Е, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х26 выбран из K и S;
Х28 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V и W, и
Х29 выбран из D и R.
3. FcRn-связывающий димер по п. 1, где ВМ по меньшей мере одной из указанных первой и второй мономерных единиц состоит из аминокислотной последовательности, выбранной из
i) 
где, независимо друг от друга,
Х2 выбран из A, D, Е, F, Н, I, K, L, N, Q, R, S, Т, V, W и Y;
Х3 выбран из A, D, Е, G, Н, K, L, М, N, Q, R, S, Т, V и Y;
Х4 выбран из A, D, Е, F, G, I, K, L, N, Q, R, S, Т, V и Y;
Х6 выбран из A, G, K, R, S и V;
Х17 выбран из F, W и Y;
X18 выбран из A, D, Е и N;
Х21 выбран из A, S, V и W;
Х25 выбран из D, G, Н, K, L, N, R, V и W;
Х28 выбран из A, D, Е, Н, K, L, N, Q, R, S, Т, W и Y;
и
ii) аминокислотной последовательности, которая по меньшей мере на 96% идентична последовательности, определенной в i).
4. FcRn-связывающий димер по любому из пп. 1-3, где Х6Х7 выбран из АН и GH в по меньшей мере одной из указанных первой и второй мономерных единиц.
5. FcRn-связывающий димер по п. 4, где Х6Х7 представляет собой АН в по меньшей мере одной из указанных первой и второй мономерных единиц.
6. FcRn-связывающий димер по п. 4, где Х6Х7 представляет собой GH в по меньшей мере одной из указанных первой и второй мономерных единиц.
7. FcRn-связывающий димер по любому из предшествующих пп., где X17X18 выбран из FD и YD в по меньшей мере одной из указанных первой и второй мономерных единиц.
8. FcRn-связывающий димер по п. 7, где X17X18 представляет собой FD в по меньшей мере одной из указанных первой и второй мономерных единиц.
9. FcRn-связывающий димер по любому из предшествующих пп., где последовательность ВМ по меньшей мере одной из указанных первой и второй мономерных единиц удовлетворяет по меньшей мере трем из шести условий I-VI:
I. Х6 выбран из A, G, K и S, например, в частности, А;
II. Х7 представляет собой Н;
III. Х17 выбран из F и Y, например, в частности, F;
IV. Х18 представляет собой D;
V. Х21 выбран из V и W, например, в частности, V;
VI. Х25 выбран из Н и R, например, в частности, Н.
10. FcRn-связывающий димер по п. 9, где последовательность удовлетворяет по меньшей мере четырем из шести условий I-VI.
11. FcRn-связывающий димер по п. 10, где последовательность удовлетворяет по меньшей мере пяти из шести условий I-VI.
12. FcRn-связывающий димер по п. 11, где последовательность удовлетворяет всем шести условиям I-VI.
13. FcRn-связывающий димер по любому из предшествующих пп., где указанные первая и вторая мономерные единицы содержат идентичные последовательности ВМ.
14. FcRn-связывающий димер по любому из пп. 1-12, где указанные первая и вторая мономерные единицы содержат разные последовательности ВМ.
15. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит FcRn-связывающий мотив ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353, такой как группа, состоящая из SEQ ID NO: 17-352.
16. FcRn-связывающий димер по п. 15, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140 и SEQ ID NO: 353.
17. FcRn-связывающий димер по п. 16, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2 и SEQ ID NO: 17-140, такой как группа, состоящая из SEQ ID NO: 17-140.
18. FcRn-связывающий димер по п. 17, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140, такой как группа, состоящая из SEQ NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140.
19. FcRn-связывающий димер по п. 16, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73 и SEQ ID NO: 75-77.
20. FcRn-связывающий димер по п. 18 или п. 19, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77.
21. FcRn-связывающий димер по п. 20, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77.
22. FcRn-связывающий димер по п. 21, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75.
23. FcRn-связывающий димер по п. 22, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 75.
24. FcRn-связывающий димер по п. 22, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 41, группа, состоящая из SEQ ID NO: 20 и SEQ ID NO: 44, или группа, состоящая из SEQ ID NO: 41 и SEQ ID NO: 44.
25. FcRn-связывающий димер по п. 22, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44.
26. FcRn-связывающий димер по п. 24 или п. 25, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности SEQ ID NO: 44.
27. FcRn-связывающий димер по любому из предшествующих пп., где обе из указанных первой и второй мономерных единиц независимо содержат ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной, как описано в любом из пп. 15-26.
28. FcRn-связывающий димер по п. 27, где обе из указанных первой и второй мономерных единиц независимо содержат ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44.
29. FcRn-связывающий димер по п. 28, где обе из указанных первой и второй мономерных единиц содержат ВМ, соответствующий последовательности от положения 8 до положения 36 в последовательности SEQ ID NO: 44.
30. FcRn-связывающий димер по любому из предшествующих пп., где указанный FcRn-связывающий мотив (ВМ) по меньшей мере в одном из указанных первого и второго мономеров является частью белкового домена с трехспиральным пучком.
31. FcRn-связывающий димер по п. 30, где указанный ВМ по существу образует часть двух спиралей с соединительной петлей в составе указанного белкового домена с трехспиральным пучком.
32. FcRn-связывающий димер по п. 31, где указанный белковый домен с трехспиральным пучком выбран из доменов бактериальных рецепторов.
33. FcRn-связывающий димер по п. 32, где указанный белковый домен с трехспиральным пучком выбран из доменов белка A Staphylococcus aureus или их производных.
34. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит связывающий модуль (BMod), который состоит из аминокислотной последовательности, выбранной из:
III) 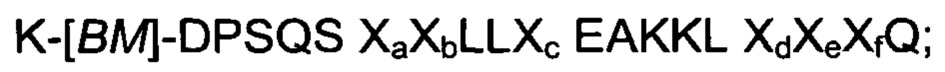
где
[ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29, при условии, что Х29 представляет собой D;
Ха выбран из А и S;
Xb выбран из N и Е;
Хс выбран из A, S и С;
Xd выбран из Е, N и S;
Хе выбран из D, Е и S;
Xf выбран из А и S;
и
IV) аминокислотной последовательности, которая по меньшей мере на 93% идентична последовательности, определенной в (III).
35. FcRn-связывающий димер по любому из пп. 1-33, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит связывающий модуль (BMod), который состоит из аминокислотной последовательности, выбранной из:
V) 
где
[ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29, при условии, что Х29 представляет собой R;
Ха выбран из А и S;
Xb выбран из N и Е;
Хс выбран из A, S и С;
Xd выбран из Е, N и S;
Хе выбран из D, Е и S;
Xf выбран из А и S;
и
IV) аминокислотной последовательности, которая по меньшей мере на 93% идентична последовательности, определенной в (V).
36. FcRn-связывающий димер по п. 34, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-353, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-352 и SEQ ID NO: 360-364.
37. FcRn-связывающий димер по п. 36, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140, SEQ ID NO: 353, SEQ ID NO: 358 и SEQ ID NO: 360-364.
38. FcRn-связывающий димер по п. 37, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-140, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-140 и SEQ ID NO: 360-364.
39. FcRn-связывающий димер по п. 38, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125, SEQ ID NO: 127-140, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125, SEQ ID NO: 127-140 и SEQ ID NO: 360-364.
40. FcRn-связывающий димер по п. 36, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77, SEQ ID NO: 353, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 360-364.
41. FcRn-связывающий димер по п. 39 или п. 40, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73, SEQ ID NO: 75-77, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 360-364.
42. FcRn-связывающий димер по п. 41, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 77, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 77 и SEQ ID NO: 360-364.
43. FcRn-связывающий димер по п. 42, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 358 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 75 и SEQ ID NO: 360-364.
44. FcRn-связывающий димер по п. 43, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 75, SEQ ID NO: 358, SEQ ID NO: 361 и SEQ ID NO: 364, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 75, SEQ ID NO: 361 и SEQ ID NO: 364.
45. FcRn-связывающий димер по п. 43, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 360, SEQ ID NO: 362 и SEQ ID NO: 363, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 360 и SEQ ID NO: 362; группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 44, SEQ ID NO: 360 и SEQ ID NO: 363; или группы, состоящей из SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 362 и SEQ ID NO: 363.
46. FcRn-связывающий димер по п. 43, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 358, SEQ ID NO: 361 и SEQ ID NO: 363, такой как группа, состоящая из SEQ ID NO: 23, SEQ ID NO: 44, SEQ ID NO: 361 и SEQ ID NO: 363.
47. FcRn-связывающий димер по п. 46, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности SEQ ID NO: 44.
48. FcRn-связывающий димер по любому из пп. 1-13 и 15-47, где обе из указанных первой и второй мономерных единиц содержат BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, как определено в любом из пп. 36-47.
49. FcRn-связывающий димер по п. 48, где обе из указанных первой и второй мономерных единиц содержат BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности, выбранной из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 75 и SEQ ID NO: 360-364, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 360, SEQ ID NO: 362 и SEQ ID NO: 363.
50. FcRn-связывающий димер по п. 49, где обе из указанных первой и второй мономерных единиц содержат BMod, соответствующий последовательности от положения 7 до положения 55 в последовательности SEQ ID NO: 44.
51. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
VII) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29, и Хс выбран из A, S и С; и
VIII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (VII).
52. FcRn-связывающий димер по любому из пп. 1-50, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
IX) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29, и Хс выбран из A, S и С; и
х) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (IX).
53. FcRn-связывающий димер по любому из пп. 1-50, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XI) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29, и Хс выбран из А и С; и
xii) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XI).
54. FcRn-связывающий димер по п. 33, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из:


где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29.
55. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XIII) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29; и
XIV) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XIII).
56. FcRn-связывающий димер по п. 55, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XIII), выбранную из группы, состоящей из: SEQ ID NO: 354-357, такую, как выбранная из SEQ ID NO: 354 и 357.
57. FcRn-связывающий димер по п. 56, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XIII), которая представляет собой SEQ ID NO: 357.
58. FcRn-связывающий димер по любому из пп. 1-54, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XV) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29; и
XVI) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XV).
59. FcRn-связывающий димер по п. 58, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XV), выбранную из группы, состоящей из SEQ ID NO: 365-367.
60. FcRn-связывающий димер по любому из пп. 1-54, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XVII) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29; и
XVIII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XVII).
61. FcRn-связывающий димер по п. 60, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XVII), выбранную из группы, состоящей из SEQ ID NO: 360-364.
62. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XIX) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29; и
XX) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XIX).
63. FcRn-связывающий димер по п. 62, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XIX), которая представляет собой SEQ ID NO: 359.
64. FcRn-связывающий димер по любому из пп. 1-54, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность, выбранную из группы, состоящей из:
XXI) 
где [ВМ] представляет собой FcRn-связывающий мотив, как определено в любом из пп. 1-29; и
XXII) аминокислотной последовательности, которая по меньшей мере на 94% идентична последовательности, определенной в (XXI).
65. FcRn-связывающий димер по п. 64, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность XXI), выбранную из группы, состоящей из SEQ ID NO: 1-353, такой как из группа, состоящая из SEQ ID NO: 17-352.
66. FcRn-связывающий димер по п. 65, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1-15, SEQ ID NO: 17-140 и SEQ ID NO: 353 или содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1-2 и SEQ ID NO: 17-140, такой как из группа, состоящая из SEQ ID NO: 17-140.
67. FcRn-связывающий димер по п. 66, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1-2, SEQ ID NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140, такой как группа, состоящая из SEQ NO: 17-92, SEQ ID NO: 94-103, SEQ ID NO: 105-125 и SEQ ID NO: 127-140.
68. FcRn-связывающий димер по п. 67, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1-8, SEQ ID NO: 13, SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73, SEQ ID NO: 75-77 и SEQ ID NO: 353, такой как группа, состоящая из SEQ ID NO: 19-20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 70, SEQ ID NO: 73 и SEQ ID NO: 75-77.
69. FcRn-связывающий димер по п. 66 или п. 68, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 28, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 73 и SEQ ID NO: 75-77.
70. FcRn-связывающий димер по п. 69, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75 и SEQ ID NO: 77.
71. FcRn-связывающий димер по п. 70, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44 и SEQ ID NO: 75.
72. FcRn-связывающий димер по п. 71, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 75, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 75.
73. FcRn-связывающий димер по п. 71, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 20, SEQ ID NO: 41 и SEQ ID NO: 44.
74. FcRn-связывающий димер по п. 71, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), выбранную из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 23 и SEQ ID NO: 44, такой как группа, состоящая из SEQ ID NO: 23 и SEQ ID NO: 44.
75. FcRn-связывающий димер по пп. 73 или 74, где по меньшей мере одна из указанных первой и второй мономерных единиц содержит последовательность (XXI), которая представляет собой SEQ ID NO: 44.
76. FcRn-связывающий димер по любому из пп. 1-13 и 15-75, где обе из указанных первой и второй мономерных единиц соответствуют последовательности, выбранной из группы, определенной в любом из пп. 55, 56 и 62-75.
77. FcRn-связывающий димер по п. 76, где обе из указанных первой и второй мономерных единиц соответствуют последовательности, выбранной из группы, состоящей из SEQ ID NO: 1, SEQ ID NO: 20, SEQ ID NO: 23, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 65, SEQ ID NO: 75, SEQ ID NO: 354, SEQ ID NO: 357 и SEQ ID NO: 360-367, такой как группа, состоящая из SEQ ID NO: 20, SEQ ID NO: 41, SEQ ID NO: 44, SEQ ID NO: 360, SEQ ID NO: 362, SEQ ID NO: 363, SEQ ID NO: 365, SEQ ID NO: 366 и SEQ ID NO: 367.
78. FcRn-связывающий димер по п. 77, где обе из указанных первой и второй мономерных единиц соответствуют SEQ ID NO: 1 или SEQ ID NO: 357.
79. FcRn-связывающий димер по п. 77, где обе из указанных первой и второй мономерных единиц соответствуют SEQ ID NO: 20, SEQ ID NO: 360 или SEQ ID NO: 365.
80. FcRn-связывающий димер по п. 77, где обе из указанных первой и второй мономерных единиц соответствуют SEQ ID NO: 41, SEQ ID NO: 362 или SEQ ID NO: 366.
81. FcRn-связывающий димер по п. 77, где обе из указанных первой и второй мономерных единиц соответствуют SEQ ID NO: 44, SEQ ID NO: 363 или SEQ ID NO: 367.
82. FcRn-связывающий димер по любому из предшествующих пп., где указанный линкер выбран из группы, состоящей из гибких аминокислотных линкеров, жестких аминокислотных линкеров и расщепляемых аминокислотных линкеров.
83. FcRn-связывающий димер по п. 82, где указанный линкер расположен между указанной первой мономерной единицей и указанной второй мономерной единицей.
84. FcRn-связывающий димер по п. 82 или п. 83, где указанный линкер представляет собой гибкий линкер, содержащий аминокислотные остатки, выбранные из группы, состоящей из глицина, серина и треонина.
85. FcRn-связывающий димер по п. 84, где указанный линкер имеет общую формулу, выбранную из
(GnSm)p и (SnGm)p,
где, независимо,
n равно от 1 до 7,
m равно от 0 до 7,
сумма n и m составялет не более 8 и
p равно от 1 до 7.
86. FcRn-связывающий димер по п. 85, где n равно от 1 до 5.
87. FcRn-связывающий димер по любому из пп. 85-86, где m равно от 0 до 5.
88. FcRn-связывающий димер по любому из пп. 85-87, где р равно от 1 до 5.
89. FcRn-связывающий димер по любому из пп. 86-88, где n равно 4, m равно 1 и р равно от 1 до 4.
90. FcRn-связывающий димер по п. 89, где указанный линкер представляет собой (G4S)3.
91. FcRn-связывающий димер по п. 89, где указанный линкер представляет собой G4S.
92. FcRn-связывающий димер по любому из предшествующих пп., у которого способность связываться с FcRn по меньшей мере в 2 раза, например по меньшей мере в 3 раза, например по меньшей мере в 4 раза, например по меньшей мере в 5 раз, например по меньшей мере в 6 раз, например по меньшей мере в 7 раз, например по меньшей мере в 8 раз, например по меньшей мере в 9 раз, например по меньшей мере в 10 раз, например по меньшей мере в 25 раз, например по меньшей мере в 50 раз, например по меньшей мере в 100 раз выше, чем у соответствующей первой мономерной единицы или второй мономерной единицы в отдельности.
93. FcRn-связывающий димер по п. 92, у которого способность связываться с FcRn при pH 6,0 по меньшей мере в 2 раза, например по меньшей мере в 3 раза выше, чем у соответствующей первой мономерной единицы или второй мономерной единицы в отдельности.
94. FcRn-связывающий димер по п. 92, у которого способность связываться с FcRn при pH 7,4 по меньшей мере в 2 раза, например по меньшей мере в 3 раза, например по меньшей мере в 4 раз, например по меньшей мере в 5 раз, например по меньшей мере в 6 раз, например по меньшей мере в 7 раз, например по меньшей мере в 8 раз, например по меньшей мере в 9 раз, например по меньшей мере в 10 раз выше, чем у соответствующей первой мономерной единицы или второй мономерной единицы в отдельности.
95. FcRn-связывающий димер по любому из предшествующих пп., который способен связываться с FcRn при pH 6,0, так что значение KD взаимодействия составляет самое большее 1×10-7 М, например самое большее 1×10-8 М, например самое большее 1×10-9 М, например самое большее 1×10-10 М, например самое большее 1×10-11 М, например самое большее 1×10-12 М.
96. FcRn-связывающий димер по любому из предшествующих пп., где значение KD взаимодействия между FcRn-связывающим полипептидом и FcRn при pH 7,4 выше значения KD указанного взаимодействия при pH 6,0, например по меньшей мере в 2 раз выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз выше, например по меньшей мере в 50 раз выше, например по меньшей мере в 100 раз выше, чем значение KD указанного взаимодействия при pH 6,0.
97. FcRn-связывающий димер по любому из предшествующих пп., где значение KD взаимодействия между FcRn-связывающим полипептидом и FcRn при pH 7,4 составляет по меньшей мере 1×10-10 М, например по меньшей мере 1×10-9 М, например по меньшей мере 1×10-8 М, например по меньшей мере 1×10-7 М, например по меньшей мере 1×10-6 М, например по меньшей мере 1×10-5М.
98. FcRn-связывающий димер по любому из пп. 1-94, где значение KD указанного взаимодействия при pH 7,4 является таким же или более низким, чем значение KD указанного взаимодействия при pH 6,0.
99. FcRn-связывающий димер по любому из пп. 1-94, где значение KD указанного взаимодействия при pH 7,4 составляет самое большее 1×10-7 М, например самое большее 1×10-8 М, например самое большее 1×10-9 М, например самое большее 1×10-10 М, например самое большее 1×10-11 М, например самое большее 1×10-12 М.
100. FcRn-связывающий димер по любому из предшествующих пп., где по меньшей мере одна из указанных первой и второй мономерных единиц содержит по меньшей мере одну дополнительную аминокислоту на C-конце и/или на N-конце.
101. FcRn-связывающий димер по п. 100, где увеличение по меньшей мере на одну дополнительную аминокислоту улучшает или упрощает получение, выделение, стабилизацию in vivo или in vitro, присоединение или выявление полипептида.
102. Слитый белок или конъюгат, содержащий
- первую группировку, состоящую из FcRn-связывающего димера по любому из предшествующих пп., и
- вторую группировку, состоящую из полипептида, обладающего желаемой биологической активностью.
103. Слитый белок или конъюгат по п. 102, где период полувыведения указанного слитого белка или конъюгата in vivo дольше, чем период полувыведения in vivo полипептида, обладающего желаемой биологической активностью самого по себе.
104. Слитый белок или конъюгат по любому из пп. 102-103, где указанная желаемая биологическая активность представляет собой терапевтическую активность.
105. Слитый белок или конъюгат по любому из пп. 100-102, где указанная желаемая биологическая активность представляет собой связывающую активность с заданной мишенью.
106. Слитый белок или конъюгат по п. 105, где указанная заданная мишень представляет собой альбумин.
107. Слитый белок или конъюгат по п. 106, где указанная альбумин-связывающая активность обеспечивается альбумин-связывающим доменом белка G стрептококка или его производным.
108. Слитый белок или конъюгат по любому из пп. 106-107, где указанная альбумин-связывающая активность увеличивает период полувыведения слитого белка или конъюгата in vivo.
109. Слитый белок или конъюгат по любому из пп. 103-104, где указанная желаемая биологическая активность представляет собой ферментативную активность.
110. Слитый белок или конъюгат по любому из пп. 103-105, где вторая группировка, обладающая желаемой биологической активностью, представляет собой терапевтически активный полипептид.
111. Слитый белок или конъюгат по любому из пп. 103-105 и 109-110, где вторая группировка, обладающая желаемой биологической активностью, выбрана из группы, состоящей из ферментов, гормонов, факторов роста, хемокинов и цитокинов.
112. FcRn-связывающий димер, слитый белок или конъюгат по любому из предшествующих пп., который ингибирует связывание IgG с FcRn.
113. FcRn-связывающий димер, слитый белок или конъюгат по п. 112, который связывается с FcRn, так что способность FcRn-связывающего димера блокировать связывание IgG с FcRn по меньшей мере в 2 раза выше, например по меньшей мере в 3 раза выше, например по меньшей мере в 4 раза выше, например по меньшей мере в 5 раз выше, например по меньшей мере в 10 раз, например по меньшей мере в 15 раз, например по меньшей мере в 20 раз, например по меньшей мере в 25 раз выше по сравнению с блокирующей способностью соответствующей первой или второй мономерной единицы в отдельности.
114. FcRn-связывающий димер, слитый белок или конъюгат по п. 112 или п. 113, где значение KD взаимодействия между указанным FcRn-связывающим полипептидом, слитым белком или конъюгатом и FcRn ниже, чем значение KD взаимодействия между IgG и FcRn.
115. FcRn-связывающий димер, слитый белок или конъюгат по любому из предшествующих пп., дополнительно содержащий метку.
116. FcRn-связывающий димер, слитый белок или конъюгат по п. 115, где указанная метка выбрана из группы, состоящей из флуоресцентных красителей и металлов, хромофорных красителей, хемилюминесцентных соединений и биолюминесцентных белков, ферментов, радионуклидов и радиоактивных частиц.
117. FcRn-связывающий димер, слитый белок или конъюгат по любому из предшествующих пп., который включает хелатирующую среду, обеспечиваемую полиаминополикарбоксилатным хелатором, конъюгированным с FcRn-связывающим полипептидом через тиоловую группу остатка цистеина или аминогруппу остатка лизина.
118. FcRn-связывающий димер, слитый белок или конъюгат по п. 117, где полиаминополикарбоксилатный хелатор представляет собой 1,4,7,10-тетраазациклододекан-1,4,7,10-тетрауксусную кислоту или ее производное.
119. FcRn-связывающий димер, слитый белок или конъюгат по п. 118, где производное 1,4,7,10-тетраазациклододекан-1,4,7,10-тетрауксусной кислоты представляет собой 1,4,7,10-тетраазациклододекан-1,4,7-трис-уксусной кислоты-10-малеимидоэтилацетамид.
120. FcRn-связывающий димер, слитый белок или конъюгат по п. 117, где полиаминополикарбоксилатный хелатор представляет собой 1,4,7-триазациклононан-1,4,7-триуксусную кислоту или ее производное.
121. FcRn-связывающий димер, слитый белок или конъюгат по п. 117, где полиаминополикарбоксилатный хелатор представляет собой диэтилентриаминпентауксусную кислоту или ее производное.
122. Полинуклеотид, кодирующий полипептид по любому из пп. 1-114.
123. Экспрессирующий вектор, содержащий полинуклеотид по п. 122.
124. Клетка-хозяин, содержащая экспрессирующий вектор по п. 123.
125. Способ получения полипептида по любому из пп. 1-114, включающий
- культивирование клетки-хозяина по п. 124 в условиях, позволяющих указанному полипептиду экспрессироваться с указанного вектора экспрессии, и
- выделение указанного полипептида.
126. Композиция, содержащая FcRn-связывающий димер, слитый белок или конъюгат по любому из пп. 1-121 и по меньшей мере один фармацевтически приемлемый эксципиент или носитель.
127. Композиция по п. 126, дополнительно содержащая по меньшей мере один дополнительный активный агент.
128. Композиция по любому из пп. 126-127, выполеннная с возможностью введения способом, выбранным из группы, состоящей из перорального введения, интраназального введения, легочного введения, вагинального введения, ректального введения, внутривенной инъекции, внутрибрюшинной инъекции, внутримышечной инъекции, подкожной инъекции и внутрикожной инъекции.
129. FcRn-связывающий димер, слитый белок или конъюгат по любому из пп. 1-121 или композиция по любому из пп. 126-128 для применения в качестве лекарственного средства.
130. FcRn-связывающий димер, слитый белок, конъюгат или композиция для применения по п. 129, где указанное лекарственное средство предназначено для лечения или профилактики аутоиммунного состояния.
131. FcRn-связывающий димер, слитый белок, конъюгат или композиция для применения по п. 129, где указанное лекарственное средство предназначено для лечения или профилактики аллоиммунного состояния.
132. FcRn-связывающий димер, слитый белок, конъюгат или композиция для применения по п. 129, где указанное лекарственное средство предназначено для лечения или профилактики состояния, выбранного из группы, состоящей из эпилепсии и судорог.
133. Способ лечения или профилактики субъекта, нуждающегося в этом, включающий введение субъекту терапевтически или профилактически активного количества FcRn-связывающего димера, слитого белка или конъюгата по любому из пп. 1-121 или композиции по любому из пп. 126-128.
134. Способ по п. 133 для лечения или профилактики аутоиммунного состояния.
135. Способ по п. 133 для лечения или профилактики аллоиммунного состояния.
136. Способ по п. 133 для лечения или профилактики состояния, выбранного из группы, состоящей из эпилепсии и судорог.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> AFFIBODY AB
<120> NEW POLYPEPTIDES
<130> 21078161
<150> EP14185140.2
<151> 2014-09-17
<160> 388
<170> PatentIn version 3.5
<210> 1
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 1
Val Asp Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 2
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 2
Val Asp Ala Lys Tyr Ala Lys Glu Trp Met Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Glu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 3
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 3
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asn Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 4
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 4
Val Asp Ala Lys Tyr Ala Lys Glu Phe Glu Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 5
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 5
Val Asp Ala Lys Tyr Ala Lys Glu Arg Ser Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Leu
20 25 30
Lys Leu Thr Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 6
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 6
Val Asp Ala Lys Tyr Ala Lys Glu Ser Asp Ser Ala Val His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 7
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 7
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Asn Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Trp Ala Gln Arg Trp Ala Phe Ile His
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 8
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 8
Val Asp Ala Lys Tyr Ala Lys Glu Asp Asp Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Glu Gln Arg Trp Ala Phe Ile His
20 25 30
Lys Leu Tyr Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 9
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 9
Val Asp Ala Lys Tyr Ala Lys Glu Gln His Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu His Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 10
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 10
Val Asp Ala Lys Tyr Ala Lys Glu Asn Gln Gly Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Trp Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 11
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 11
Val Asp Ala Lys Tyr Ala Lys Glu Arg Thr Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 12
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 12
Val Asp Ala Lys Tyr Ala Lys Glu Arg Lys Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Ser Ala Phe Ile Lys
20 25 30
Lys Leu Glu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 13
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 13
Val Asp Ala Lys Tyr Ala Lys Glu Arg Gln Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 14
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 14
Val Asp Ala Lys Tyr Ala Lys Glu Glu Asp Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asn Gln Arg Ala Ala Phe Ile Asp
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 15
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 15
Val Asp Ala Lys Tyr Ala Lys Glu Asn Gln Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Gly
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 16
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 16
Val Asp Ala Lys Tyr Ala Lys Glu Ser Gly Tyr Ala Val His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 17
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 17
Val Asp Ala Lys Tyr Ala Lys Glu Ser Lys Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 18
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 18
Val Asp Ala Lys Tyr Ala Lys Glu Lys Lys Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 19
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 19
Val Asp Ala Lys Tyr Ala Lys Glu Trp His Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 20
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 20
Val Asp Ala Lys Tyr Ala Lys Glu Trp Thr Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 21
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 21
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 22
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 22
Val Asp Ala Lys Tyr Ala Lys Glu Gln Gln Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 23
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 23
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Lys Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 24
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 24
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Leu Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 25
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 25
Val Asp Ala Lys Tyr Ala Lys Glu Leu Lys Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 26
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 26
Val Asp Ala Lys Tyr Ala Lys Glu His Val Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 27
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 27
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Ala Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 28
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 28
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Ile Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 29
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 29
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asp Glu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 30
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 30
Val Asp Ala Lys Tyr Ala Lys Glu Leu Arg Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 31
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 31
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 32
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 32
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Lys Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 33
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 33
Val Asp Ala Lys Tyr Ala Lys Glu Ala His Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 34
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 34
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 35
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 35
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 36
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 36
Val Asp Ala Lys Tyr Ala Lys Glu Trp Lys Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 37
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 37
Val Asp Ala Lys Tyr Ala Lys Glu Trp Lys Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 38
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 38
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asp Leu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 39
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 39
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Ala Ala Arg His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 40
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 40
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Thr Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 41
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 41
Val Asp Ala Lys Tyr Ala Lys Glu Trp Gln Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 42
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 42
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 43
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 43
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ser Lys Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 44
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 44
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 45
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 45
Val Asp Ala Lys Tyr Ala Lys Glu Phe Met Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 46
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 46
Val Asp Ala Lys Tyr Ala Lys Glu Ser Lys Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 47
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 47
Val Asp Ala Lys Tyr Ala Lys Glu Val Ser Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 48
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 48
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Ser Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 49
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 49
Val Asp Ala Lys Tyr Ala Lys Glu Leu Met Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 50
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 50
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asn Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 51
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 51
Val Asp Ala Lys Tyr Ala Lys Glu Val His Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 52
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 52
Val Asp Ala Lys Tyr Ala Lys Glu Ser Thr Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 53
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 53
Val Asp Ala Lys Tyr Ala Lys Glu Trp Tyr Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 54
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 54
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asn Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 55
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 55
Val Asp Ala Lys Tyr Ala Lys Glu Val Glu Val Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 56
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 56
Val Asp Ala Lys Tyr Ala Lys Glu Phe Asn Phe Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 57
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 57
Val Asp Ala Lys Tyr Ala Lys Glu His Asp Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 58
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 58
Val Asp Ala Lys Tyr Ala Lys Glu Trp Met Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 59
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 59
Val Asp Ala Lys Tyr Ala Lys Glu Phe Ser Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 60
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 60
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asn Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 61
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 61
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 62
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 62
Val Asp Ala Lys Tyr Ala Lys Glu Ser Gln Ile Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 63
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 63
Val Asp Ala Lys Tyr Ala Lys Glu Val Ser Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 64
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 64
Val Asp Ala Lys Tyr Ala Lys Glu Asp Gln Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 65
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 65
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 66
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 66
Val Asp Ala Lys Tyr Ala Lys Glu Ser Lys Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 67
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 67
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Val Lys Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 68
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 68
Val Asp Ala Lys Tyr Ala Lys Glu Phe Ser Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 69
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 69
Val Asp Ala Lys Tyr Ala Lys Glu Trp Gln Phe Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 70
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 70
Val Asp Ala Lys Tyr Ala Lys Glu Trp Gln Ile Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 71
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 71
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 72
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 72
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Arg Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 73
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 73
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ala Ser Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 74
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 74
Val Asp Ala Lys Tyr Ala Lys Glu Val Gln Ser Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 75
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 75
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 76
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 76
Val Asp Ala Lys Tyr Ala Lys Glu Asn Gln Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 77
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 77
Val Asp Ala Lys Tyr Ala Lys Glu Ala Thr Ser Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 78
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 78
Val Asp Ala Lys Tyr Ala Lys Glu Asp Glu Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 79
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 79
Val Asp Ala Lys Tyr Ala Lys Glu Gln Asn Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 80
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 80
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Thr Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 81
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 81
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 82
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 82
Val Asp Ala Lys Tyr Ala Lys Glu Glu Met Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 83
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 83
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ser Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 84
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 84
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asp Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 85
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 85
Val Asp Ala Lys Tyr Ala Lys Glu Ala Glu Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 86
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 86
Val Asp Ala Lys Tyr Ala Lys Glu Glu Asp Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 87
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 87
Val Asp Ala Lys Tyr Ala Lys Glu Gln Lys Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 88
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 88
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asp Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 89
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 89
Val Asp Ala Lys Tyr Ala Lys Glu Ala Lys Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 90
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 90
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ser Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 91
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 91
Val Asp Ala Lys Tyr Ala Lys Glu Thr Glu Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 92
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 92
Val Asp Ala Lys Tyr Ala Lys Glu Ala Lys Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 93
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 93
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ser Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 94
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 94
Val Asp Ala Lys Tyr Ala Lys Glu Lys Glu Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 95
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 95
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asp Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 96
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 96
Val Asp Ala Lys Tyr Ala Lys Glu Glu Lys Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 97
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 97
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Trp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 98
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 98
Val Asp Ala Lys Tyr Ala Lys Glu Thr Lys Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 99
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 99
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 100
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 100
Val Asp Ala Lys Tyr Ala Lys Glu Trp Ala Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 101
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 101
Val Asp Ala Lys Tyr Ala Lys Glu Ser Gln Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 102
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 102
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ser Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 103
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 103
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 104
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 104
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 105
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 105
Val Asp Ala Lys Tyr Ala Lys Glu Gln Asp Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 106
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 106
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ala Ser Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 107
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 107
Val Asp Ala Lys Tyr Ala Lys Glu Val Ala Lys Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 108
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 108
Val Asp Ala Lys Tyr Ala Lys Glu Val Gln Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 109
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 109
Val Asp Ala Lys Tyr Ala Lys Glu Ser Tyr Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 110
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 110
Val Asp Ala Lys Tyr Ala Lys Glu Thr Ala Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 111
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 111
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 112
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 112
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 113
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 113
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Val Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 114
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 114
Val Asp Ala Lys Tyr Ala Lys Glu Ile Gln Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 115
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 115
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ala Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 116
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 116
Val Asp Ala Lys Tyr Ala Lys Glu Trp Met Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 117
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 117
Val Asp Ala Lys Tyr Ala Lys Glu Arg Glu Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 118
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 118
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 119
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 119
Val Asp Ala Lys Tyr Ala Lys Glu His Asn Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 120
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 120
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 121
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 121
Val Asp Ala Lys Tyr Ala Lys Glu Arg Ala Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 122
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 122
Val Asp Ala Lys Tyr Ala Lys Glu Ser Glu Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Trp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 123
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 123
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Arg Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 124
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 124
Val Asp Ala Lys Tyr Ala Lys Glu Thr Ala Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 125
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 125
Val Asp Ala Lys Tyr Ala Lys Glu Trp Tyr Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 126
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 126
Val Asp Ala Lys Tyr Ala Lys Glu Glu Gln Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 127
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 127
Val Asp Ala Lys Tyr Ala Lys Glu His Asp Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 128
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 128
Val Asp Ala Lys Tyr Ala Lys Glu Trp Tyr Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 129
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 129
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 130
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 130
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Thr Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 131
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 131
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 132
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 132
Val Asp Ala Lys Tyr Ala Lys Glu Asn Asp Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 133
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 133
Val Asp Ala Lys Tyr Ala Lys Glu Ser Glu Ile Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 134
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 134
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ala Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 135
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 135
Val Asp Ala Lys Tyr Ala Lys Glu Thr Glu Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 136
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 136
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Asp Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 137
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 137
Val Asp Ala Lys Tyr Ala Lys Glu His Leu Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 138
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 138
Val Asp Ala Lys Tyr Ala Lys Glu Trp Leu Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 139
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 139
Val Asp Ala Lys Tyr Ala Lys Glu Asn Ala Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 140
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 140
Val Asp Ala Lys Tyr Ala Lys Glu Ala Glu Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 141
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 141
Val Asp Ala Lys Tyr Ala Lys Glu Val Thr Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 142
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 142
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asp Ser Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 143
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 143
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asn Leu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 144
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 144
Val Asp Ala Lys Tyr Ala Lys Glu Ser Glu Val Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 145
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 145
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ala Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 146
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 146
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Ser Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asp
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 147
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 147
Val Asp Ala Lys Tyr Ala Lys Glu Thr Asn Asn Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Ile
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 148
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 148
Val Asp Ala Lys Tyr Ala Lys Glu Val Glu Phe Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 149
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 149
Val Asp Ala Lys Tyr Ala Lys Glu Val Glu Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu His Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 150
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 150
Val Asp Ala Lys Tyr Ala Lys Glu Val Leu Lys Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Thr
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 151
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 151
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ala Asn Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 152
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 152
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Met Lys Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 153
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 153
Val Asp Ala Lys Tyr Ala Lys Glu His Ala Asn Ala Gln His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 154
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 154
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Ile Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Trp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 155
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 155
Val Asp Ala Lys Tyr Ala Lys Glu Glu Val Phe Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 156
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 156
Val Asp Ala Lys Tyr Ala Lys Glu Phe Asn Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 157
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 157
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Val Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 158
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 158
Val Asp Ala Lys Tyr Ala Lys Glu Trp Ser Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 159
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 159
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asp Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 160
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 160
Val Asp Ala Lys Tyr Ala Lys Glu Arg His Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 161
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 161
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Asp Ala Ile His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 162
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 162
Val Asp Ala Lys Tyr Ala Lys Glu Trp Glu Thr Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 163
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 163
Val Asp Ala Lys Tyr Ala Lys Glu Arg Tyr Trp Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 164
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 164
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asp Trp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 165
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 165
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ser Lys Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 166
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 166
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Ala Ala Gln His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 167
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 167
Val Asp Ala Lys Tyr Ala Lys Glu His Glu Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Val Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 168
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 168
Val Asp Ala Lys Tyr Ala Lys Glu Ala Glu Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 169
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 169
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Tyr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 170
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 170
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Ser Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 171
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 171
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ala Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 172
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 172
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Arg Val Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 173
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 173
Val Asp Ala Lys Tyr Ala Lys Glu Val Val Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 174
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 174
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ala Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 175
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 175
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Ser Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 176
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 176
Val Asp Ala Lys Tyr Ala Lys Glu Gln Lys Glu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 177
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 177
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Ile Ala Gly Lys Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 178
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 178
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Lys Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 179
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 179
Val Asp Ala Lys Tyr Ala Lys Glu Ser Val Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Trp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 180
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 180
Val Asp Ala Lys Tyr Ala Lys Glu Ile Gln Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 181
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 181
Val Asp Ala Lys Tyr Ala Lys Glu Ile Thr Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 182
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 182
Val Asp Ala Lys Tyr Ala Lys Glu Gln Asp Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 183
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 183
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 184
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 184
Val Asp Ala Lys Tyr Ala Lys Glu Asn Gln Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 185
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 185
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 186
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 186
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ala Asn Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Tyr
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 187
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 187
Val Asp Ala Lys Tyr Ala Lys Glu Trp Gln Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 188
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 188
Val Asp Ala Lys Tyr Ala Lys Glu Arg Lys Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 189
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 189
Val Asp Ala Lys Tyr Ala Lys Glu Ile Thr Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 190
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 190
Val Asp Ala Lys Tyr Ala Lys Glu Phe Ile Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 191
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 191
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asn Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 192
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 192
Val Asp Ala Lys Tyr Ala Lys Glu Lys Phe Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 193
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 193
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Ser Ala Gly Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 194
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 194
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ser Val Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 195
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 195
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Leu Ala Gly Arg Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 196
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 196
Val Asp Ala Lys Tyr Ala Lys Glu Gln Glu Arg Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 197
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 197
Val Asp Ala Lys Tyr Ala Lys Glu Ile Trp Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 198
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 198
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asn Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Glu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 199
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 199
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 200
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 200
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asn Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 201
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 201
Val Asp Ala Lys Tyr Ala Lys Glu Leu Val Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 202
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 202
Val Asp Ala Lys Tyr Ala Lys Glu Leu Thr Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 203
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 203
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asn Ala Ala Ala Arg Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 204
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 204
Val Asp Ala Lys Tyr Ala Lys Glu Ile Leu His Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 205
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 205
Val Asp Ala Lys Tyr Ala Lys Glu Val Leu Thr Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 206
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 206
Val Asp Ala Lys Tyr Ala Lys Glu Asn Ser Lys Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 207
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 207
Val Asp Ala Lys Tyr Ala Lys Glu Val Met Thr Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 208
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 208
Val Asp Ala Lys Tyr Ala Lys Glu Ala Arg Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 209
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 209
Val Asp Ala Lys Tyr Ala Lys Glu Arg Ser Lys Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 210
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 210
Val Asp Ala Lys Tyr Ala Lys Glu Ile Tyr Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 211
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 211
Val Asp Ala Lys Tyr Ala Lys Glu Val Gln Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 212
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 212
Val Asp Ala Lys Tyr Ala Lys Glu Thr Leu Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 213
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 213
Val Asp Ala Lys Tyr Ala Lys Glu Gln Met Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 214
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 214
Val Asp Ala Lys Tyr Ala Lys Glu Asn Lys Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 215
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 215
Val Asp Ala Lys Tyr Ala Lys Glu Thr Glu Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Thr Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 216
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 216
Val Asp Ala Lys Tyr Ala Lys Glu Thr Val Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 217
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 217
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ala Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 218
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 218
Val Asp Ala Lys Tyr Ala Lys Glu Val Met Asp Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 219
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 219
Val Asp Ala Lys Tyr Ala Lys Glu Thr Asp Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 220
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 220
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Ile Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 221
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 221
Val Asp Ala Lys Tyr Ala Lys Glu Trp Lys Asp Ala Ala Gln Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 222
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 222
Val Asp Ala Lys Tyr Ala Lys Glu Arg Asp Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 223
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 223
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ala Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 224
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 224
Val Asp Ala Lys Tyr Ala Lys Glu Ser Val Lys Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 225
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 225
Val Asp Ala Lys Tyr Ala Lys Glu Asn Glu Arg Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 226
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 226
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Lys Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 227
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 227
Val Asp Ala Lys Tyr Ala Lys Glu Val Arg Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 228
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 228
Val Asp Ala Lys Tyr Ala Lys Glu Asp Lys Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 229
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 229
Val Asp Ala Lys Tyr Ala Lys Glu Ser Glu Lys Ala Gly Lys Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 230
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 230
Val Asp Ala Lys Tyr Ala Lys Glu Ile Asn Arg Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 231
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 231
Val Asp Ala Lys Tyr Ala Lys Glu Thr Gln Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 232
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 232
Val Asp Ala Lys Tyr Ala Lys Glu Asn Gln Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 233
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 233
Val Asp Ala Lys Tyr Ala Lys Glu Ala Lys Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 234
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 234
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 235
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 235
Val Asp Ala Lys Tyr Ala Lys Glu Val Gln Tyr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 236
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 236
Val Asp Ala Lys Tyr Ala Lys Glu Leu Arg Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 237
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 237
Val Asp Ala Lys Tyr Ala Lys Glu Gln Arg Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 238
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 238
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ser Glu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 239
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 239
Val Asp Ala Lys Tyr Ala Lys Glu Ser Val Ile Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 240
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 240
Val Asp Ala Lys Tyr Ala Lys Glu Ile Leu Arg Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 241
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 241
Val Asp Ala Lys Tyr Ala Lys Glu Ser Lys Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 242
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 242
Val Asp Ala Lys Tyr Ala Lys Glu Leu Ala Glu Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Phe Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 243
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 243
Val Asp Ala Lys Tyr Ala Lys Glu Ala Thr Thr Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 244
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 244
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 245
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 245
Val Asp Ala Lys Tyr Ala Lys Glu Ala Lys Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 246
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 246
Val Asp Ala Lys Tyr Ala Lys Glu Arg Leu Glu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 247
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 247
Val Asp Ala Lys Tyr Ala Lys Glu Gln Met Glu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 248
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 248
Val Asp Ala Lys Tyr Ala Lys Glu Val Lys Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 249
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 249
Val Asp Ala Lys Tyr Ala Lys Glu Ser Phe Glu Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 250
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 250
Val Asp Ala Lys Tyr Ala Lys Glu Ile Lys Ser Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 251
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 251
Val Asp Ala Lys Tyr Ala Lys Glu Ile Lys Asn Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 252
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 252
Val Asp Ala Lys Tyr Ala Lys Glu Leu Gln Glu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 253
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 253
Val Asp Ala Lys Tyr Ala Lys Glu Arg Gln Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 254
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 254
Val Asp Ala Lys Tyr Ala Lys Glu Val Leu Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 255
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 255
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asn Val Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 256
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 256
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asp Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 257
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 257
Val Asp Ala Lys Tyr Ala Lys Glu Thr Ala Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 258
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 258
Val Asp Ala Lys Tyr Ala Lys Glu Trp Lys Gln Ala Ala Ser Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 259
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 259
Val Asp Ala Lys Tyr Ala Lys Glu Thr Ala Ser Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 260
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 260
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ile Val Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 261
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 261
Val Asp Ala Lys Tyr Ala Lys Glu Ile Lys Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 262
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 262
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ala Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 263
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 263
Val Asp Ala Lys Tyr Ala Lys Glu Leu Asn Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 264
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 264
Val Asp Ala Lys Tyr Ala Lys Glu Val Lys Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 265
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 265
Val Asp Ala Lys Tyr Ala Lys Glu Ser Arg Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 266
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 266
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ile Thr Ala Ser Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 267
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 267
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 268
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 268
Val Asp Ala Lys Tyr Ala Lys Glu Val Tyr Ala Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 269
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 269
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ser Arg Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 270
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 270
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Val Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 271
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 271
Val Asp Ala Lys Tyr Ala Lys Glu His Ile Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 272
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 272
Val Asp Ala Lys Tyr Ala Lys Glu Ile Leu Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 273
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 273
Val Asp Ala Lys Tyr Ala Lys Glu Asn Ser Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 274
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 274
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Arg Val Ala Ala Lys Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 275
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 275
Val Asp Ala Lys Tyr Ala Lys Glu Ile Tyr Asn Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 276
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 276
Val Asp Ala Lys Tyr Ala Lys Glu Ser Asn Glu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 277
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 277
Val Asp Ala Lys Tyr Ala Lys Glu Ser Gln Leu Ala Ala Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 278
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 278
Val Asp Ala Lys Tyr Ala Lys Glu Leu Lys Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 279
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 279
Val Asp Ala Lys Tyr Ala Lys Glu Thr Arg Val Ala Ser Val Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 280
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 280
Val Asp Ala Lys Tyr Ala Lys Glu Leu Arg Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 281
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 281
Val Asp Ala Lys Tyr Ala Lys Glu Lys Thr Tyr Ala His Phe Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Ser
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 282
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 282
Val Asp Ala Lys Tyr Ala Lys Glu Glu Ala Gln Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 283
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 283
Val Asp Ala Lys Tyr Ala Lys Glu Ile Thr Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 284
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 284
Val Asp Ala Lys Tyr Ala Lys Glu Val Lys Thr Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 285
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 285
Val Asp Ala Lys Tyr Ala Lys Glu Thr Lys Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 286
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 286
Val Asp Ala Lys Tyr Ala Lys Glu Asp Leu Val Ala Gln His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 287
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 287
Val Asp Ala Lys Tyr Ala Lys Glu Thr Gln Thr Ala Phe Asn Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Ala Ala Phe Ile Leu
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 288
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 288
Val Asp Ala Lys Tyr Ala Lys Glu Ile Lys Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 289
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 289
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Lys Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 290
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 290
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ala Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 291
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 291
Val Asp Ala Lys Tyr Ala Lys Glu Gln Glu Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 292
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 292
Val Asp Ala Lys Tyr Ala Lys Glu Trp Phe Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 293
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 293
Val Asp Ala Lys Tyr Ala Lys Glu Ile Ile Gln Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 294
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 294
Val Asp Ala Lys Tyr Ala Lys Glu Leu Thr Asn Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 295
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 295
Val Asp Ala Lys Tyr Ala Lys Glu Ile Gln Leu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 296
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 296
Val Asp Ala Lys Tyr Ala Lys Glu Ile His Asp Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 297
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 297
Val Asp Ala Lys Tyr Ala Lys Glu Val Lys Ile Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 298
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 298
Val Asp Ala Lys Tyr Ala Lys Glu Gln His Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 299
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 299
Val Asp Ala Lys Tyr Ala Lys Glu Val Phe Ala Ala Ser Ala Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 300
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 300
Val Asp Ala Lys Tyr Ala Lys Glu Thr Asp Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 301
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 301
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asn Phe Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 302
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 302
Val Asp Ala Lys Tyr Ala Lys Glu Phe Glu Thr Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Thr
20 25 30
Lys Leu Trp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 303
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 303
Val Asp Ala Lys Tyr Ala Lys Glu Val Asn Leu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 304
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 304
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Thr Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Tyr
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 305
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 305
Val Asp Ala Lys Tyr Ala Lys Glu Phe Val Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 306
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 306
Val Asp Ala Lys Tyr Ala Lys Glu Asp His Lys Ala Glu His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 307
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 307
Val Asp Ala Lys Tyr Ala Lys Glu Thr Val Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 308
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 308
Val Asp Ala Lys Tyr Ala Lys Glu Ser Gln Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Thr
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 309
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 309
Val Asp Ala Lys Tyr Ala Lys Glu Trp Ser Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 310
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 310
Val Asp Ala Lys Tyr Ala Lys Glu Val Ala Val Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 311
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 311
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ala Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 312
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 312
Val Asp Ala Lys Tyr Ala Lys Glu Ala Val Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 313
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 313
Val Asp Ala Lys Tyr Ala Lys Glu Phe Gln Ile Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 314
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 314
Val Asp Ala Lys Tyr Ala Lys Glu Leu Met Val Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 315
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 315
Val Asp Ala Lys Tyr Ala Lys Glu Tyr Asp Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Leu
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 316
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 316
Val Asp Ala Lys Tyr Ala Lys Glu Val Leu Glu Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 317
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 317
Val Asp Ala Lys Tyr Ala Lys Glu Ser Ile Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 318
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 318
Val Asp Ala Lys Tyr Ala Lys Glu Val Ala Glu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Ser
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 319
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 319
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ala Lys Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 320
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 320
Val Asp Ala Lys Tyr Ala Lys Glu Arg Asp Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 321
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 321
Val Asp Ala Lys Tyr Ala Lys Glu Ala Lys Asp Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 322
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 322
Val Asp Ala Lys Tyr Ala Lys Glu Ala Ser Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 323
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 323
Val Asp Ala Lys Tyr Ala Lys Glu Trp Met Glu Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 324
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 324
Val Asp Ala Lys Tyr Ala Lys Glu Gln Lys Asn Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Glu
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 325
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 325
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Asn Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 326
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 326
Val Asp Ala Lys Tyr Ala Lys Glu Val Asn Arg Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Leu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 327
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 327
Val Asp Ala Lys Tyr Ala Lys Glu Arg Leu Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 328
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 328
Val Asp Ala Lys Tyr Ala Lys Glu Val Ser Ile Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 329
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 329
Val Asp Ala Lys Tyr Ala Lys Glu Lys Glu Val Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 330
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 330
Val Asp Ala Lys Tyr Ala Lys Glu Ser Glu Arg Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 331
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 331
Val Asp Ala Lys Tyr Ala Lys Glu Trp Asn Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 332
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 332
Val Asp Ala Lys Tyr Ala Lys Glu Asn Val Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 333
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 333
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 334
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 334
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Ser Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ile Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 335
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 335
Val Asp Ala Lys Tyr Ala Lys Glu Glu Gln Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 336
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 336
Val Asp Ala Lys Tyr Ala Lys Glu Phe Glu Leu Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 337
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 337
Val Asp Ala Lys Tyr Ala Lys Glu Ala Phe Val Ala Gln His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile Val
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 338
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 338
Val Asp Ala Lys Tyr Ala Lys Glu Ala Leu Lys Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Asn
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 339
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 339
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Arg Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Lys
20 25 30
Lys Leu Thr Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 340
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 340
Val Asp Ala Lys Tyr Ala Lys Glu Val Glu Trp Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 341
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 341
Val Asp Ala Lys Tyr Ala Lys Glu Lys Ala Ser Ala Gln His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 342
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 342
Val Asp Ala Lys Tyr Ala Lys Glu Thr Glu Ile Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 343
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 343
Val Asp Ala Lys Tyr Ala Lys Glu Val Asn Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Thr Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 344
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 344
Val Asp Ala Lys Tyr Ala Lys Glu Ala Glu Glu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 345
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 345
Val Asp Ala Lys Tyr Ala Lys Glu Thr Asp Arg Ala Lys His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 346
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 346
Val Asp Ala Lys Tyr Ala Lys Glu Phe Ala Gln Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Lys Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 347
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 347
Val Asp Ala Lys Tyr Ala Lys Glu Thr Asp Glu Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Arg
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 348
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 348
Val Asp Ala Lys Tyr Ala Lys Glu Asn Ala Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 349
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 349
Val Asp Ala Lys Tyr Ala Lys Glu Ser Thr Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Gln Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 350
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 350
Val Asp Ala Lys Tyr Ala Lys Glu Gln Ala Leu Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asn Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 351
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 351
Val Asp Ala Lys Tyr Ala Lys Glu Ala His Ala Ala Ser His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Asp Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 352
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 352
Val Asp Ala Lys Tyr Ala Lys Glu Val Asp Asn Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile Gln
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 353
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 353
Val Asp Ala Lys Tyr Ala Lys Glu Ala Gly Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Trp Asp Gln Arg Val Ala Phe Ile Trp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 354
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 354
Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 355
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 355
Ala Glu Ala Lys Tyr Ala Lys Glu Phe Glu Ser Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Tyr Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ser Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 356
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 356
Ala Glu Ala Lys Tyr Ala Lys Glu Trp Met Arg Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Glu Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 357
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 357
Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 358
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 358
Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 359
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 359
Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Arg Gln Pro Glu Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 360
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 360
Val Asp Ala Lys Tyr Ala Lys Glu Trp Thr Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 361
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 361
Val Asp Ala Lys Tyr Ala Lys Glu Leu Glu Lys Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 362
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 362
Val Asp Ala Lys Tyr Ala Lys Glu Trp Gln Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 363
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 363
Val Asp Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 364
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 364
Val Asp Ala Lys Tyr Ala Lys Glu Ile Glu Asp Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 365
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 365
Ala Glu Ala Lys Phe Ala Lys Glu Trp Thr Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 366
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 366
Ala Glu Ala Lys Phe Ala Lys Glu Trp Gln Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 367
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 367
Ala Glu Ala Lys Phe Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
50 55
<210> 368
<211> 133
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding dimer
<400> 368
Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Gly Thr Gly Gly Gly Gly
50 55 60
Ser Pro Arg Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala
65 70 75 80
His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala
85 90 95
Phe Ile His Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu
100 105 110
Ser Glu Ala Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Leu Glu His
115 120 125
His His His His His
130
<210> 369
<211> 141
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding dimer
<400> 369
Ala Glu Ala Lys Tyr Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Ala Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Gly Thr Gly Gly Gly Gly
50 55 60
Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Ala Glu Ala Lys Tyr
65 70 75 80
Ala Lys Glu Gln Asp Ala Ala Ala His Glu Ile Arg Trp Leu Pro Asn
85 90 95
Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His Lys Leu Ala Asp Asp
100 105 110
Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala Lys Lys Leu Asn Asp
115 120 125
Ser Gln Ala Pro Lys Leu Glu His His His His His His
130 135 140
<210> 370
<211> 144
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding dimer
<400> 370
Gly Ser Ser His His His His His His Leu Gln Ala Glu Ala Lys Tyr
1 5 10 15
Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile Arg Trp Leu Pro Asn
20 25 30
Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His Lys Leu Arg Asp Asp
35 40 45
Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala Lys Lys Leu Asn Asp
50 55 60
Ser Gln Ala Pro Lys Gly Thr Gly Gly Gly Gly Ser Gly Gly Gly Gly
65 70 75 80
Ser Gly Gly Gly Gly Ser Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp
85 90 95
Ala Ala Gly His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln
100 105 110
Arg Val Ala Phe Ile His Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser
115 120 125
Glu Leu Leu Ser Glu Ala Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
130 135 140
<210> 371
<211> 201
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding dimer
<400> 371
Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Gly Ala Pro Gly Gly Gly
50 55 60
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ser Leu Ala
65 70 75 80
Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser
85 90 95
Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val
100 105 110
Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro Gly Thr Gly Gly
115 120 125
Gly Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Arg Ala
130 135 140
Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile Arg
145 150 155 160
Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His Lys
165 170 175
Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala Lys
180 185 190
Lys Leu Asn Asp Ser Gln Ala Pro Lys
195 200
<210> 372
<211> 201
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding dimer
<400> 372
Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Gly Ala Pro Gly Gly Gly
50 55 60
Gly Ser Gly Gly Gly Gly Ser Gly Gly Gly Gly Ser Thr Ser Ala Glu
65 70 75 80
Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile Arg Trp
85 90 95
Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His Lys Leu
100 105 110
Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala Lys Lys
115 120 125
Leu Asn Asp Ser Gln Ala Pro Lys Gly Thr Gly Gly Gly Gly Ser Gly
130 135 140
Gly Gly Gly Ser Gly Gly Gly Gly Ser Pro Arg Leu Ala Glu Ala Lys
145 150 155 160
Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser Asp Phe Tyr
165 170 175
Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val Glu Ala Leu
180 185 190
Lys Asp Ala Ile Leu Ala Ala Leu Pro
195 200
<210> 373
<211> 173
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 373
Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys Ala Ser Gly Ser Leu Ala
50 55 60
Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser
65 70 75 80
Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val
85 90 95
Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro Gly Thr Gly Gly
100 105 110
Gly Gly Ser Ala Glu Ala Lys Tyr Ala Lys Glu Ala Asp Ala Ala Gly
115 120 125
His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala
130 135 140
Phe Ile His Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu
145 150 155 160
Ser Glu Ala Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
165 170
<210> 374
<211> 173
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 374
Ala Glu Ala Lys Phe Ala Lys Glu Trp Thr Asp Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys Ala Ser Gly Ser Leu Ala
50 55 60
Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser
65 70 75 80
Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val
85 90 95
Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro Gly Thr Gly Gly
100 105 110
Gly Gly Ser Ala Glu Ala Lys Phe Ala Lys Glu Trp Thr Asp Ala Ala
115 120 125
His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala
130 135 140
Phe Ile His Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu
145 150 155 160
Ser Glu Ala Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
165 170
<210> 375
<211> 173
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 375
Ala Glu Ala Lys Phe Ala Lys Glu Trp Gln Gln Ala Ala His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys Ala Ser Gly Ser Leu Ala
50 55 60
Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser
65 70 75 80
Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val
85 90 95
Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro Gly Thr Gly Gly
100 105 110
Gly Gly Ser Ala Glu Ala Lys Phe Ala Lys Glu Trp Gln Gln Ala Ala
115 120 125
His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala
130 135 140
Phe Ile His Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu
145 150 155 160
Ser Glu Ala Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
165 170
<210> 376
<211> 173
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered FcRn binding polypeptide
<400> 376
Ala Glu Ala Lys Phe Ala Lys Glu Ala Asp Ala Ala Gly His Glu Ile
1 5 10 15
Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala Phe Ile His
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys Ala Ser Gly Ser Leu Ala
50 55 60
Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly Val Ser
65 70 75 80
Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu Gly Val
85 90 95
Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro Gly Thr Gly Gly
100 105 110
Gly Gly Ser Ala Glu Ala Lys Phe Ala Lys Glu Ala Asp Ala Ala Gly
115 120 125
His Glu Ile Arg Trp Leu Pro Asn Leu Thr Phe Asp Gln Arg Val Ala
130 135 140
Phe Ile His Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu
145 150 155 160
Ser Glu Ala Lys Lys Leu Ser Glu Ser Gln Ala Pro Lys
165 170
<210> 377
<211> 46
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered albumin binding polypeptide
<400> 377
Leu Ala Glu Ala Lys Glu Ala Ala Asn Ala Glu Leu Asp Ser Tyr Gly
1 5 10 15
Val Ser Asp Phe Tyr Lys Arg Leu Ile Asp Lys Ala Lys Thr Val Glu
20 25 30
Gly Val Glu Ala Leu Lys Asp Ala Ile Leu Ala Ala Leu Pro
35 40 45
<210> 378
<211> 58
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> Engineered binding polypeptide
<400> 378
Ala Glu Ala Lys Tyr Ala Lys Glu Leu Gly Trp Ala Thr Trp Glu Ile
1 5 10 15
Phe Asn Leu Pro Asn Leu Thr Gly Val Gln Val Lys Ala Phe Ile Asp
20 25 30
Lys Leu Arg Asp Asp Pro Ser Gln Ser Ser Glu Leu Leu Ser Glu Ala
35 40 45
Lys Lys Leu Asn Asp Ser Gln Ala Pro Lys
50 55
<210> 379
<211> 267
<212> PRT
<213> HOMO SAPIENS
<400> 379
Ala Glu Ser His Leu Ser Leu Leu Tyr His Leu Thr Ala Val Ser Ser
1 5 10 15
Pro Ala Pro Gly Thr Pro Ala Phe Trp Val Ser Gly Trp Leu Gly Pro
20 25 30
Gln Gln Tyr Leu Ser Tyr Asn Ser Leu Arg Gly Glu Ala Glu Pro Cys
35 40 45
Gly Ala Trp Val Trp Glu Asn Gln Val Ser Trp Tyr Trp Glu Lys Glu
50 55 60
Thr Thr Asp Leu Arg Ile Lys Glu Lys Leu Phe Leu Glu Ala Phe Lys
65 70 75 80
Ala Leu Gly Gly Lys Gly Pro Tyr Thr Leu Gln Gly Leu Leu Gly Cys
85 90 95
Glu Leu Gly Pro Asp Asn Thr Ser Val Pro Thr Ala Lys Phe Ala Leu
100 105 110
Asn Gly Glu Glu Phe Met Asn Phe Asp Leu Lys Gln Gly Thr Trp Gly
115 120 125
Gly Asp Trp Pro Glu Ala Leu Ala Ile Ser Gln Arg Trp Gln Gln Gln
130 135 140
Asp Lys Ala Ala Asn Lys Glu Leu Thr Phe Leu Leu Phe Ser Cys Pro
145 150 155 160
His Arg Leu Arg Glu His Leu Glu Arg Gly Arg Gly Asn Leu Glu Trp
165 170 175
Lys Glu Pro Pro Ser Met Arg Leu Lys Ala Arg Pro Ser Ser Pro Gly
180 185 190
Phe Ser Val Leu Thr Cys Ser Ala Phe Ser Phe Tyr Pro Pro Glu Leu
195 200 205
Gln Leu Arg Phe Leu Arg Asn Gly Leu Ala Ala Gly Thr Gly Gln Gly
210 215 220
Asp Phe Gly Pro Asn Ser Asp Gly Ser Phe His Ala Ser Ser Ser Leu
225 230 235 240
Thr Val Lys Ser Gly Asp Glu His His Tyr Cys Cys Ile Val Gln His
245 250 255
Ala Gly Leu Ala Gln Pro Leu Arg Val Glu Leu
260 265
<210> 380
<211> 99
<212> PRT
<213> HOMO SAPIENS
<400> 380
Ile Gln Arg Thr Pro Lys Ile Gln Val Tyr Ser Arg His Pro Ala Glu
1 5 10 15
Asn Gly Lys Ser Asn Phe Leu Asn Cys Tyr Val Ser Gly Phe His Pro
20 25 30
Ser Asp Ile Glu Val Asp Leu Leu Lys Asn Gly Glu Arg Ile Glu Lys
35 40 45
Val Glu His Ser Asp Leu Ser Phe Ser Lys Asp Trp Ser Phe Tyr Leu
50 55 60
Leu Tyr Tyr Thr Glu Phe Thr Pro Thr Glu Lys Asp Glu Tyr Ala Cys
65 70 75 80
Arg Val Asn His Val Thr Leu Ser Gln Pro Lys Ile Val Lys Trp Asp
85 90 95
Arg Asp Met
<210> 381
<211> 99
<212> PRT
<213> MUS MUSCULUS
<400> 381
Ile Gln Lys Thr Pro Gln Ile Gln Val Tyr Ser Arg His Pro Pro Glu
1 5 10 15
Asn Gly Lys Pro Asn Ile Leu Asn Cys Tyr Val Thr Gln Phe His Pro
20 25 30
Pro His Ile Glu Ile Gln Met Leu Lys Asn Gly Lys Lys Ile Pro Lys
35 40 45
Val Glu Met Ser Asp Met Ser Phe Ser Lys Asp Trp Ser Phe Tyr Ile
50 55 60
Leu Ala His Thr Glu Phe Thr Pro Thr Glu Thr Asp Thr Tyr Ala Cys
65 70 75 80
Arg Val Lys His Ala Ser Met Ala Glu Pro Lys Thr Val Tyr Trp Asp
85 90 95
Arg Asp Met
<210> 382
<211> 365
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> hFcRn - eGFP fusion protein
<400> 382
Met Gly Val Pro Arg Pro Gln Pro Trp Ala Leu Gly Leu Leu Leu Phe
1 5 10 15
Leu Leu Pro Gly Ser Leu Gly Ala Glu Ser His Leu Ser Leu Leu Tyr
20 25 30
His Leu Thr Ala Val Ser Ser Pro Ala Pro Gly Thr Pro Ala Phe Trp
35 40 45
Val Ser Gly Trp Leu Gly Pro Gln Gln Tyr Leu Ser Tyr Asn Ser Leu
50 55 60
Arg Gly Glu Ala Glu Pro Cys Gly Ala Trp Val Trp Glu Asn Gln Val
65 70 75 80
Ser Trp Tyr Trp Glu Lys Glu Thr Thr Asp Leu Arg Ile Lys Glu Lys
85 90 95
Leu Phe Leu Glu Ala Phe Lys Ala Leu Gly Gly Lys Gly Pro Tyr Thr
100 105 110
Leu Gln Gly Leu Leu Gly Cys Glu Leu Gly Pro Asp Asn Thr Ser Val
115 120 125
Pro Thr Ala Lys Phe Ala Leu Asn Gly Glu Glu Phe Met Asn Phe Asp
130 135 140
Leu Lys Gln Gly Thr Trp Gly Gly Asp Trp Pro Glu Ala Leu Ala Ile
145 150 155 160
Ser Gln Arg Trp Gln Gln Gln Asp Lys Ala Ala Asn Lys Glu Leu Thr
165 170 175
Phe Leu Leu Phe Ser Cys Pro His Arg Leu Arg Glu His Leu Glu Arg
180 185 190
Gly Arg Gly Asn Leu Glu Trp Lys Glu Pro Pro Ser Met Arg Leu Lys
195 200 205
Ala Arg Pro Ser Ser Pro Gly Phe Ser Val Leu Thr Cys Ser Ala Phe
210 215 220
Ser Phe Tyr Pro Pro Glu Leu Gln Leu Arg Phe Leu Arg Asn Gly Leu
225 230 235 240
Ala Ala Gly Thr Gly Gln Gly Asp Phe Gly Pro Asn Ser Asp Gly Ser
245 250 255
Phe His Ala Ser Ser Ser Leu Thr Val Lys Ser Gly Asp Glu His His
260 265 270
Tyr Cys Cys Ile Val Gln His Ala Gly Leu Ala Gln Pro Leu Arg Val
275 280 285
Glu Leu Glu Ser Pro Ala Lys Ser Ser Val Leu Val Val Gly Ile Val
290 295 300
Ile Gly Val Leu Leu Leu Thr Ala Ala Ala Val Gly Gly Ala Leu Leu
305 310 315 320
Trp Arg Arg Met Arg Ser Gly Leu Pro Ala Pro Trp Ile Ser Leu Arg
325 330 335
Gly Asp Asp Thr Gly Val Leu Leu Pro Thr Pro Gly Glu Ala Gln Asp
340 345 350
Ala Asp Leu Lys Asp Val Asn Val Ile Pro Ala Thr Ala
355 360 365
<210> 383
<211> 369
<212> PRT
<213> ARTIFICIAL SEQUENCE
<220>
<223> mFcRn - eGFP fusion protein
<400> 383
Met Gly Met Pro Leu Pro Trp Ala Leu Ser Leu Leu Leu Val Leu Leu
1 5 10 15
Pro Gln Thr Trp Gly Ser Glu Thr Arg Pro Pro Leu Met Tyr His Leu
20 25 30
Thr Ala Val Ser Asn Pro Ser Thr Gly Leu Pro Ser Phe Trp Ala Thr
35 40 45
Gly Trp Leu Gly Pro Gln Gln Tyr Leu Thr Tyr Asn Ser Leu Arg Gln
50 55 60
Glu Ala Asp Pro Cys Gly Ala Trp Met Trp Glu Asn Gln Val Ser Trp
65 70 75 80
Tyr Trp Glu Lys Glu Thr Thr Asp Leu Lys Ser Lys Glu Gln Leu Phe
85 90 95
Leu Glu Ala Leu Lys Thr Leu Glu Lys Ile Leu Asn Gly Gln Lys Arg
100 105 110
Gly Thr Tyr Thr Leu Gln Gly Leu Leu Gly Cys Glu Leu Ala Ser Asp
115 120 125
Asn Ser Ser Val Pro Thr Ala Val Phe Ala Leu Asn Gly Glu Glu Phe
130 135 140
Met Lys Phe Asn Pro Arg Ile Gly Asn Trp Thr Gly Glu Trp Pro Glu
145 150 155 160
Thr Glu Ile Val Ala Asn Leu Trp Met Lys Gln Pro Asp Ala Ala Arg
165 170 175
Lys Glu Ser Glu Phe Leu Leu Asn Ser Cys Pro Glu Arg Leu Leu Gly
180 185 190
His Leu Glu Arg Gly Arg Arg Asn Leu Glu Trp Lys Glu Pro Pro Ser
195 200 205
Met Arg Leu Lys Ala Arg Pro Gly Asn Ser Gly Ser Ser Val Leu Thr
210 215 220
Cys Ala Ala Phe Ser Phe Tyr Pro Pro Glu Leu Lys Phe Arg Phe Leu
225 230 235 240
Arg Asn Gly Leu Ala Ser Gly Ser Gly Asn Cys Ser Thr Gly Pro Asn
245 250 255
Gly Asp Gly Ser Phe His Ala Trp Ser Leu Leu Glu Val Lys Arg Gly
260 265 270
Asp Glu His His Tyr Gln Cys Gln Val Glu His Glu Gly Leu Ala Gln
275 280 285
Pro Leu Thr Val Asp Leu Asp Ser Ser Ala Arg Ser Ser Val Pro Val
290 295 300
Val Gly Ile Val Leu Gly Leu Leu Leu Val Val Val Ala Ile Ala Gly
305 310 315 320
Gly Val Leu Leu Trp Gly Arg Met Arg Ser Gly Leu Pro Ala Pro Trp
325 330 335
Leu Ser Leu Ser Gly Asp Asp Ser Gly Asp Leu Leu Pro Gly Gly Asn
340 345 350
Leu Pro Pro Glu Ala Glu Pro Gln Gly Ala Asn Ala Phe Pro Ala Thr
355 360 365
Ser
<210> 384
<211> 273
<212> PRT
<213> MUS MUSCULUS
<400> 384
Ser Glu Thr Arg Pro Pro Leu Met Tyr His Leu Thr Ala Val Ser Asn
1 5 10 15
Pro Ser Thr Gly Leu Pro Ser Phe Trp Ala Thr Gly Trp Leu Gly Pro
20 25 30
Gln Gln Tyr Leu Thr Tyr Asn Ser Leu Arg Gln Glu Ala Asp Pro Cys
35 40 45
Gly Ala Trp Met Trp Glu Asn Gln Val Ser Trp Tyr Trp Glu Lys Glu
50 55 60
Thr Thr Asp Leu Lys Ser Lys Glu Gln Leu Phe Leu Glu Ala Leu Lys
65 70 75 80
Thr Leu Glu Lys Ile Leu Asn Gly Gln Lys Arg Gly Thr Tyr Thr Leu
85 90 95
Gln Gly Leu Leu Gly Cys Glu Leu Ala Ser Asp Asn Ser Ser Val Pro
100 105 110
Thr Ala Val Phe Ala Leu Asn Gly Glu Glu Phe Met Lys Phe Asn Pro
115 120 125
Arg Ile Gly Asn Trp Thr Gly Glu Trp Pro Glu Thr Glu Ile Val Ala
130 135 140
Asn Leu Trp Met Lys Gln Pro Asp Ala Ala Arg Lys Glu Ser Glu Phe
145 150 155 160
Leu Leu Asn Ser Cys Pro Glu Arg Leu Leu Gly His Leu Glu Arg Gly
165 170 175
Arg Arg Asn Leu Glu Trp Lys Glu Pro Pro Ser Met Arg Leu Lys Ala
180 185 190
Arg Pro Gly Asn Ser Gly Ser Ser Val Leu Thr Cys Ala Ala Phe Ser
195 200 205
Phe Tyr Pro Pro Glu Leu Lys Phe Arg Phe Leu Arg Asn Gly Leu Ala
210 215 220
Ser Gly Ser Gly Asn Cys Ser Thr Gly Pro Asn Gly Asp Gly Ser Phe
225 230 235 240
His Ala Trp Ser Leu Leu Glu Val Lys Arg Gly Asp Glu His His Tyr
245 250 255
Gln Cys Gln Val Glu His Glu Gly Leu Ala Gln Pro Leu Thr Val Asp
260 265 270
Leu
<210> 385
<211> 24
<212> DNA
<213> ARTIFICIAL SEQUENCE
<220>
<223> PCR primer
<400> 385
tgcttccggc tcgtatgttg tgtg 24
<210> 386
<211> 21
<212> DNA
<213> ARTIFICIAL SEQUENCE
<220>
<223> PCR primer
<400> 386
cggaaccaga gccaccaccg g 21
<210> 387
<211> 21
<212> DNA
<213> ARTIFICIAL SEQUENCE
<220>
<223> Sequencing primer
<400> 387
cggaaccaga gccaccaccg g 21
<210> 388
<211> 147
<212> DNA
<213> ARTIFICIAL SEQUENCE
<220>
<223> Library polynucleotide
<220>
<221> misc_feature
<222> (39)..(47)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (51)..(56)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (63)..(68)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (84)..(89)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (93)..(98)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (108)..(110)
<223> n is a, c, g, or t
<220>
<221> misc_feature
<222> (117)..(119)
<223> n is a, c, g, or t
<400> 388
aaataaatct cgaggtagat gccaaatacg ccaaagaann nnnnnnngcg nnnnnngaga 60
tcnnnnnntt acctaactta accnnnnnnc aannnnnngc cttcatcnnn aaattannng 120
atgacccaag ccagagctca ttattta 147
<---