Результат интеллектуальной деятельности: Способ идентификации генов клинически значимых семейств β-лактамаз у грамотрицательных микроорганизмов
Вид РИД
Изобретение
Изобретение относится к медицине, а точнее - к медицинской диагностике, и применяется для обнаружения генов, ответственных за синтез следующих разрушающих антибиотики β-лактамаз, имеющих важное клиническое значение: SHV, СТХ-М, VIM, IMP, NDM, ОХА, KPC, GES, GIM, SPM у актуальных для медицины грамотрицательных микроорганизмов, в том числе у Acinetobacter baumannii, Pseudomonas aeruginosa, Klebsiella pneumoniae, Burkholderia spp., Enterobacter spp., Stenotrophomonas maltophilia, Elizabethkingia meningoseptica, Serratia marcescens.
Известно, что для обнаружения генов, ответственных за синтез клинически значимых семейств β-лактамаз у грамотрицательных микроорганизмов, существует ряд способов.
Известен способ определения нуклеотидных последовательностей генов β-лактамаз грамотрицательных бактерий, который выполняется методом секвенирования по Сэнгеру (Lee СС, Lui G, Ip М, Ling TK, Lee N. Frequent detection of plasmid-mediated quinolone resistance (qnr) genes in multidrug-resistant Enterobacteriaceae blood isolates, Hong Kong. Eur J Clin Microbiol Infect Dis. 2012 Nov; 31(11):3183-3189). В процессе секвенирования одна из цепочек анализируемой ДНК выступает в качестве матрицы для синтеза комплементарной цепочки ферментом ДНК-полимеразой. В полимеразную цепную реакцию добавляются дидезоксинуклеотиды с флуоресцентными метками с разными длинами волн испускания. У дидезоксирибонуклеотидов отсутствует 3'-гидроксильная группа, поэтому после их включения в цепь дальнейший синтез обрывается. В результате образуется набор фрагментов ДНК разной длины, которые заканчиваются тем или иным нуклеотидом. Реакционную смесь разделяют капиллярным электрофорезом в растворе, фрагменты ДНК, выходящие из капиллярной колонки, регистрируются детектором флуоресценции. Результаты анализируют с помощью компьютера и представляют в виде последовательности разноцветных пиков, соответствующих четырем нуклеотидам. Главный недостаток секвенирования генов β-лактамаз заключается в длительности его выполнения, - он реализуется в течение нескольких суток, что бывает недопустимым в тяжелых случаях заболеваний, когда требуется срочная диагностика по жизненным показаниям. Кроме этого способ секвенирование генов требует использования специализированного дорогостоящего оборудования.
Известен способ выявления генов β-лактамаз при помощи специализированных олигонуклеотидных микрочипов (Т. Naas, G. Cuzon, P. Bogaerts, Y. Glupczynski and P. Nordmann. Evaluation of a DNA Microarray (Check-MDR CT102) for Rapid Detection of ТЕМ, SHV, and CTX-M Extended-Spectrum β-Lactamases and of KPC, OXA-48, VIM, IMP, and NDM-1 Carbapenemases. J Clin Microbiol. 2011 Apr; 49(4):1608-1613). Олигонуклеотидные микрочипы представляют собой набор микроскопических ячеек, прикрепленных к твердой подложке. Каждая ячейка содержит несколько пикомоль одноцепочной ДНК со специфической последовательностью (ДНК-зонд), иммобилизованной в ячейке. Способ основан на гибридизации между двумя цепями ДНК - ДНК-зондом на чипе и ДНК-мишенью в растворе, чем выше их комплиментарность, тем стабильнее образующийся ДНК-дуплекс. Флуоресцентно меченая ДНК-мишень, связавшись с ДНК-зондом, производит флуоресцентный сигнал, зависящий от условий гибридизации. Сравнение уровней сигналов от различных ячеек позволяет сделать вывод о наличии специфических нуклеотидных последовательностей в исследуемом образце. Главный недостаток способа выявления генов β-лактамаз при помощи специализированных олигонуклеотидных микрочипов заключается в длительности его выполнения, - он реализуется не менее чем за двое суток. Кроме этого, указанный способ требует использования специализированного дорогостоящего оборудования для считывания сигналов с микрочипов и анализа полученной информации.
Наиболее близким к предлагаемому способу идентификации генов клинически значимых семейств β-лактамаз у грамотрицательных микроорганизмов является способ полимеразной цепной реакции (ПЦР) в реальном времени, который используется для выявления генов β-лактамаз класса ОХА (Hemarajata Р, Yang S, Hindler JA, Humphries RM. Development of a Novel Real-Time PCR Assay with High-Resolution Melt Analysis To Detect and Differentiate OXA-48-Like β-Lactamases in Carbapenem-Resistant Enterobacteriaceae. Antimicrob Agents Chemother. 2015 Sep; 59(9):5574-5580). Указанный способ использует общие принципы ПЦР и выполняется в несколько этапов. На первом этапе проводится подготовка материала для исследования. Для этого из биологического материал (кровь, гной, моча, ликвор, экссудат, аспират и т.д.), полученного от пациента, производится экстракция ДНК при помощи коммерческих наборов для лизиса клеток и экстракции ДНК». На втором этапе производится ПЦР-амплификация фрагментов целевых генов β-лактамаз при помощи ПЦР в реальном времени. Предварительно подготовленный лизат смешивается с другими компонентами реакции - праймерами, ДНК-полимеразов, азотистыми основаниями, специальными буферными растворами - и загружается в амплификатор для проведения ПЦР в реальном времени. В способе используются следующие нуклеотидные последовательности праймеров для выявления генов ОХА-β-лактамаз -  и
и 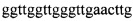 . Режим проведения ПЦР - по варианту «горячего старта» в реальном времени. Оценка реакции производится по нарастанию уровня флуоресценции в последовательных циклах ПЦР, что свидетельствует о наличии у исследуемых штаммов генов ОХА-β-лактамаз. Позитивный результат, отражающий наличие конкретных генов β-лактамаз в исследуемом образце, подтверждается на основе опережения амплификации в пробах с праймерами, соответствующими данному гену. Отрицательные результаты, отражающие отсутствие генов β-лактамаз в исследуемом образце, подтверждаются на основе отсутствии разницы в скорости амплификации в пробах с праймерами, соответствующими данным генам. Главным недостатком способа ПЦР в реальном времени является то, что он позволяет выявлять одновременно только очень ограниченное число генов клинически значимых семейств β-лактамаз, принадлежащих лишь к семейству ОХА, тогда как другие клинически значимые гены β-лактамаз (SHV, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM) при помощи этого способа не могут быть обнаружены.
. Режим проведения ПЦР - по варианту «горячего старта» в реальном времени. Оценка реакции производится по нарастанию уровня флуоресценции в последовательных циклах ПЦР, что свидетельствует о наличии у исследуемых штаммов генов ОХА-β-лактамаз. Позитивный результат, отражающий наличие конкретных генов β-лактамаз в исследуемом образце, подтверждается на основе опережения амплификации в пробах с праймерами, соответствующими данному гену. Отрицательные результаты, отражающие отсутствие генов β-лактамаз в исследуемом образце, подтверждаются на основе отсутствии разницы в скорости амплификации в пробах с праймерами, соответствующими данным генам. Главным недостатком способа ПЦР в реальном времени является то, что он позволяет выявлять одновременно только очень ограниченное число генов клинически значимых семейств β-лактамаз, принадлежащих лишь к семейству ОХА, тогда как другие клинически значимые гены β-лактамаз (SHV, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM) при помощи этого способа не могут быть обнаружены.
Таким образом, в настоящее время существует необходимость в создании нового способа одновременного обнаружения многих генов, ответственных за синтез клинически значимых семейств β-лактамаз, не требующего предварительной культивации патогенных микроорганизмов, дорогостоящих расходных материалов и оборудования, а также отличающегося простотой и быстротой проведения анализа, высокой специфичностью и чувствительностью.
Задачей настоящего изобретения является создание способа одновременного обнаружения клинически значимых семейств β-лактамаз у грамотрицательных микроорганизмов, включая β-лактамазы SHV, СТХ-М, VIM, IMP, NDM, ОХА, KPC, GES, GIM, SPM.
Техническим решением поставленной задачи является быстрое и одновременное получение информации о наличии у исследуемых грамотрицательных бактерий генов β-лактамаз SHV, СТХ-М, VIM, IMP, NDM, ОХА, KPC, GES, GIM, SPM.
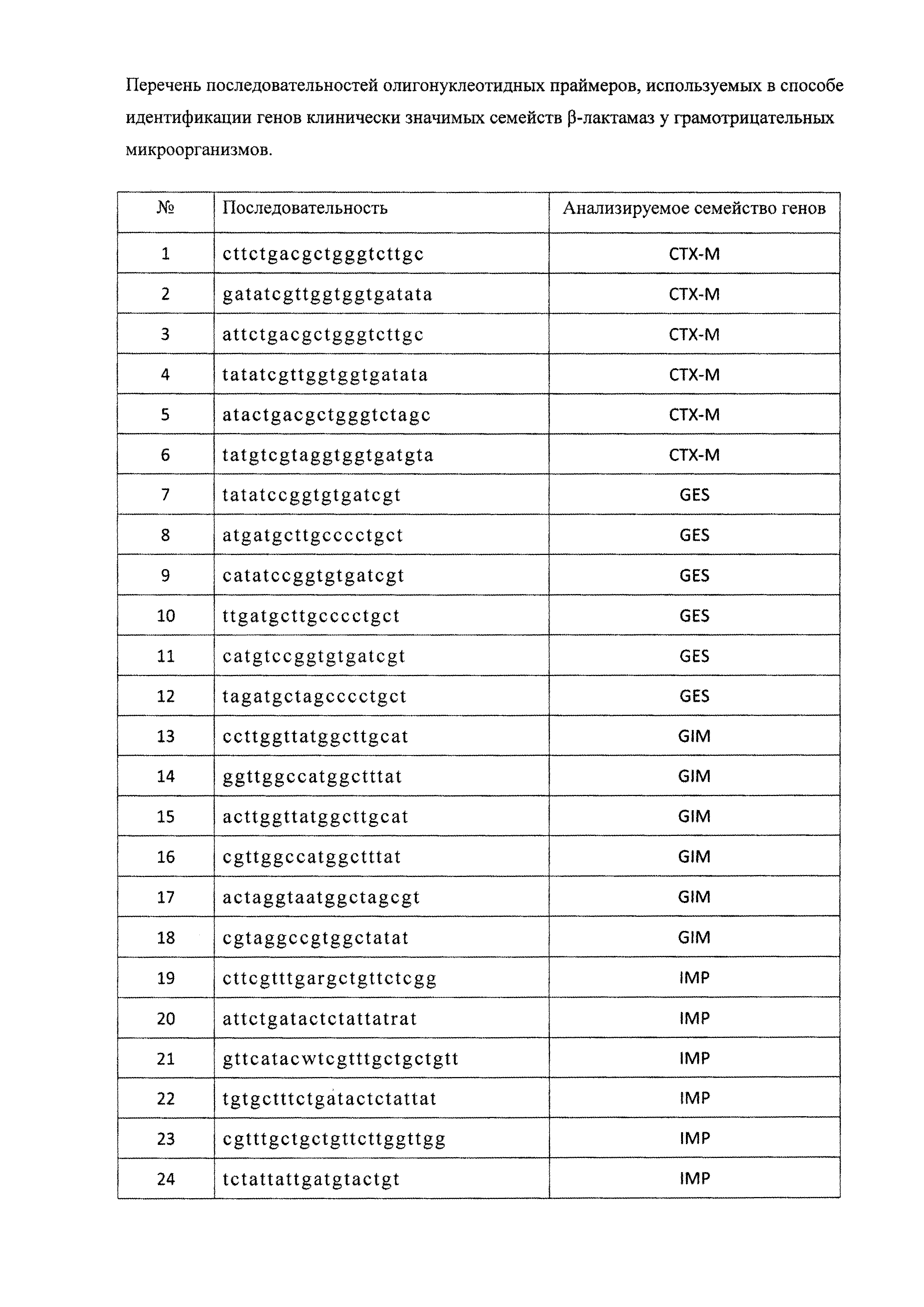
Поставленная задача решается путем амплификации фрагментов генов с помощью полимеразной цепной реакции с набором специфических олигонуклеотидных праймеров и последующим разделением продуктов реакции путем электрофореза в агарозном геле. Набор специфичных олигонуклеотидных праймеров представлен: тремя парами олигонуклеотидных праймеров для амлификации генов семейства СТХ-М с нуклеотидными последовательностями 
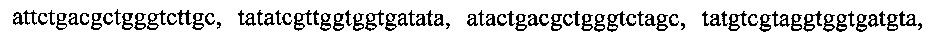 тремя парами олигонуклеотидных праймеров для амлификации генов семейства GES с нуклеотидными последовательностями
тремя парами олигонуклеотидных праймеров для амлификации генов семейства GES с нуклеотидными последовательностями 
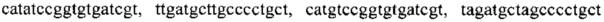 , тремя парами олигонуклеотидных праймеров для амлификации генов семейства GIM с нуклеотидными последовательностями
, тремя парами олигонуклеотидных праймеров для амлификации генов семейства GIM с нуклеотидными последовательностями 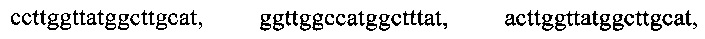
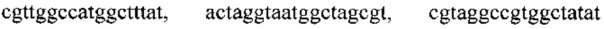 , одиннадцатью парами олигонуклеотидных праймеров для амлификации генов семейства IMP с нуклеотидными последовательностями
, одиннадцатью парами олигонуклеотидных праймеров для амлификации генов семейства IMP с нуклеотидными последовательностями 
 тремя парами олигонуклеотидных праймеров для амлификации генов семейства KPC с нуклеотидными последовательностями
тремя парами олигонуклеотидных праймеров для амлификации генов семейства KPC с нуклеотидными последовательностями 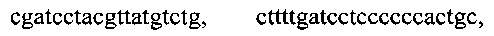
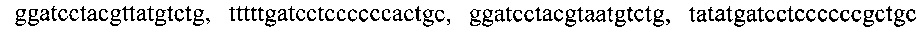 , тремя парами олигонуклеотидных праймеров для амлификации генов семейства NDM с нуклеотидными последовательностями
, тремя парами олигонуклеотидных праймеров для амлификации генов семейства NDM с нуклеотидными последовательностями 
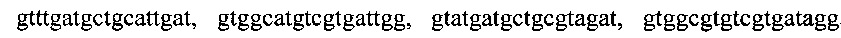 , двенадцатью парами олигонуклеотидных праймеров для амлификации генов семейства ОХА с нуклеотидными последовательностями
, двенадцатью парами олигонуклеотидных праймеров для амлификации генов семейства ОХА с нуклеотидными последовательностями 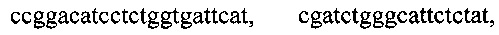

 , тремя парами олигонуклеотидных праймеров для амлификации генов семейства SHV с нуклеотидными последовательностями
, тремя парами олигонуклеотидных праймеров для амлификации генов семейства SHV с нуклеотидными последовательностями 
 тремя парами олигонуклеотидных праймеров для амлификации генов семейства SPM с нуклеотидными последовательностями
тремя парами олигонуклеотидных праймеров для амлификации генов семейства SPM с нуклеотидными последовательностями 
 , тремя парами олигонуклеотидных праймеров для амлификации генов семейства ТЕМ с нуклеотидными последовательностями
, тремя парами олигонуклеотидных праймеров для амлификации генов семейства ТЕМ с нуклеотидными последовательностями 
 , тремя парами олигонуклеотидных праймеров для амлификации генов семейства VIM с нуклеотидными последовательностями
, тремя парами олигонуклеотидных праймеров для амлификации генов семейства VIM с нуклеотидными последовательностями 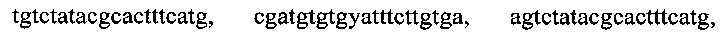
 .
.
Способ выполняется в несколько этапов.
ЭТАП 1. Подготовка материала для исследования
На 1-м этапе из биологического материал (кровь, гной, моча, ликвор, экссудат, аспират и т.д.), полученного от пациента, производится экстракция ДНК. Для этого биологический материал в количестве от 1 до 10 мл центрифугируется при 10000 оборотах в минуту в течение 2 минут, надосадочная жидкость удаляется, к осадку добавляется 0,35 мл ТЕ-буфера из коммерческого «Набора для лизиса ДНК» (производство AmliSens). Осадок в ТЕ-буфере обрабатывается на вортексе 30 секунд.
ЭТАП 2. ПЦР-амплификация фрагментов целевых генов
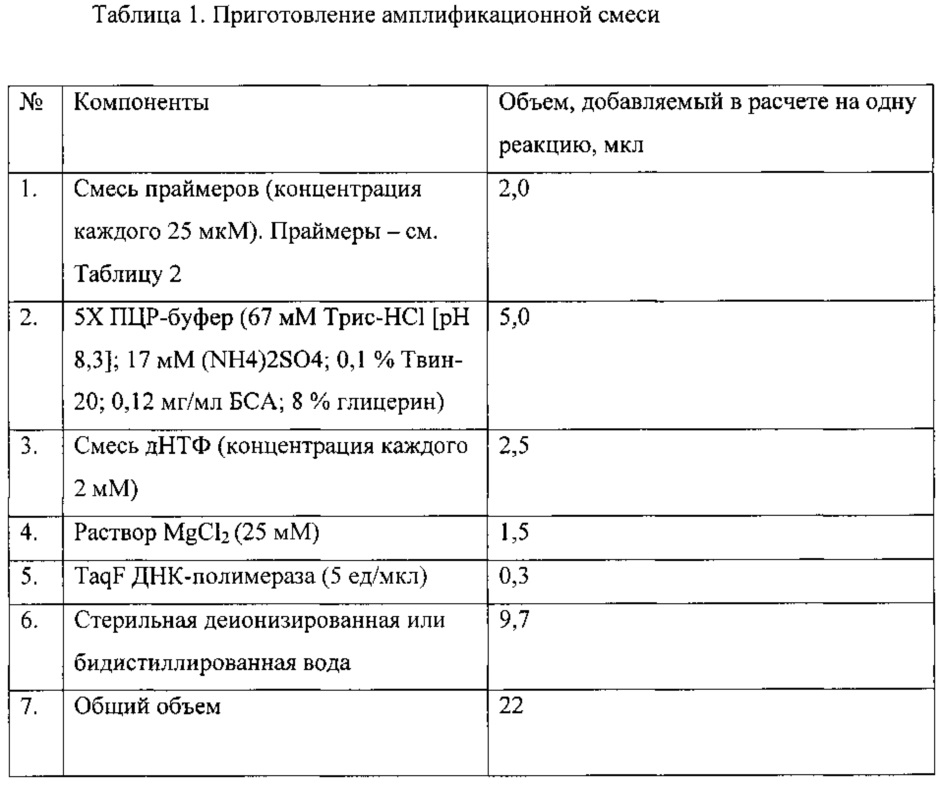
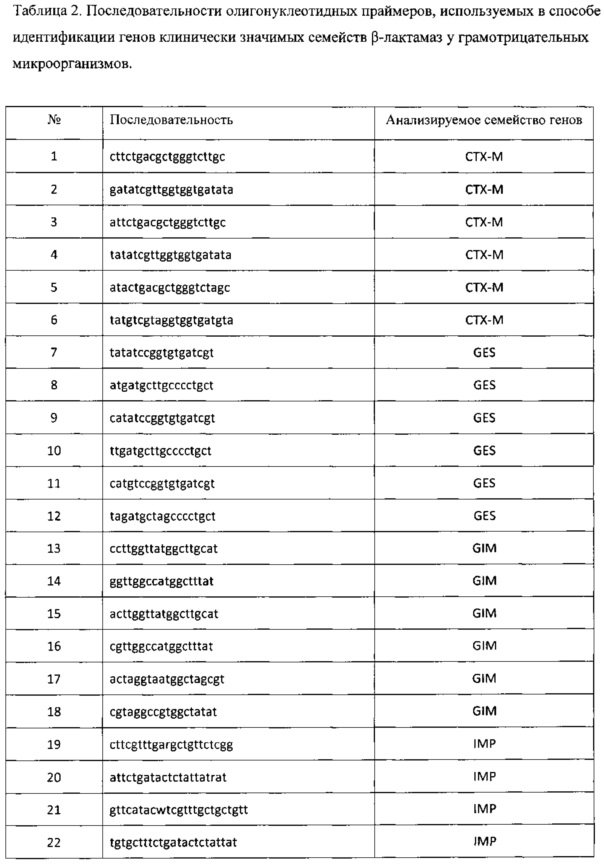
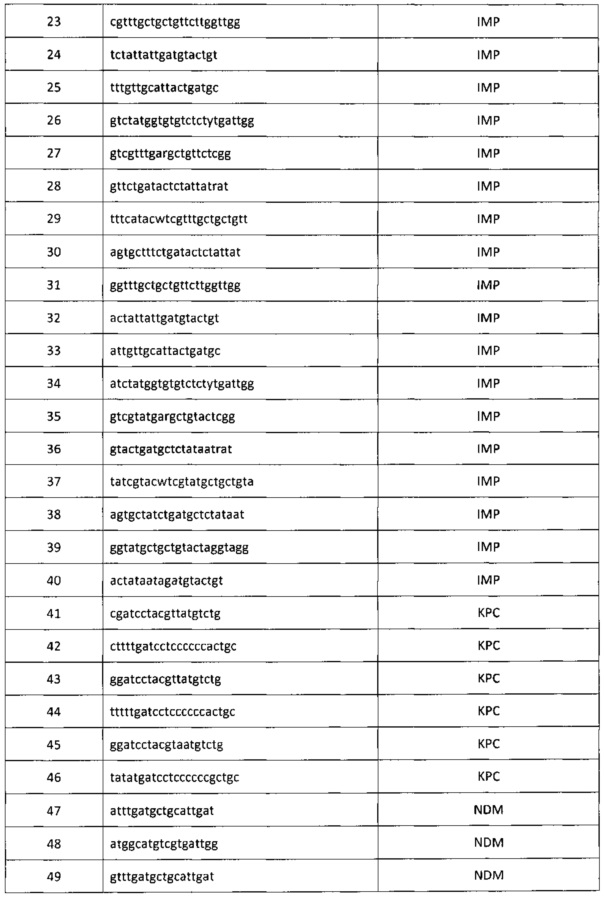
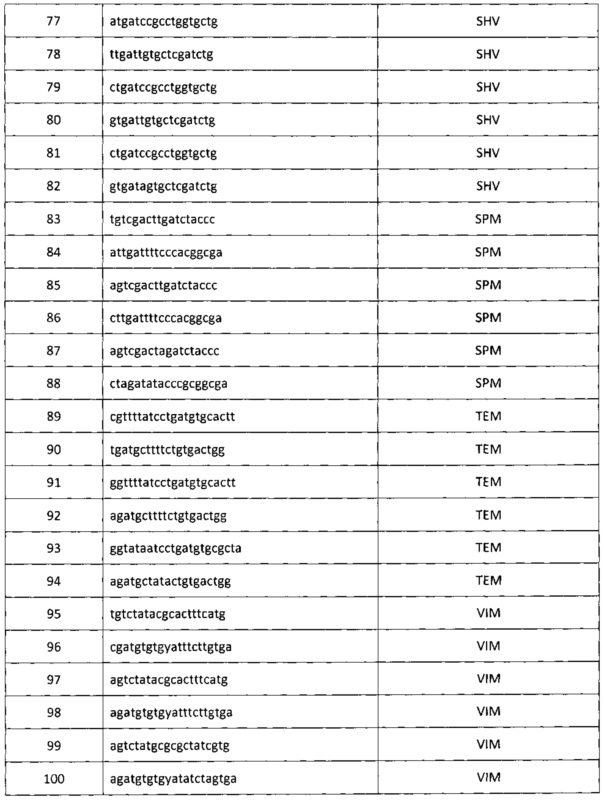
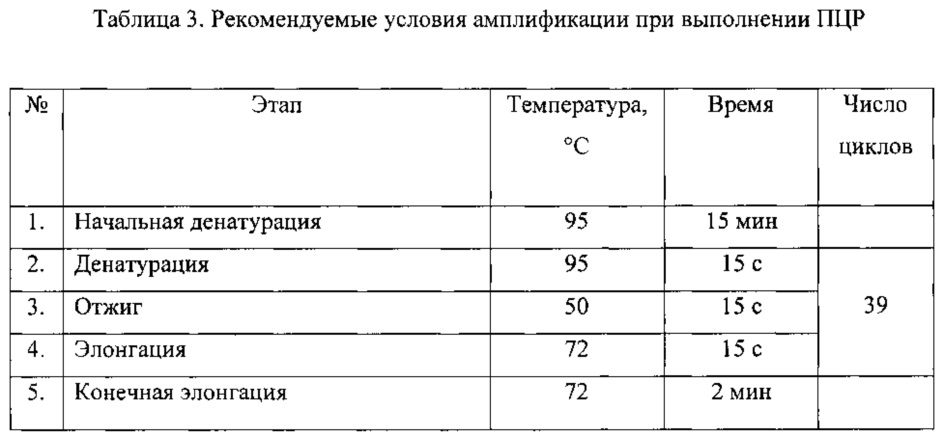
На 2-м этапе производится амплификация в исследуемом образце генов β-лактамаз (SHV, ОХА, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM) при помощи на ДНК-амплификаторе. Предварительно подготовленный лизат (см. выше - «Этап 1») в количестве 3 мкл смешивается с другими компонентами реакции в количествах и порядке, указанном в Таблице 1. Конечный объем реакционной смеси составляет 25 мкл с учетом добавления матричной ДНК. Нуклеотидные последовательности праймеров для выявления генов β-лактамаз, указаны в Таблице 2 и Перечне нуклеотидных последовательностей праймеров, прилагаемом к настоящей заявке. Праймеры для проведения полимеразной цепной реакции были синтезированы на автоматическом синтезаторе 394 DNA/RNA synthesizer (Applied Biosystems, США). Режим проведения ПЦР отражен в Таблице 3. В качестве положительного контроля используют синтетические цепочки ДНК, идентичные естественным генам β-лактамаз.
ЭТАП 3. Оценка результатов
Продукты амплификации, полученные с помощью полимеразной цепной реакции с использованием данного набора праймеров, подвергают разделению путем проведения электрофореза в агарозном геле в камере для электрофореза. Извлекают гель из электрофорезной камеры и просматривают в УФ-свете на транс иллюминаторе. Результаты документируют путем фотографирования геля и сохранения полученных изображений. Далее, проводится анализ полученных изображений электрофореграмм. По расположению специфических полос для продуктов амплификации проводится интерпретация электрофореграммы, вывод о наличии у анализируемых образцов идентифицируемых генов β-лактамаз делается не основе сравнения изображений исследуемых образцов и положительных контролей (см. Этап 2).
Эффективность предлагаемого способа продемонстрирована на следующих примерах.
Пример 1
В качестве объекта исследования использовали референсные музейные штаммы бактерий, несущих заведомо известные гены β-лактамаз: Acinetobacter baumannii, несущий ген β-лактамазы ОХА, Pseudomonas aeruginosa, несущая гены β-лактамаз VIM и IMP, Klebsiella pneumoniae, несущая гены β-лактамаз KPC и NDM, Burkholderia cepacia, несущая ген β-лактамазы SHV, Enterobacter faecalis, несущий ген β-лактамазы СТХ-М, Stenotrophomonas maltophilia, несущая ген β-лактамазы GES, Elizabethkingia meningoseptica, несущая ген β-лактамазы GIM, Serratia marcescens, несущая ген β-лактамазы SPM. Из суточных бульонных (бульон Мюллера-Хинтона) культур перечисленных выше бактерий было отобрано по 1 мл жидкости. Отобранный материал в количестве от 1 мл центрифугировался при 10000 оборотах в минуту в течение 2 минут, надосадочная жидкость была удалена, к осадку был добавлен ТЕ-буфера (0,35) мл из коммерческого «Набора для лизиса ДНК» (производство AmliSens). Осадок в ТЕ-буфере был обработан на вортексе 30 секунд. Затем в исследуемом образце была произведена амплификация генов β-лактамаз (SHV, ОХА, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM) на ДНК-амплификаторе «BioRad ThermoCycler С1000» со смесью праймеров, указанных в Таблице 2 в условиях, описанных выше. В качестве положительного контроля использовали синтетические цепочки ДНК, идентичные естественным генам β-лактамаз. Продукты амплификации, полученные с помощью полимеразной цепной реакции с использованием данного набора праймеров, были подвергнуты разделению путем проведения электрофореза в агарозном геле в камере для электрофореза. Результаты документированы путем фотографирования геля в трансиллюминаторе и сохранения полученных изображений. Далее, было проведено сравнение специфических полос положительного контроля и полос анализируемых образцов. Была выявлена идентичность полос в образцах с синтетическими цепочками ДНК, соответствующими генам SHV, ОХА, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM, и полос в анализируемых образцах. Результаты отражены в таблице 4. Данный пример показывает возможность быстрого и правильного выявления генов β-лактамаз SHV, ОХА, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM у возбудителей музейных штаммов.
Пример 2
В качестве объекта исследования использовали клинический материал - отделяемое из гнойной раны от больного из хирургического отделения. Предварительно путем классического микробиологического исследования в материале были обнаружены бактерии, относящиеся к виду Pseudomonas aeruginosa. Полученный материал в количестве от 1 мл центрифугировался при 10000 оборотах в минуту в течение 2 минут, надосадочная жидкость была удалена, к осадку был добавлен ТЕ-буфера (0,35) мл из коммерческого «Набора для лизиса ДНК» (производство AmliSens). Осадок в ТЕ-буфере был обработан на вортексе 30 секунд. Затем в исследуемом образце была произведена амплификация генов β-лактамаз (SHV, ОХА, СТХ-М, VIM, IMP, NDM, KPC, GES, GIM, SPM) на ДНК-амплификаторе «BioRad ThermoCycler C1000» со смесью праймеров, указанных в Таблице 2, в условиях, описанных выше. В качестве положительного контроля использовали синтетические цепочки ДНК, идентичные естественным генам β-лактамаз. Продукты амплификации, полученные с помощью полимеразной цепной реакции с использованием данного набора праймеров, были подвергнуты разделению путем проведения электрофореза в агарозном геле в камере для электрофореза. Результаты документированы путем фотографирования геля в трансиллюминаторе и сохранения полученных изображений. Далее, было проведено сравнение специфических полос положительного контроля и полос анализируемых образцов. Была выявлена идентичность полос в образцах с синтетическими цепочками ДНК, соответствующими генам ОХА, СТХ-М, VIM, IMP, и полос в анализируемом образце. Это позволило сделать вывод о наличии генов β-лактамаз ОХА, СТХ-М, VIM, IMP у синегнойной палочки (Pseudomonas aeruginosa), изолированной из гнойной раны. Данный пример показывает возможность быстрого одновременного выявления генов β-лактамаз ОХА, СТХ-М, VIM, IMP у возбудителя из клинического образца.