Результат интеллектуальной деятельности: СПОСОБ ВИДОВОЙ И ШТАММОВОЙ ИДЕНТИФИКАЦИИ БИФИДОБАКТЕРИЙ ФИЛОТИПА Bifidobacterium longum
Вид РИД
Изобретение
Область техники, к которой относится изобретение
Изобретение относится к биотехнологии, в частности к производству бактериальных препаратов и продуктов питания, а также к медицинской микробиологии, и может быть использовано для идентификации штаммов бифидобактерий.
Уровень техники
Известно, что клинические проявления дисбактериоза кишечника, как правило, отмечаются на фоне отсутствия или дефицита бифидобактерий, которые в норме являются основой микрофлоры кишечника людей всех возрастов. (Бондаренко В.М., Грачева Н.М., Мацулевич Т.В. Дисбактериозы кишечника у взрослых. М.: КМК Scientifi с Press, 2003, 224 с; Leahy S.C., Higgins D.G., Fitzgerald G.F and van Sinderen D. Getting better with bifidobacteria. Journal of Applied Microbiology, 2005, 98, 1303-1315). Именно поэтому бифидобактерии широко используются в составе заквасок для приготовления различных кисломолочных продуктов и в виде бакпрепаратов, применяемых в лечебно-профилактических целях. В связи с этим очень важно разработать точные методы идентификации видов и штаммов бифидобактерий (Мазанкова Л.Н., Лыкова Е.А. Пробиотики: характеристика препаратов и выбор в педиатрической практике // Детские инфекции. 2004, №1, с.18-23; Бифидобактерии и их использование в клинике, медицинской промышленности и сельском хозяйстве / Под ред. Никитина В.А. М., 1986, 119 с. Бондаренко В.М., Воробьев А.А. Дисбиозы и препараты с пробиотической функцией // Журн. Микробиолог.- 2004. - №1. - С.84-92; Stanton С., Ross R.P., Fitzgerald G.F. and Van Sinderen, D. (2005) Fermented functional foods based on probiotics and their biogenic metabolites. Curr. Opin.Biotechnol. 16, 198-203).
В настоящее время для идентификации вида и рода бифидобактерий применяются следующие методы:
1. Метод, основанный на амплификации фрагментов гена 16S pPHK. (Youn SY, Seo JM, Ji GE. Evaluation of the PCR method for identification of Bifidobacterium species. Lett Appl Microbiol. 2008, Jan, 46(1):7-13; Krizova, J., Spanova, A. and Rittich, B. Evaluation of amplified ribosomal DNA restriction analysis (ARDRA) and species-specific PCR for identification of Bifidobacterium species. Syst Appl Microbiol. 2006, 29, 36-44; Kwon, H.S., Yang, E.H., Lee, S.H., Yeon, S.W., Kang, B.H. and Kim, T.Y. Rapid identification of potentially probiotic Bifidobacterium species by multiplex PCR using species-specific primers based on the region extending from 16S rRNA through 23S rRNA. FEMS Microbiol Lett. 2005, 250, 55-62; Mullie, С., Odou, M.F., Singer, E., Romond, M.D. and Izard, D. Multiplex PCR using 16S rRNA gene-targeted primers for the identification of Bifidobacteria from human origin. FEMS Microbiol Lett. 2003, 222, 129-136; Патент Япония, United States Patent 7,321,032, 2008; Патент P 200601788 ES, 2006; Патент KR 20050012505, 2005).
По нуклеотидным последовательностям генов 16S рРНК разработаны родо-, группо- и видоспецифичные праймеры, амплификация с которыми в ряде случаев может проводиться одновременно в общей реакционной смеси (так называемая мультиплексная ПЦР - multiplex PCR) (Matsuki Т., Watanabe К., Fujimoto J., Kado Y., Takada Т., Matsumoto K., Tanaka R. Quantitative PCR with 16S rRNA-gene-targeted species-specific primers for analysis of human intestinal Bifidobacteria. Applied and environmental microbiology, Jan. 2004, p.167-173). Однако межвидовой уровень сходства по гену 16S рРНК варьирует в пределах 93-99%, и многие виды бифидобактерий филогенетически близки между собой.
2. Метод двулокусного секвенирования, в частности, с использованием β-субъединицы F0F1-АТФазы. (Ventura М., Canchaya С., van Sinderen D,, Fitzgerald G.F., Zink R. Bifidobacterium lactis DSM 10140: Identification of the atp (atpBEFHAGDC) Operon and Analysis of Its Genetic Structure, Characteristics, and Phytogeny. Applied and environmental microbiology, May 2004, p.3110-3121.).
3. Метод с использованием 7 консервативных генов «домашнего хозяйства» clpC, dnaB, dnaG, dnaJ1, purF, rpoC и xfp, комбинация последовательностей которых позволяет выявить филогенетические отличия между видами бифидобактерий. (Ventura, М., Canchaya, С., Del Casale, A., Dellaglio, F., Neviani, E., .Fitzgerald, G. F. and van Sinderen, D. Analysis of bifidobacterial evolution using a multilocus approach. Int. J. Syst. Evol. Microbiol. 2006, 56, 2783-2792.).
Однако перечисленные выше методы не позволяют проводить штаммоспецифическую идентификацию, поскольку нуклеотидные последовательности генов, используемых для видовой идентификации, полностью идентичны для разных штаммов одного вида.
Для генотипирования штаммов вида В. animalis subsp. lactis разработан метод, основанный на однонуклеотидных полиморфизмах, вставках и делениях. (Briczinski Е.Р., Loquasto J.R., Barrangou R., Dudley E.G., Roberts A.M., Roberts R.F. Strain-specific genotyping of Bifidobacterium animalis subsp. lactis by using single-nucleotide polymorphisms, insertions, and deletions. Appl Environ Microbiol. 2009 Dec; 75(23):7501-8. Epub 2009 Oct 2.).
Для штаммов других видов Bifidobacterium (в том числе В.longum) до настоящего времени не разработаны методы идентификации.
Раскрытие изобретения
Задачей настоящего изобретения является создание экспресс-метода молекулярно-генетического типирования штаммов бифидобактерий и, в частности, В.longum.
В заявленном изобретении предложен метод идентификации штаммов филотипа В.longum, основанный на комбинации и полиморфизме генов токсин-антитоксин системы relBE. Действие систем токсин-антитоксин в ряде работ квалифицируют как адаптацию к стрессовым условиям (субстратное голодание или другие стрессы), что позволяет выжить остальным бактериям. (Прозоров А.А., Даниленко В.Н. Системы "токсин-антитоксин" у бактерий: инструмент апоптоза или модуляторы метаболизма? Микробиология. 2010. Т.79. №2. С.147-159).
1. Биоинформатический анализ наличия генов систем токсин-антитоксин mazEF и relBE у бифидобактерий
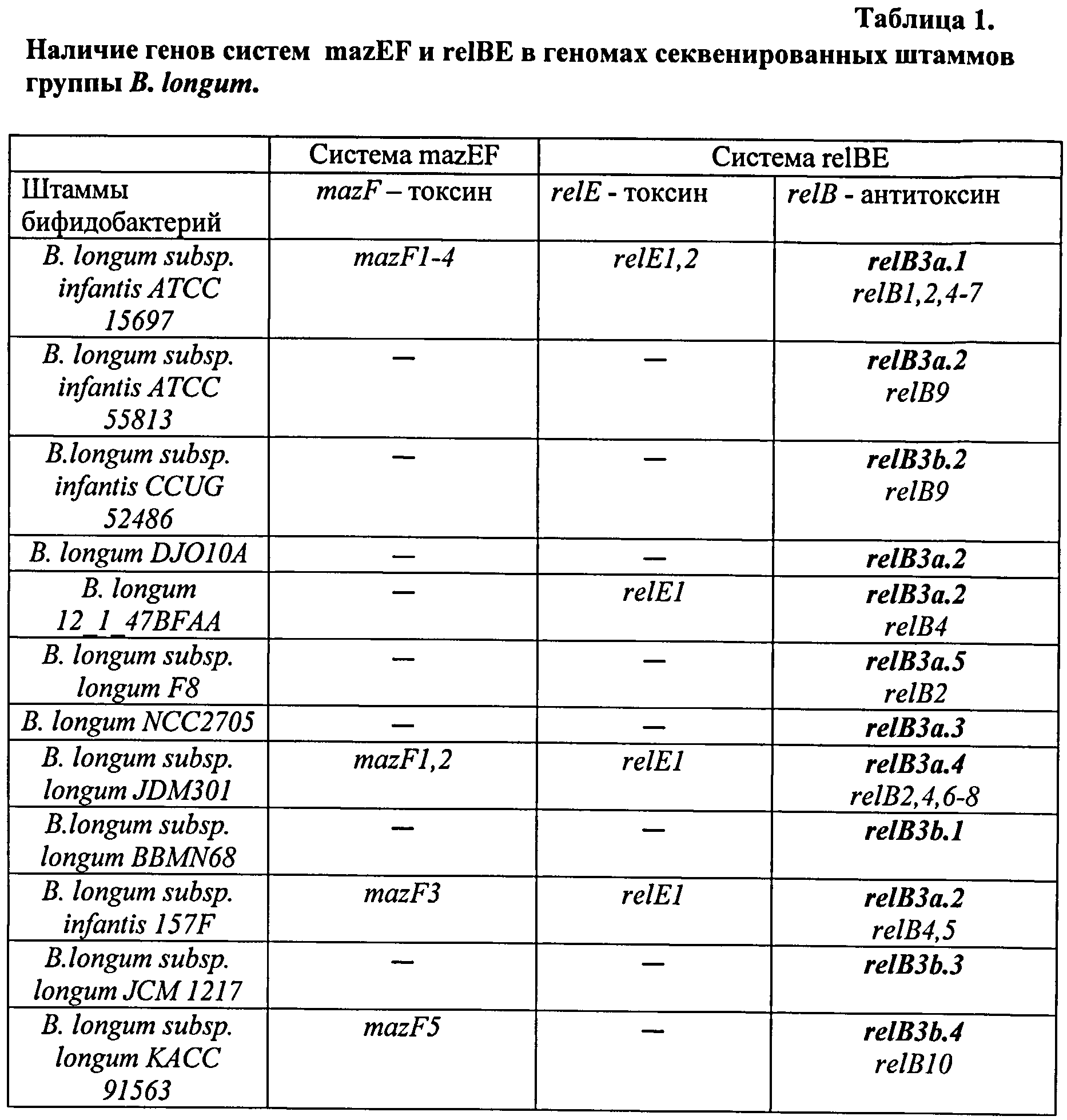
По данным на 1.10.2011 года, в базе данных представлено 11 геномов, относящихся к группе В.longum (7 штаммов подвида В.longum subsp. longum и 4 штамма подвида В.longum subsp. infantis), а также штамма Bifidobacterium sp. 12_1_47BFAA, также относящегося к виду В.longum.
Авторами настоящего изобретения был проведен компьютерный поиск генов систем MazEF и RelBE в геномах всех секвенированных штаммов бифидобактерий с использованием программ National Center for Biotechnology Information (NCBI) (http://www.ncbi.nlm.nih.gov/) и UniProt (http://www.uniprot.org/). Сравнительный анализ последовательностей обнаруженных генов проводили с использованием программ Blast (http://blast.ncbi.nlm.nih.gov/) и CLUSTAL W (http://www.ch.embnet.org/software/ClustalW.html).
В результате биоинформатического анализа обнаружены существенные различия штаммов филотипа B.longum по наличию генов данных систем токсин-антитоксин (см. табл.1). Модули системы mazEF (перекрывающиеся гены) присутствуют только в геномах 3 штаммов В.longum - АТСС 15697, JDM301 и 157F. Гены токсина relE присутствуют только в геномах 4 штаммов - АТСС 15697, JDM301,157F и 12_1_47BFAA.
Во всех геномах штаммов группы В.longum обнаружены гены антитоксина relB. В штамме В. longum subsp. infantis АТСС 15697 присутствует наибольшее количество (7) генов антитоксина relB (обозначены нами как relB1-relB7 в порядке расположения в геноме). В штамме В.longum subsp. longum JDM301 присутствует 6 генов антитоксина relB (отсутствуют гены, гомологичные генам relB1 и relB5, но присутствует ген, обозначенный как relB8). В других штаммах В.longum обнаружено от 1 до 3 генов антитоксина relB. Таким образом, нами был обнаружен полиморфизм генов антитоксина relB на штаммовом уровне.
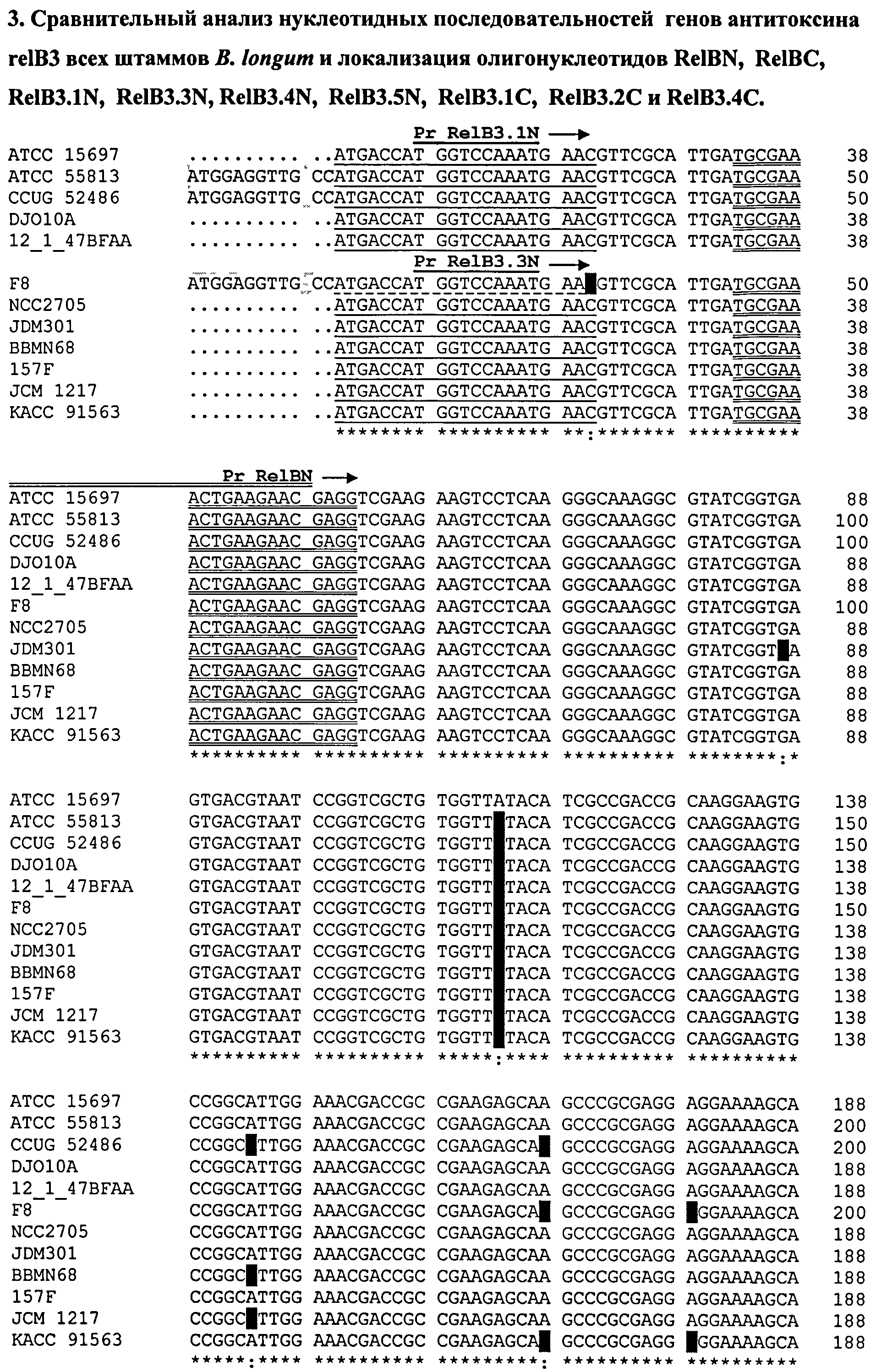
Ген, обозначенный как relB3, присутствует во всех геномах B.longum, имеет высокую гомологию от 98 до 99% по всей нуклеотидной последовательности, за исключением С-концевой области, и может быть использован для видовой идентификации штаммов филотипа В.longum.
Гены антитоксина relB1 - relB10 имеют низкую гомологию между собой (15-30%), что может служить штаммоспецифическим признаком, кроме того, в нуклеотидных последовательностях генов relB имеются точечные отличия и вставки, что позволяет разработать штаммоспецифические олигонуклеотиды.
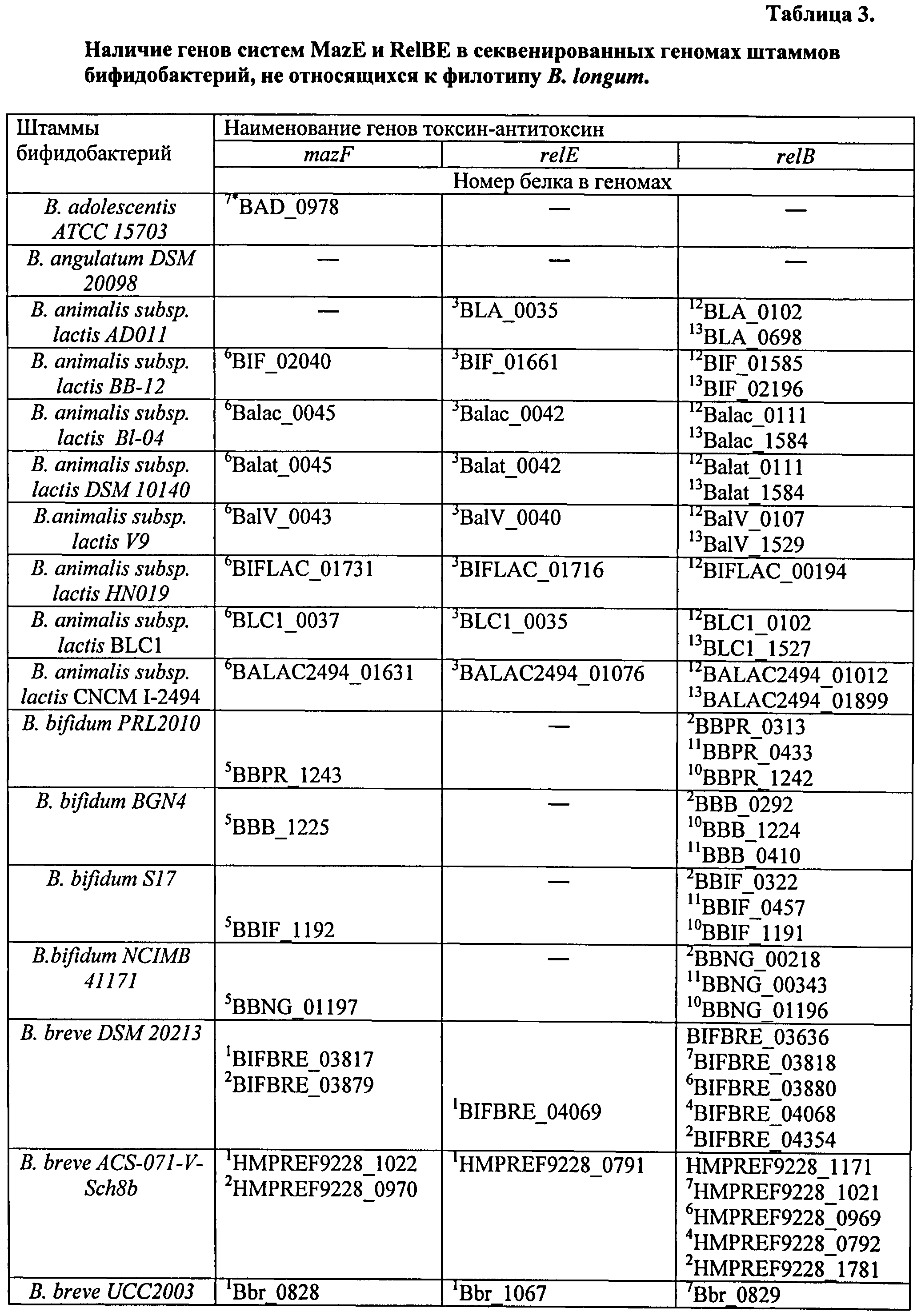
Гены систем токсин-антитоксин mazEF и relBE обнаружены и в геномах других видов рода Bifidobacterium: в геномах 6 штаммов вида В.animalis subsp. lactis присутствует по 1 модулю системы mazEF (перекрывающиеся гены), по 1 гену токсина relE и по 2 гена антитоксина relB; в геномах 3 штаммов вида В.bifidum - по 1 модулю системы mazEF и по 3 гена антитоксина relB; в геномах 3 штаммов вида В.breve - по 2 модуля системы mazEF, по 1 гену токсина relE и от 4 до 5 генов антитоксина relB. В геноме штамма B.adolescentis АТСС 15703 присутствует 1 модуль системы mazEF.
Таким образом, с использованием генов систем mazEF и relBE можно также проводить видовую идентификацию всех видов бифидобактерий.
2. Разработка олигонуклеотидов для идентификации В.longum
2.1. Идентификация филотипа В.longum
Поскольку ген антитоксина, обозначенный нами как relB3, присутствует во всех геномах B.longum, на основании нуклеотидных последовательностей консервативного гена были сконструированы олигонуклеотиды для видовой идентификации с использованием Программ CLUSTALW (http://www.ch.embnet.org/software/ClustalW.html) и Adv.BLAST (http://www.ncbi.nlm.nih.gov/blast/Blast.cgi).
На последовательности 3 (см. перечень последовательностей) представлен сравнительный анализ нуклеотидных последовательностей генов антитоксина relB3 штаммов B.longum и локализация олигонуклеотидов ReBN и ReBC для видовой идентификации. На всех представленных нуклеотидных последовательностях отличия выделены красным цветом, вставки - желтым цветом.
2.2. Штаммоспецифическая идентификация
С увеличением числа доступных секвенированных геномов бифидобактерий появилась возможность идентифицировать участки ДНК, специфичные для каждого конкретного штамма.
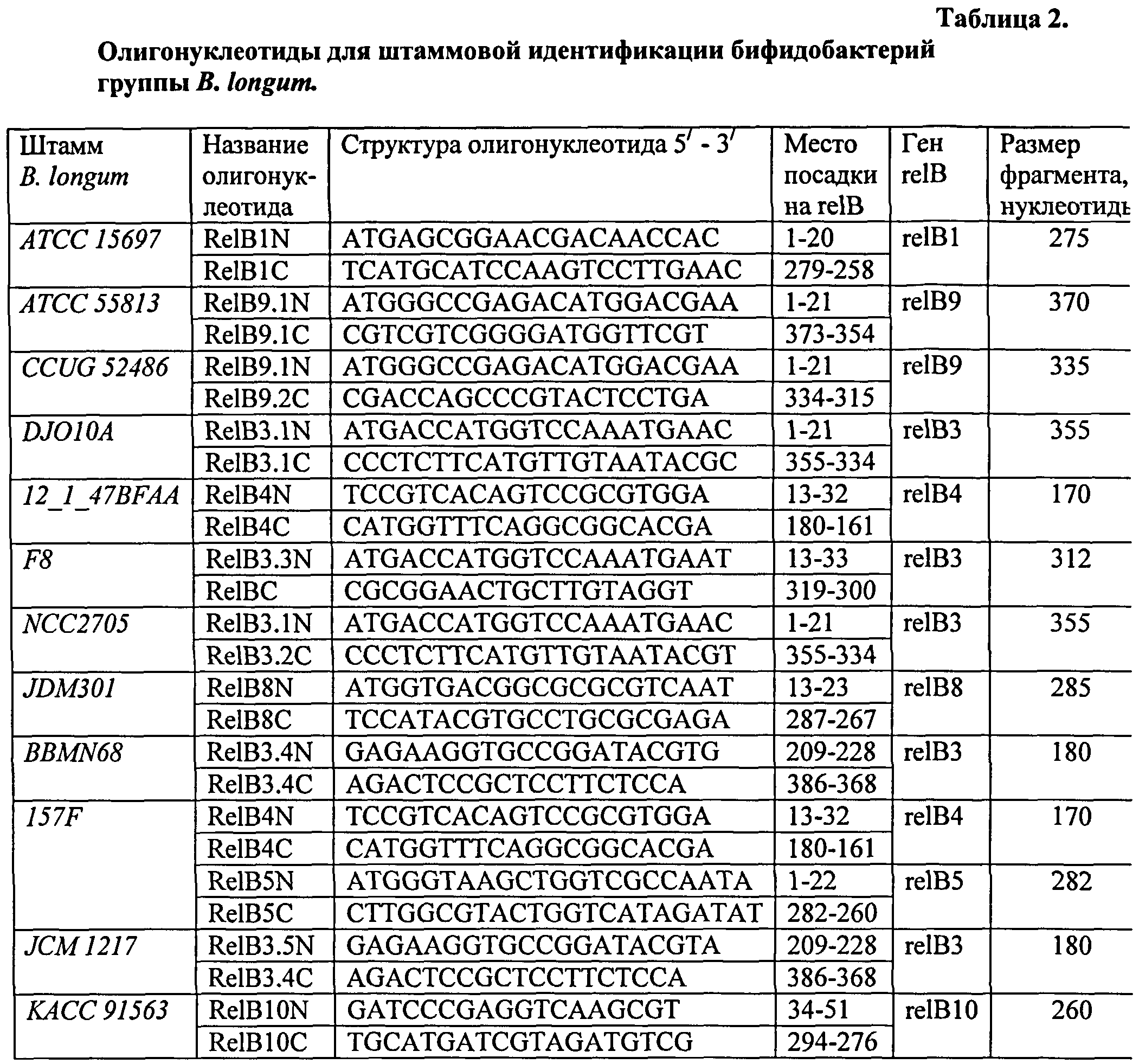
Как было сказано выше, наибольшее количество (7) генов антитоксина relB присутствует в геноме штамма АТСС 15697, причем ген relB1 является штаммоспецифическим, т.к. отсутствует в геномах остальных штаммов. В связи с этим, для идентификации данного штамма нами были сконструированы олигонуклеотиды на основе нуклеотидной последовательности гена relB1 (последовательность 1).
Также гены relB8 и relB10 являются специфическими для штаммов JDM301 и ААСС 91563 соответственно (последовательности 6, 8).
Ген relB2 присутствует в геномах штаммов АТСС 15697, JDM301 и F8; ген relB4 - в геномах штаммов АТСС 15697, JDM301, 157F и 12_1_47BFAA; ген relB5 - геномах штаммов АТСС 15697 и 157F; ген relB9 - геномах штаммов АТСС 55813 и CCUG 52486. Сравнительный анализ последовательностей данных генов для разных штаммов выявил однонуклеотидные отличия, что также позволило сконструировать штаммоспецифические олигонуклеотиды (последовательности 2, 4, 5, 7).
Геномы штаммов DJO10A, NCC2705, BBMN68 и JCM 1217 содержат только ген антитоксина relB3, присутствующий также в геномах всех штаммов B.longum (последовательность 3).
По нуклеотидной последовательности гена антитоксина relB3 штаммы В.longum можно разбить на 2 основные группы:
Группа relB3a. К ней относятся штаммы: АТСС 15697, АТСС 55813, DJO10A, 12_1_47BFAA, F8, NCC2705, JDM301,157F.
Группа relB3b. В отличие от группы relB3A (нумерация дана для штамма АТСС 15697) нуклеотидная последовательность содержит замены (A114→G; C216→T; A217→G; C309→T; A310→G; A312→G; G315→А; T316→G; A317→C; C318→Т; G324→А; А326→G; A327→G; Т330→G; G331→A; A334→G; T337→C; A338→G; C342→T; A344→G; C345→A; T347→G; A350→G; G351→C; A352→G; G354→A; C356→T; G358→T; A359→G) и вставки (15 нуклеотидов в области между 332 и 333 нуклеотидами; 4 нуклеотида в области между 342 и 343 нуклеотидами; 2 нуклеотида в области между 360 и 361 нуклеотидами).
К ней относятся штаммы: CCUG 52486, BBMN68, JCM 1217 и КАСС 91563.
Группу relB3a можно разбить на подгруппы:
Подгруппа relB3a.1. К ней относится штамм АТСС 15697.
Подгруппа relB3a.2. Отличия от relB3A.1 - A114→G; C216→Т; А217→G; G228→А; А334→G.
К ней относятся штаммы: АТСС 55813 (содержит дополнительно ген relB9),
DJO10A (содержит только ген relB3),
12_1_47BFAA (содержит дополнительно ген relB4),
157F (содержит дополнительно гены relB4 и relB5).
Подгруппа relB3a.3. Отличия от relB3A.1 - A114→G; С216→T; A217→G; G228→А.
К ней относится штамм NCC2705.
Подгруппа relB3a.4. Отличия от relB3A.1 - G87→A; А114→G; C216→T; A217→G; А334→G.
К ней относится штамм JDM301.
Подгруппа relB3a.5. Отличия от relB3A.1 - C21→T; A114→G; A168→G; A179→C; C216→T; А217→G; C356→T. К ней относится штамм F8.
Группу relB3b можно разбить на подгруппы:
Подгруппа relB3b.1. Содержит замену A144→С.
К ней относится штамм BBMN68.
Подгруппа relB3b.2. Содержит замены А144→C; A168→G.
К ней относится штамм CCUG 52486.
Подгруппа relB3b.3. Содержит замены А144→С; G228→А.
К ней относится штамм JCM 1217.
Подгруппа relB3b.4. Содержит замены А168→G; A179→C.
К ней относится штамм КАСС 91563.
Все выявленные при сравнении последовательностей генов антитоксина relB отличия позволяют проводить идентификацию штаммов В.longum.
3. Разработка олигонуклеотидов для видовой идентификации Bifidobacterium, не относящихся к филотипу В.longum
Биоинформатический анализ наличия генов токсин-антитоксин систем MazEF и RelBE у других видов бифидобактерий представлен в таблице 3.
В геномах штаммов вида В.bifidum присутствует по 3 гена антитоксина relB. Сравнительный анализ нуклеотидных последовательностей генов relB штаммов В.bifidum с генами антитоксина relB1-relB10 штаммов В.longum показал, что 2 гена имеют высокую идентичность (от 98 до 99%) с генами relB2 и relB10, третий ген антитоксина имеет низкую идентичность (15-30%) и обозначен нами как relB11. В связи с этим, нами были сконструированы олигонуклеотиды RelB11BbN и RelB11BbC для проведения видовой идентификации В.bifidum на основе нуклеотидной последовательности гена relB11 (последовательность 9).
В геноме штамма В.adolescentis АТСС 15703 присутствует только 1 ген токсина mazF. Сравнительный анализ его нуклеотидной последовательности с mzzF генами показал низкую идентичность с подобными генами штаммов В.longum, В.animalis subsp. lactis, В.bifidum и В.breve. В связи с этим, для проведения идентификации бифидобактерий, относящихся к виду В.adolescentis, нами были сконструированы олигонуклеотиды MazBaN и MazBaC на основе последовательности гена токсина mazF (последовательность 10).
Аналогично можно разработать олигонуклеотиды для идентификации других видов бифидобактерий - В.animalis subsp. lactis и В.breve.
Таким образом:
1) Метод идентификации бифидобактерий по настоящему изобретению основан на комбинации и полиморфизме генов токсин-антитоксин систем RelBE и MazEF.
2) Предлагаемый метод предусматривает проведение амплификации с геномной ДНК с использованием предлагаемого набора пар олигонуклеотидов для видовой и штаммовой идентификации бифидобактерий группы В.longum.
3) Предлагаемый метод может быть использован и для видовой идентификации Bifidobacterium bifidum и Bifidobacterium adolescentis и других видов бифидобактерий.
Осуществление изобретения
Культуру бифидобактерий выращивают на агаризованной среде MPC с 0,05% цистеина в течение 48 часов при 37°C при анаэробных условиях в анаэростате HiAnaerobicTM (Компания "HiMedia" (Индия)), после чего выделяют геномную ДНК.
Культуру бифидобактерий центрифугируют при 8000 об/мин в течение 10 минут. Осадок суспендируют в 0,6 мл буфера TEST (10 mM трис HCl pH 8,0; 5 mM ЭДТА pH 8,0; 1М NaCl; 1/200 объема Тритон-X100). Осторожно перемешивают. Инкубируют при 37°C в течение 12 часов. Добавляют 150 мкл свежеприготовленного раствора лизоцима (исходная концентрация 100 мг/мл); добавляют 10 мкл раствора РНКазы (исходный раствор 10 мг/мл). Осторожно перемешивают. Инкубируют при 37°C в течение 3 часов (суспензия должна стать слегка вязкой). Добавляют 150 мкл раствора протеиназы K (исходный раствор 20 мг/мл) и 150 мкл 10% саркозила. Осторожно перемешивают. Инкубируют при 37°C в течение 24 часов до полного лизиса. Добавляют 100 мкл 25% додецилсульфата натрия, перемешивают, инкубируют при 55°C в течение 2,5 часов (суспензия должна посветлеть). К лизату клеток добавляют 1 объем фенола. Содержимое эппендорфа интенсивно, но осторожно перемешивают переворачиванием в течение 1 мин. Центрифугируют 10 мин при 12000 об/мин. Водную фазу отбирают в новый эппендорф, повторяют обработку фенолом. Водную фазу отбирают в чистый эппендорф и добавляют 1 объем хлороформа с изоамиловым спиртом. Содержимое эппендорфа перемешивают переворачиванием в течение 1 мин. Центрифугируют 5 мин при 12000 об/мин. Водную фазу переносят в чистый эппендорф, добавляют 1 мл изопропанола, перемешивают переворачиванием (появится осадок в виде тяжей). Центрифугируют в течение 1 мин при 10000 об/мин, супернатант удаляют. Осадок промывают 70% этиловым спиртом, высушивают и растворяют в 150 мкл воды.
Амплификацию ДНК проводят с использованием набора «Амплификация» фирмы «Dialat Ltd» на приборе «Терцик» («ДНК-технология»). Состав смеси для ПЦР (на 100 мкл): 10 мкл 10×ПЦР буфера, 10 мкл смеси 2,5 mM ΣdNTPs, 4 мкл 50 mM MgCl2, 0,3 мкг геномной ДНК и 0,8 мкл фермента Taq-полимеразы. Олигонуклеотидные праймеры добавляют в концентрации 20 пмоль на 100 мкл смеси.
Параметры ПЦР реакции: 95°C в течение 5 мин (лизис клеток и денатурация геномной ДНК); затем 30 циклов амплификации - 94°C - 1 мин (денатурация), 56°C в течение 1 мин (отжиг олигонуклеотидов), 72°C - 2 мин (достройка (элонгация) цепи); финальная элонгация фрагментов при 72°C - 10 мин, хранение при 4°C.
Результаты исследования учитывают путем анализа продуктов амплификации исследуемых образцов методом электрофореза в 1% агарозном геле. В пробирки с исследуемыми образцами после завершения амплификации вносят аккуратно под масло 1/5 объема раствора 6Х DNA Loading Dye («Fermentas»), перемешивают и 8 мкл полученного образца вносят в лунки агарозного геля. Электрофорез проводят в камере для горизонтального электрофореза "SE-2" (Компания «Хеликон») с источником питания "Эльф-4" («ДНК-технология») при напряжении 120 В в течение 60 мин. Результаты электрофореза учитывают в ультрафиолетовом свете с длиной волны 254 нм на трансиллюминаторе ТСР-20 МС («Vilber Lourmat», Франция). В качестве контроля размера полученного фрагмента используют ДНК маркер GeneRuler™ 100+ п.н. («Fermentas»).
Примеры идентификации вида и штаммов B.longum по настоящему изобретению
Пример 1. Видовая идентификация филотипа В.longum
Видовую идентификацию бифидобактерий проводят путем амплификации ДНК с геномной ДНК бифидобактерий при описанных выше параметрах с использованием видоспецифических олигонуклеотидов RelBN (5′TGCGAAACTGAAGAACGAGG3′) и RelBC (5′CGCGGAACTGCTTGTAGGT3′).
При амплификации с ДНК штаммов, относящихся к филотипу В.longum, должен быть наработан фрагмент размером 275 п.н.
Пример 2. Идентификация штаммов В.longum
Для штамм-специфической идентификации проводят амплификацию с геномной ДНК с использованием олигонуклеотидов, представленых в таблице 2. ПЦР-продукты анализируют в 1% агарозном геле. Размер полученного фрагмента определяют с помощью ДНК-маркера GeneRuler™ 100+ п.н. («Fermentas»).
Исследуемый штамм идентичен штамму В.longum subsp. infantis АТСС 15697 в случае наработки фрагментов при использовании олигонуклеотидов ReB1N-ReB1C (штамм-специфические), ReB2N-ReB2C и http://ReB3.lN-ReB3.2C. При использовании остальных олигонуклеотидов не должно быть ПЦР-продуктов.
Исследуемый штамм идентичен штамму В.longum subsp. infantis АТСС 55813 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB9.1N-ReB9.1C.
Исследуемый штамм идентичен штамму B.longum subsp. infantis CCUG 52486 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов ReB3.1N-ReB3.1C, http://ReB3.4N-ReB3.4C и ReB9.1N-ReB9.2C.
Исследуемый штамм идентичен штамму В.longum DJO10A в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.lC.
Исследуемый штамм идентичен штамму Bifidobacterium sp. (B.longum) 12_1_47BFAA в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.lC и ReB4N-ReB4C.
Исследуемый штамм идентичен штамму В.longum subsp. longum F8 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов ReB2N-ReB2C и ReB3.3N-ReBC.
Исследуемый штамм идентичен штамму В.longum NCC2705 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.2C.
Исследуемый штамм идентичен штамму В.longum subsp. longum JDM301 в случае амплификации фрагментов только при использовании олигонуклеотидов ReB8N-ReB8C (штамм-специфические), ReB2N-ReB2C и http://ReB3.lN-ReB3.lC.
Исследуемый штамм идентичен штамму В.longum subsp. longum BBMN68 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.lCи http://ReB3.4N-ReB3.4C.
Исследуемый штамм идентичен штамму В.longum subsp. infantis 157F в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.lC, ReB4N-ReB4C и ReB5N-ReB5C.
Исследуемый штамм идентичен штамму B.longum subsp. longum JCM1217 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов http://ReB3.lN-ReB3.lCи http://ReB3.5N-ReB3.4C.
Исследуемый штамм идентичен штамму В.longum subsp. longum КАСС 91563 в случае наличия ПЦР-продуктов только при использовании олигонуклеотидов ReB10N-ReB10C и ReB3.1N-ReB3.4C.
Пример 3. Идентификация видов Bifidobacterium bifidum и Bifidobacterium adolescentis
Идентификацию бифидобактерий, относящихся к виду B.bifidum, проводят путем амплификации с геномной ДНК бифидобактерий при описанных выше параметрах с использованием олигонуклеотидов RelB11BbN (5′ATGGCGAGCATACCCAC3′) и RelB11BbC (5′ATCTGCCATTCGACGTTTCCTT3′).
При амплификации с ДНК штаммов, относящихся к виду B.bifidum, должен быть наработан фрагмент размером 178 п.н.
Идентификацию бифидобактерий В.adolescentis проводят путем амплификации с геномной ДНК бифидобактерий при описанных выше параметрах с использованием олигонуклеотидов MazBaN (5′GTGAGATCTGGACTGTGCT3′) и MazBaC (5′CTGGCGCATGACATCATCT3′).
При амплификации с ДНК штаммов, относящихся к виду В.adolescentis, должен быть наработан фрагмент размером 277 п.н.
Пример 4. Идентификация штаммов бифидобактерий из российской коллекции
С использованием сконструированных праймеров для генов систем токсин-антитоксин mazEF и relBE проведена идентификация 35 штаммов бифидобактерий из коллекции лаборатории генетики микроорганизмов Института общей генетики им. Н.И. Вавилова РАН, выделенных из гастроэнтерологического тракта практически здоровых людей Центрального региона России.
На первом этапе была проведена видовая идентификация. Из них 23 штамма были отнесены к филотипу B.longum, 6 штаммов - к виду В.adolescentis и 6 штаммов - к виду В.bifidum.
На втором этапе проводили штаммовую идентификацию B.longum. Исходя из композиции генов системы relBE среди 23 штаммов B.longum не обнаружено ни одного идентичного, т.е. данный подход позволяет идентифицировать штаммовую специфичность.
Способ видовой и штаммовой идентификации бифидобактерий филотипа Bifidobacterium longum, отличающийся тем, что основан на комбинации и полиморфизме генов токсин-антитоксин суперсемейства Re1BE и характеризующийся тем, что для идентификации проводят амплификацию с геномной ДНК с использованием набора видо- и штаммоспецифических олигонуклеотидов, ПЦР продукты анализируют в агарозном геле, а размер полученного фрагмента определяют с помощью ДНК-маркера, видовую идентификацию проводят с использованием специфичных олигонуклеотидов, причем исследуемые штаммы относят к виду B. longum в случае наработки целевого фрагмента размером 275 п.н.; штаммовую идентификацию проводят с использованием олигонуклеотидов, представленных в таблице 2, исследуемый штамм признают идентичным штамму B. longum subsp. infantis ATCC 15697 в случае наработки фрагментов при использовании олигонуклеотидов ReB1N-ReB1C, ReB2N-ReB2C и ReB3.1N-ReB3.2C; исследуемый штамм признают идентичным штамму В. longum subsp. infantis ATCC 55813 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB9.1N-ReB9.1C; исследуемый штамм признают идентичным штамму B. longum subsp. infantis CCUG 52486 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C, ReB3.4N-ReB3.4C и ReB9.1N-ReB9.2C; исследуемый штамм признают идентичным штамму В. longum DJO10A в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C; исследуемый штамм признают идентичным штамму Bifidobacterium sp. (B. longum) 12_1_47BFAA в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB4N-ReB4C; исследуемый штамм признают идентичным штамму B. longum subsp. longum F8 в случае наработки фрагментов при использовании олигонуклеотидов ReB2N-ReB2C и ReB3.3N-ReBC; исследуемый штамм признают идентичным штамму B. longum NCC2705 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.2C; исследуемый штамм признают идентичным штамму B. longum subsp. longum JDM301 в случае наработки фрагментов при использовании олигонуклеотидов ReB8N-ReB8C, ReB2N-ReB2C и ReB3.1N-ReB3.1C; исследуемый штамм признают идентичным штамму B. longum subsp. longum BBMN68 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB3.4N-ReB3.4C; исследуемый штамм признают идентичным штамму B. longum subsp. infantis 157F в случае наработки фрагментов только при использовании олигонуклеотидов ReB3.1N-ReB3.1C, ReB4N-ReB4C и ReB5N-ReB5C; исследуемый штамм признают идентичным штамму B. longum subsp. longum JCM 1217 в случае наработки фрагментов при использовании олигонуклеотидов ReB3.1N-ReB3.1C и ReB3.5N-ReB3.4C; исследуемый штамм признают идентичным штамму B. longum subsp. longum КАСС 91563 в случае наработки фрагментов при использовании олигонуклеотидов ReB10N-ReB10C и ReB3.1N-ReB3.4C.