КОМПОЗИЦИЯ ДЛЯ ОБЛЕГЧЕНИЯ ИЛИ ЛЕЧЕНИЯ БОЛИ
Вид РИД
Изобретение
Область техники, к которой относится изобретение
Настоящее изобретение относится к композиции для облегчения или лечения боли и к способу облегчения или лечения боли с ее применением.
Уровень техники
Боль представляет переживание фактического или потенциального повреждения ткани или беспокоящие сенсорные ощущения и эмоциональные чувства, связанные с таким повреждением. Боль защищает области тела, которые подверглись повреждению во время заживления поврежденных тканей, от возможных потенциальных ситуаций, приводящих к повреждениям, и обеспечивает мотивацию, чтобы избежать подобных переживаний в будущем. Боль в значительной степени, постепенно ослабляется, когда устраняется причинный раздражитель, но иногда боль сохраняется, даже когда ткани зажили по мере исчезновения раздражителя, и повреждение явно зажило, или боль возникает в состоянии без наличия какого-либо раздражения, повреждения или заболевания.
Для лечения боли в основном широко применяются наркотические анальгетики, такие как морфин, который является алкалоидом опия, или ненаркотические анальгетики, такие как нестероидные противовоспалительные препараты (НПВП), содержащие активный ингредиент из ацетилсалициловой кислоты, ибупрофена или ацетаминофена.
Наркотические анальгетики обладают преимуществом, заключающимся в том, что они проявляют зависимость доза-эффект и высокую эффективность, но они могут привести к побочным эффектам со стороны нервной системы, и при длительном применении они могут привести к резистентности и физической зависимости, и боль может усилиться.
Если аспирин, ненаркотический анальгетик, имеющий в качестве активного ингредиента ацетилсалициловую кислоту, используется для обеспечения анальгетического действия, то его следует вводить в высокой дозе, по меньшей мере, 500 мг. Однако аспирин представляет собой нестероидный противовоспалительный анальгетик, блокирующий фермент циклооксигеназу-1 (COX-1), который способствует выработке простагландинов, которые защищают желудок, тем самым предотвращая нарушение слизистой оболочки желудка. Следовательно, желудок может быть легко поврежден желудочной кислотой и может возникнуть желудочно-кишечное кровотечение. Кроме того, ибупрофен также является нестероидным противовоспалительным анальгетиком, который может вызывать желудочные расстройства. Также в случае анальгетиков, содержащих ацетаминофен в качестве активного ингредиента, таких как тиленол, ацетаминофен в основном метаболизируется в печени, и может быть индуцировано повреждение печени.
Даже если вышеуказанные анальгетики эффективны на ранней стадии, то часто при длительном применении они становятся неэффективными за счет резистентности. В частности, в случае нейропатической боли существует проблема, заключающаяся в том, что боль не отвечает на максимальную дозу нестероидного противовоспалительного средства и, таким образом, его требуется вводить в высокой дозе в течение короткого периода времени.
Недавно были разработаны новые терапевтические агенты для лечения нейропатической боли, но они по-прежнему имеют побочные эффекты. Например, блокаторы натриевых каналов в основном находятся в форме небольших молекул и проявляют низкую селективность в отношении изоформ белков. Кроме того, они проявляют побочные эффекты, такие как токсичность для сердца и двигательные расстройства.
Следовательно, существует настоятельная необходимость в разработке нового анальгетика для нейропатической боли, который обладает высокой анальгетической эффективностью при одновременном снижении проявления побочных эффектов.
Техническая проблема
Следовательно, заявители настоящего изобретения попытались разработать новый анальгетик для нейропатической боли, проявляющий высокую анальгетическую эффективность даже в низких дозировках. В результате заявители настоящего изобретения обнаружили, что при использовании комбинации двух или более генов глутаматдекарбоксилазы, противовоспалительного цитокина и глиального нейротрофического фактора, можно значительно облегчить или лечить боль по сравнению с их применением по отдельности и завершили настоящее изобретение.
Решение проблемы
Настоящее изобретение обеспечивает фармацевтическую композицию для облегчения или лечения боли, содержащую два или более генов, выбранных из группы, состоящей из гена, кодирующего глутаматдекарбоксилазу (GAD), гена, кодирующего интерлейкин-10 (IL-10), и гена, кодирующего глиальный нейротрофический фактор (GDNF).
Кроме того, настоящее изобретение обеспечивает способ облегчения или лечения боли, включающий введение фармацевтической композиции по настоящему изобретению.
Преимущественные эффекты изобретения
Фармацевтическая композиция по настоящему изобретению включает два или более генов, выбранных из группы, состоящей из генов, кодирующих GAD, IL-10 и GDNF. Следовательно, фармацевтическая композиция по настоящему изобретению проявляет высокую анальгетическую эффективность в дозировке ниже, чем при введении по отдельности, когда гены вводятся совместно, и, таким образом, обычные побочные эффекты и токсичность могут быть снижены. Следовательно, фармацевтическая композиция по настоящему изобретению может быть пригодна для облегчения или лечения боли.
Краткое описание фигур
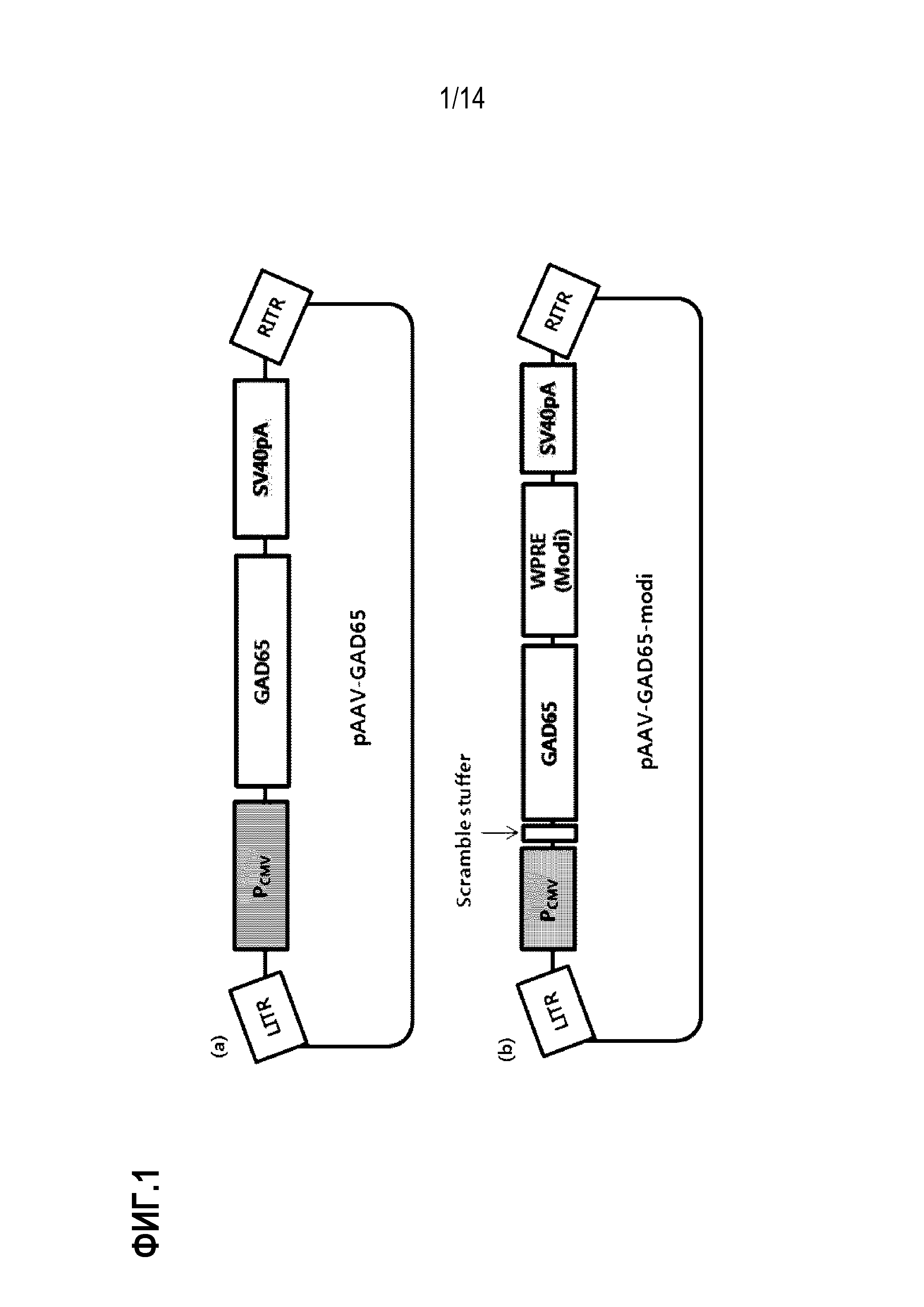
На фиг. 1 представлено схематическое изображение плазмид pAAV-GAD65 и pAAV-GAD65-modi, использованных для конструирования рекомбинантного аденоассоциированного вируса:
(а) представлено схематическое изображение pAAV-GAD65 и (b) представлено схематическое изображение pAAV-GAD65-modi.
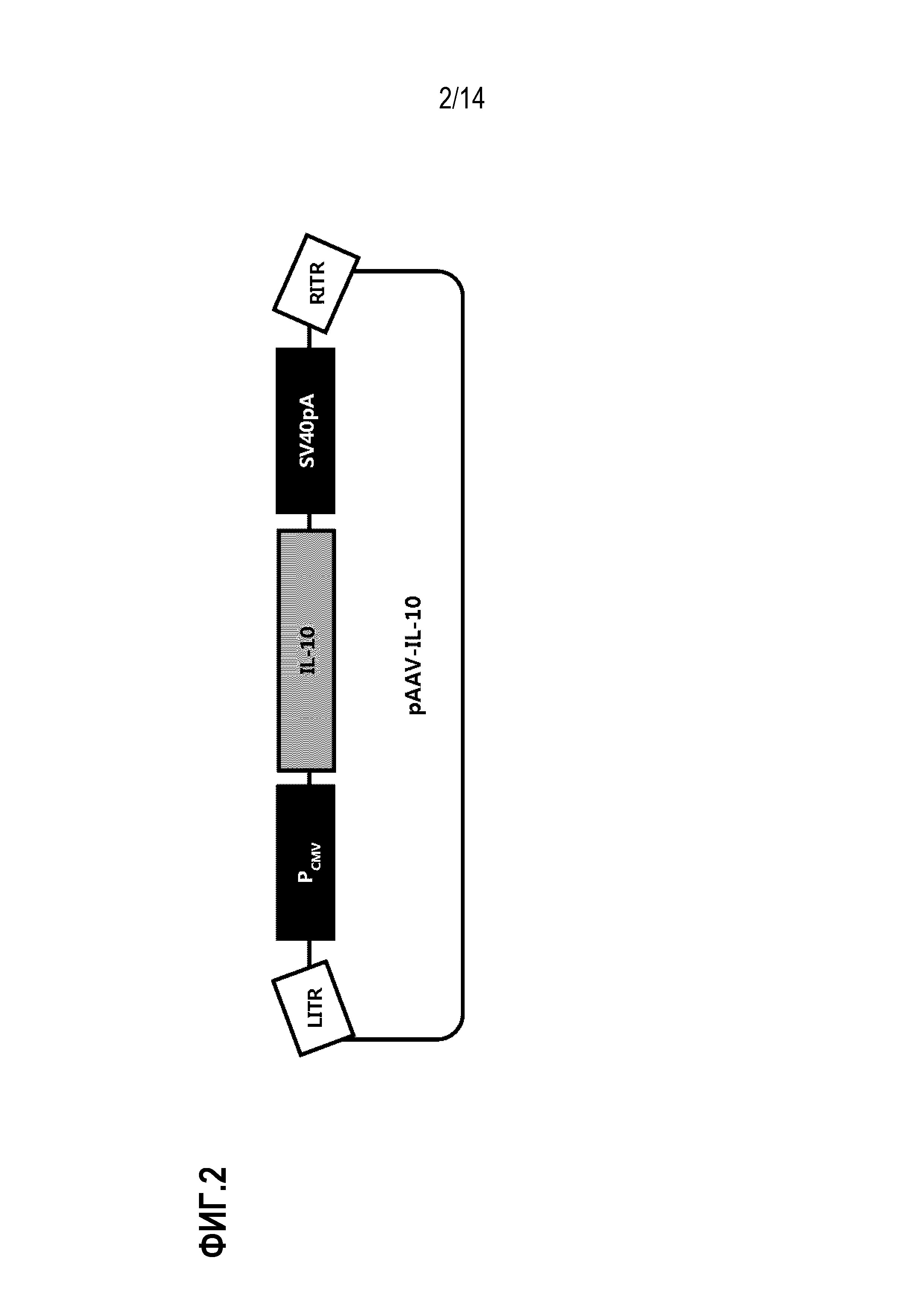
На фиг.2 представлено схематическое изображение плазмиды pAAV-IL-10, использованной для конструирования рекомбинантного аденоассоциированного вируса.
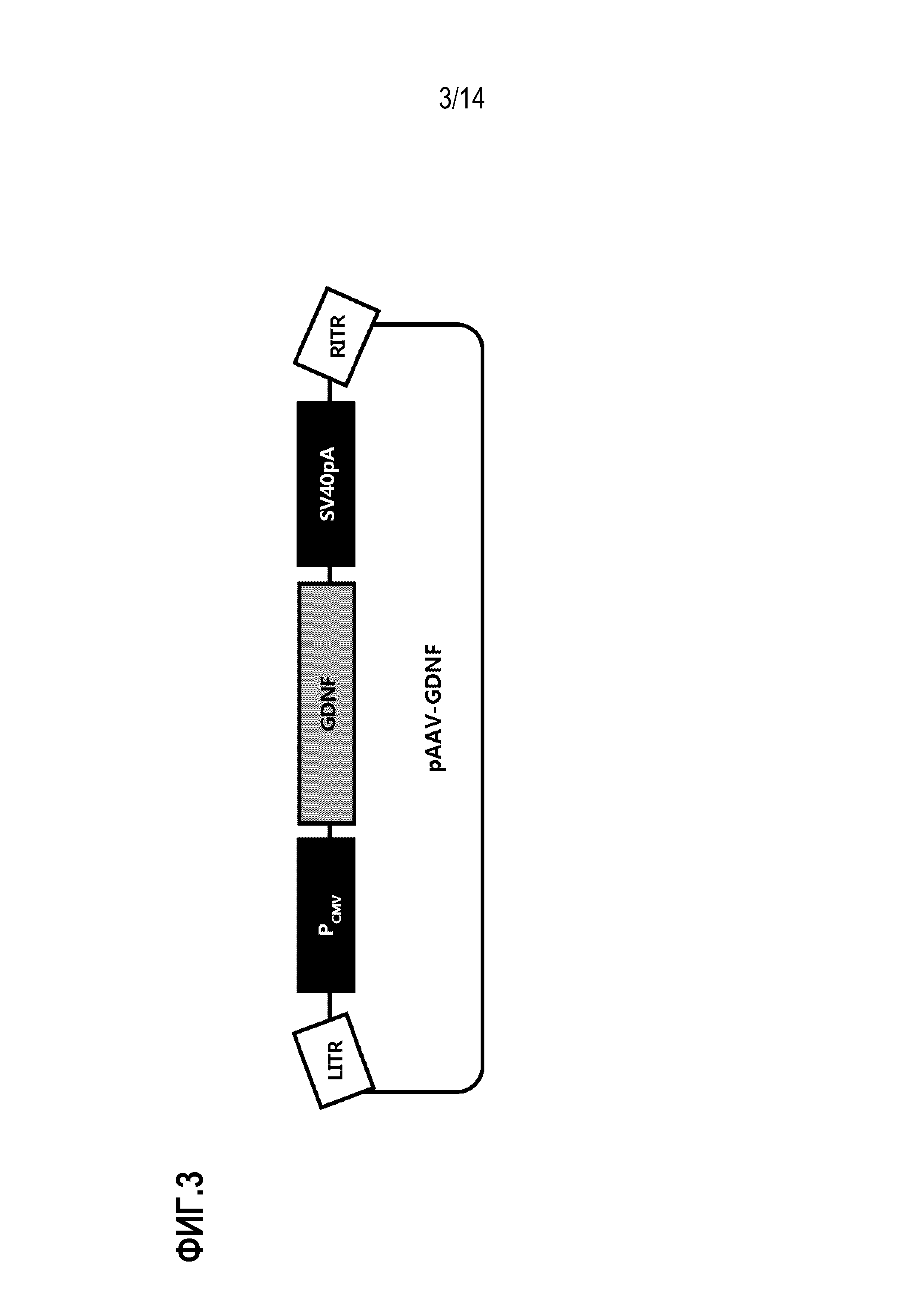
На фиг.3 представлено схематическое изображение плазмиды pAAV-GDNF, использованной для конструирования рекомбинантного аденоассоциированного вируса.
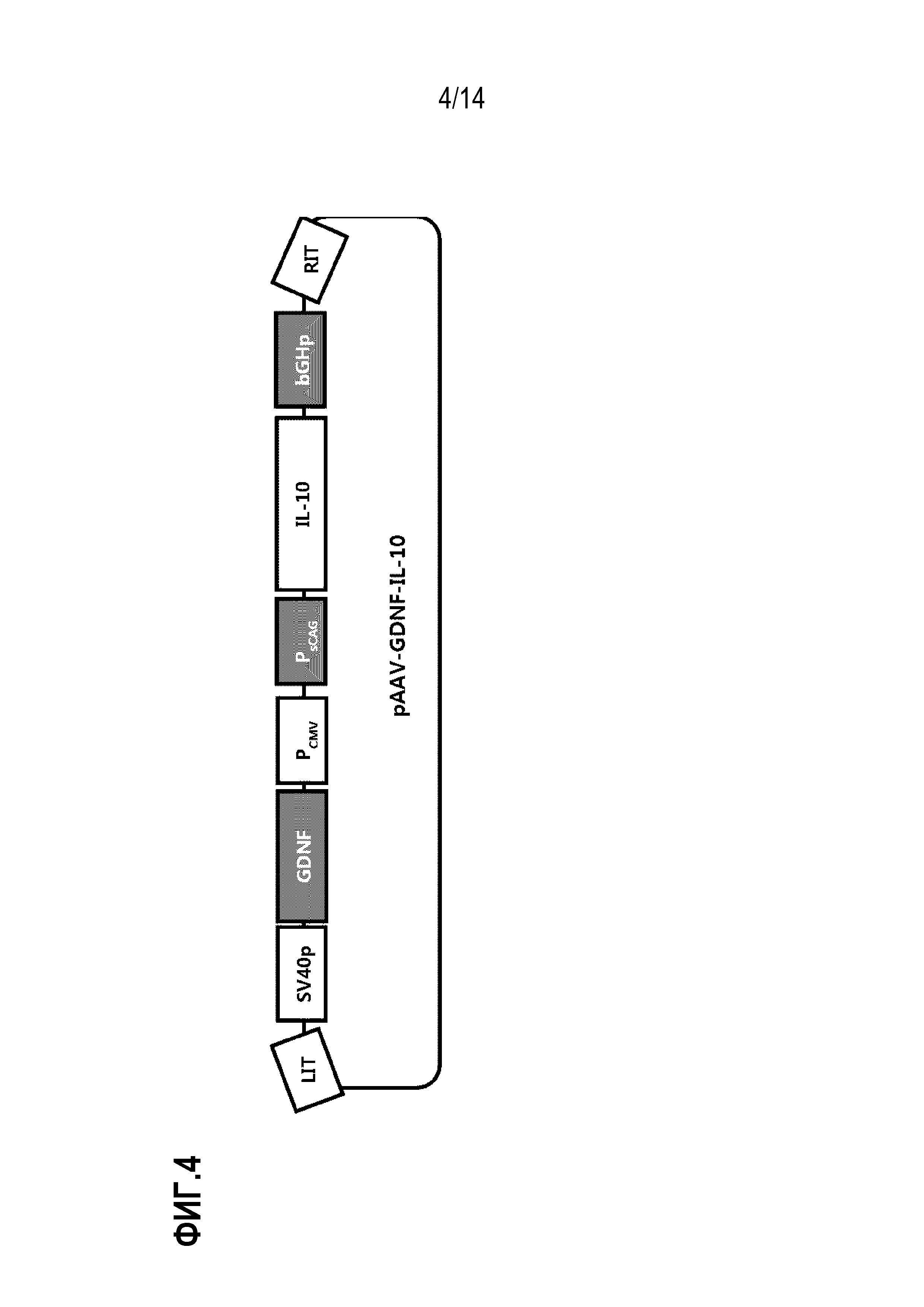
На фиг.4 представлено схематическое изображение, показывающая плазмиду pAAV-GDNF-IL-10.
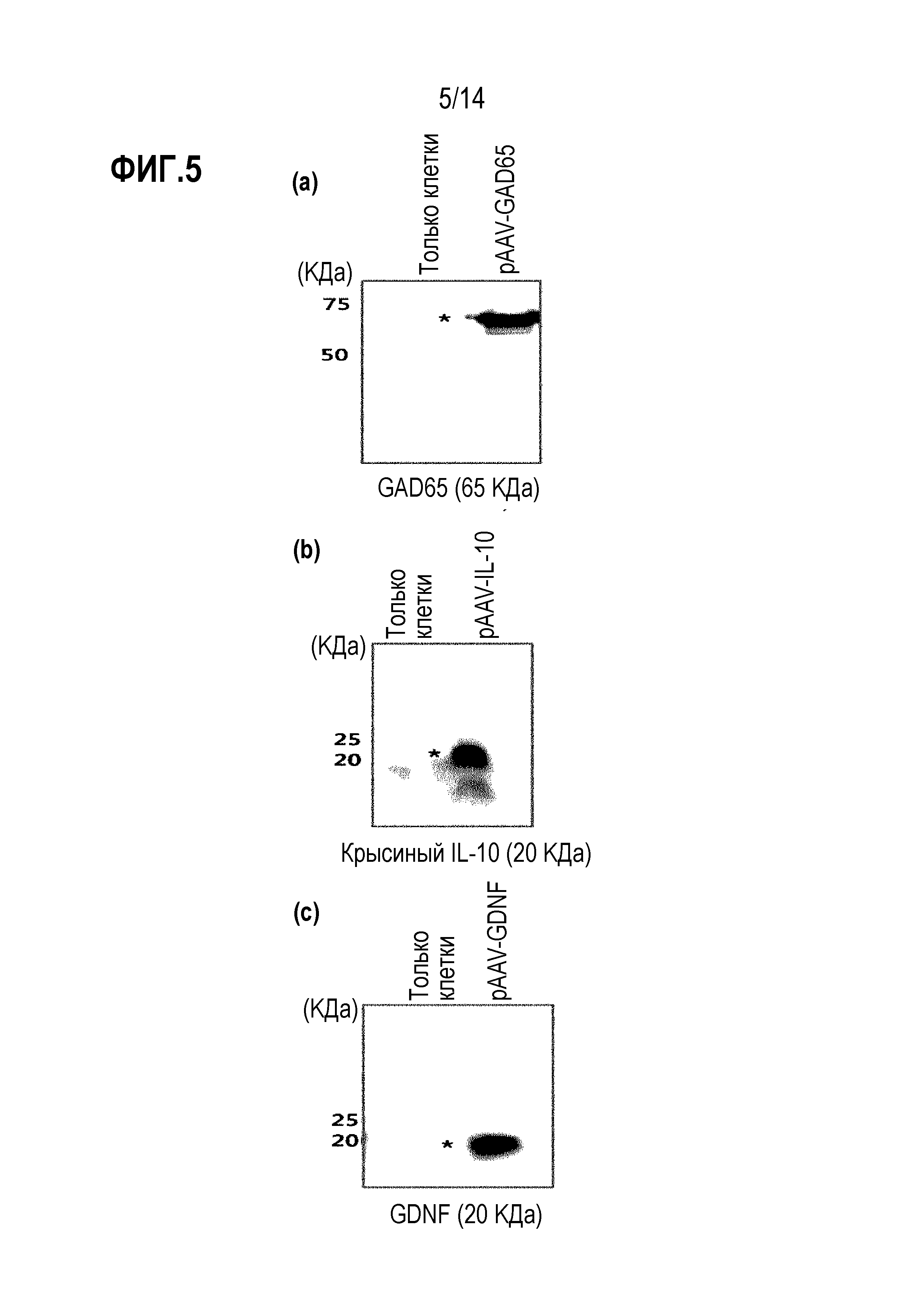
На фиг.5 показана экспрессия каждого введенного гена плазмидой pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF:
(а) показана экспрессия GAD65 плазмидой pAAV-GAD65; (b) показана экспрессия IL-10 плазмидой pAAV-IL-10; и (с) показана экспрессия GDNF плазмидой pAAV-GDNF.
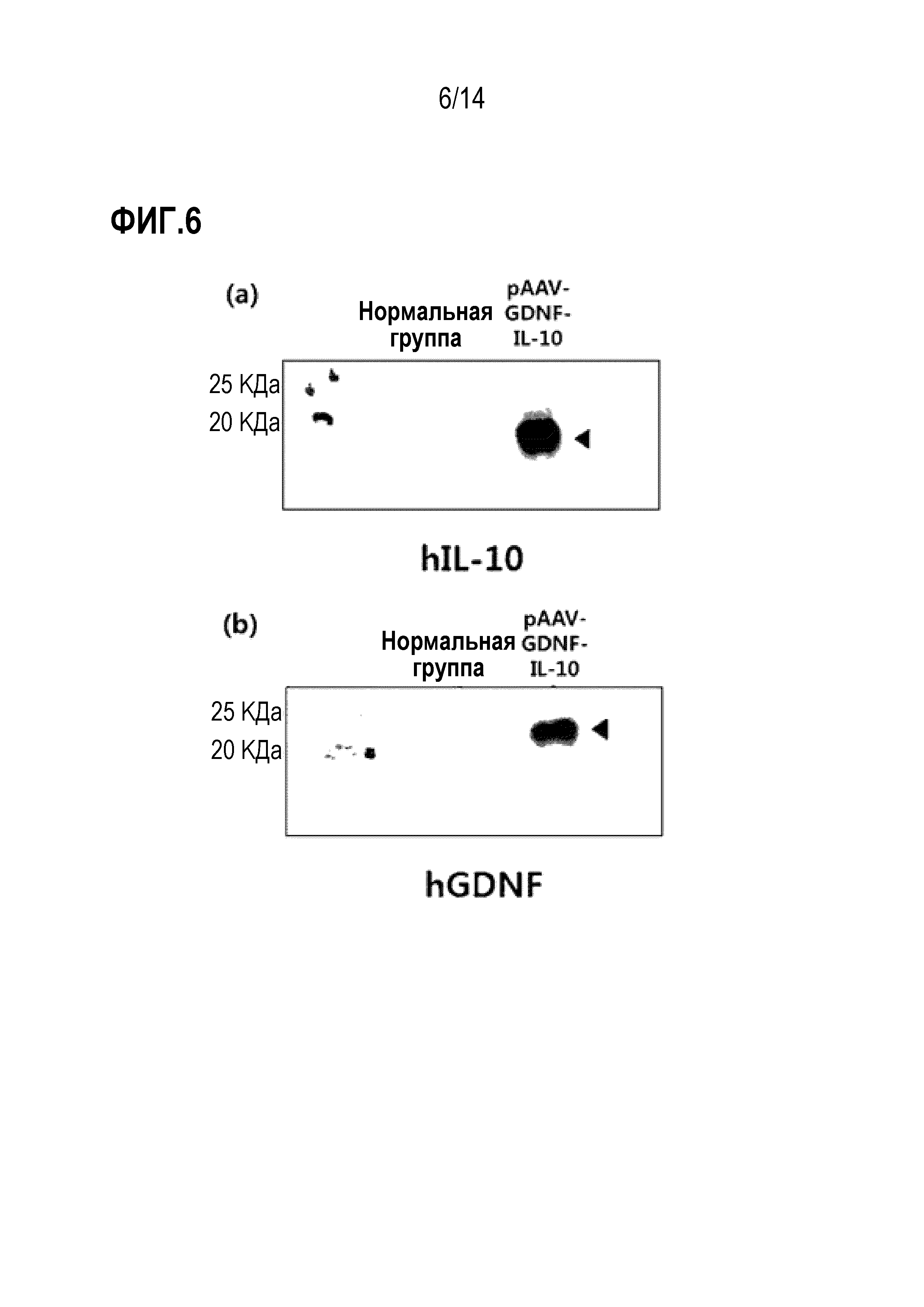
На фиг.6 показана экспрессия гена GDNF и гена IL-10 плазмидой pAAV-GDNF-IL-10:
(а) показана экспрессия IL-10 плазмидой pAAV-GDNF-IL-10; и (b) показана экспрессия GDNF плазмидой pAAV-GDNF-IL-10.
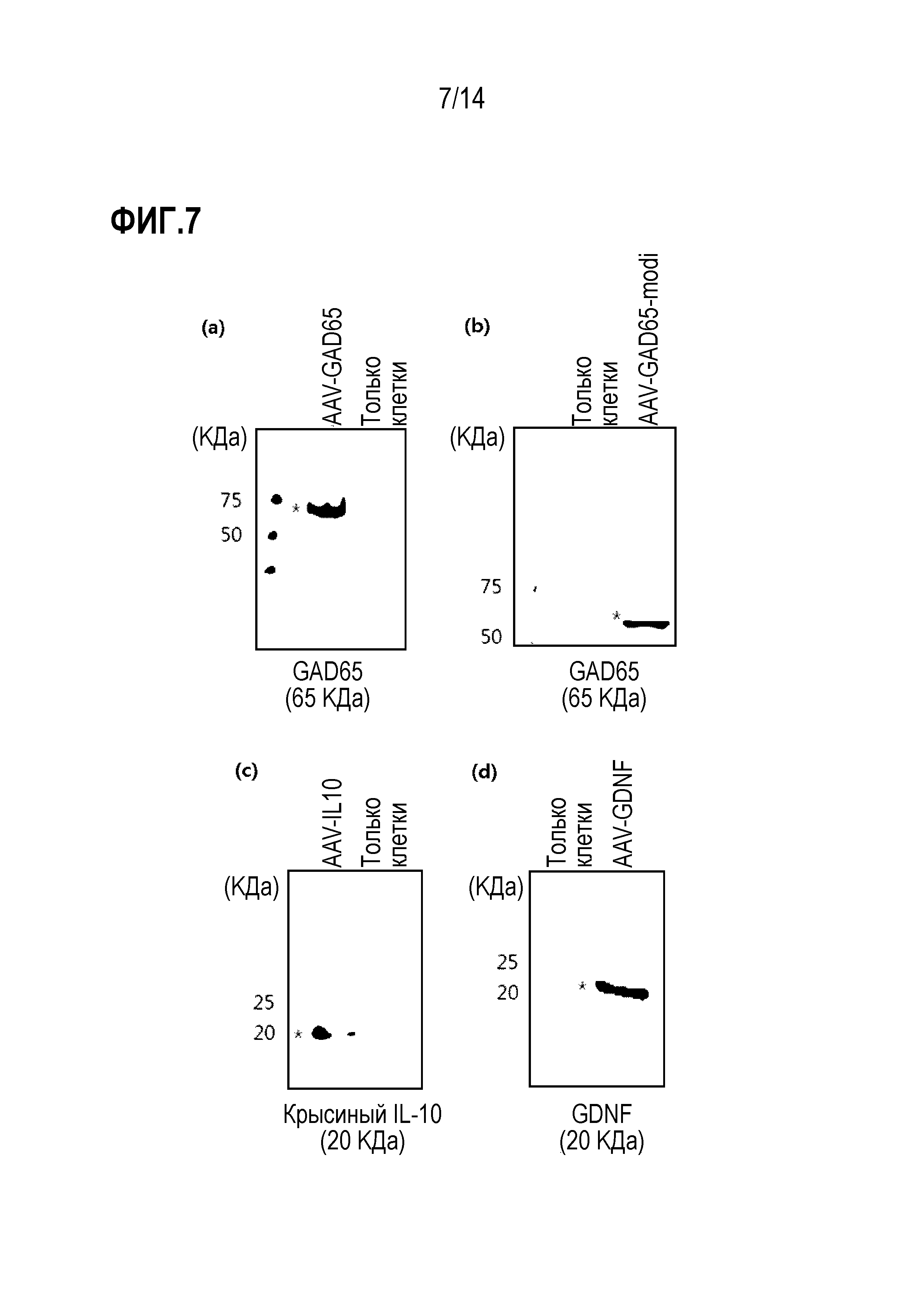
На фиг.7 представлены результаты вестерн-блоттинга, показывающие экспрессию каждого белка после обработки клеток 293T или HeLa каждым рекомбинантным аденоассоциированным вирусом после конструирования рекомбинантных аденоассоциированных вирусов, в которые были вставлены ген GAD65, ген IL-10 и ген GDNF соответственно:
(а) показана экспрессия GAD65 после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GAD65; (b) показана экспрессия GAD65 после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GAD65-modi; (c) показана экспрессия IL-10 после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-IL-10; и (d) показана экспрессия GDNF после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GDNF.
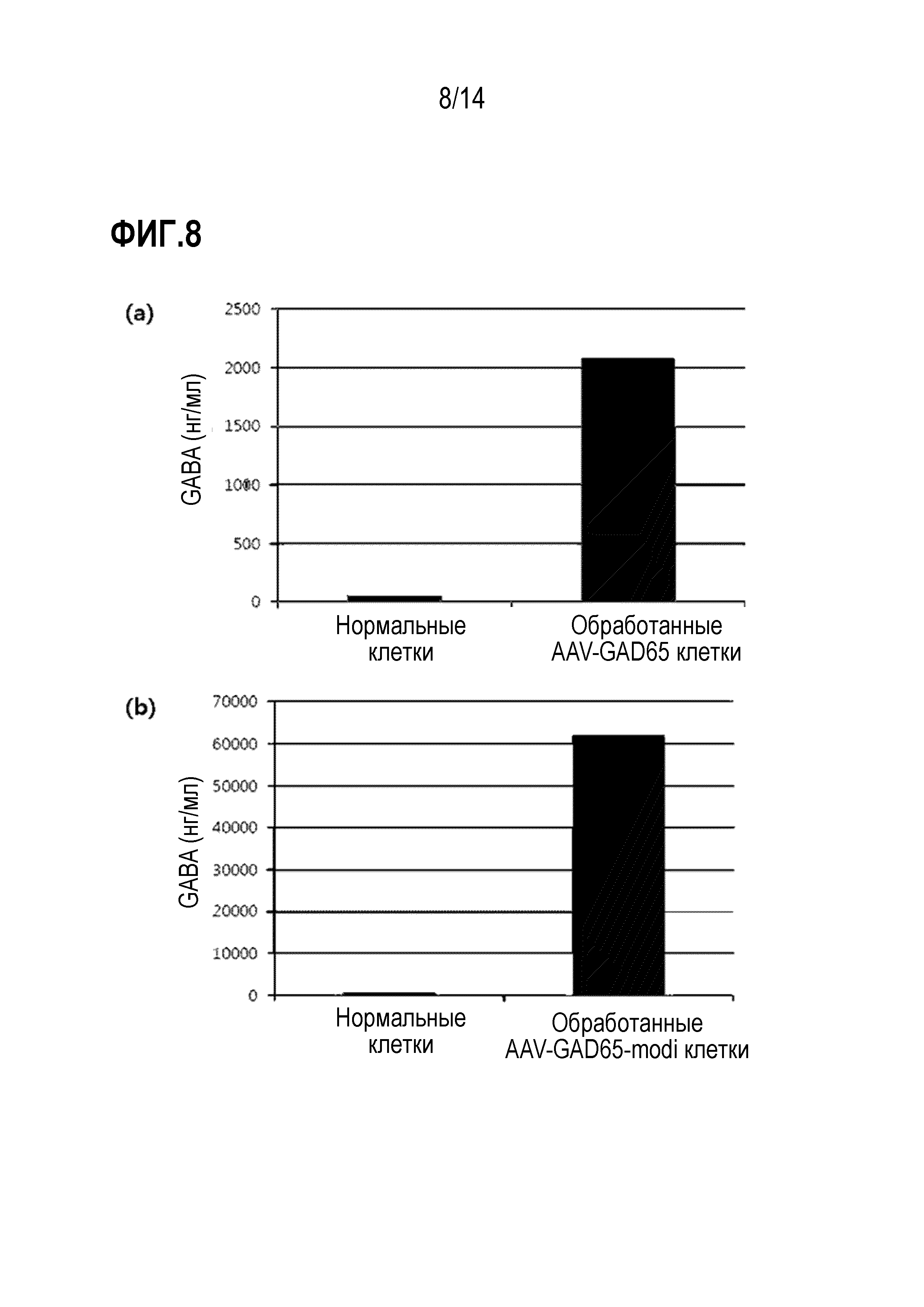
На фиг.8 показаны уровни экспрессии GABA, измеренные с использованием ELISA после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GAD65 или AAV-GAD65-modi:
(а) представлен график, показывающий уровень экспрессии GABA после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GAD65; и (b) представлен график, показывающий уровень экспрессии GABA после обработки клеток 293T или HeLa рекомбинантным аденоассоциированным вирусом AAV-GAD65-modi.
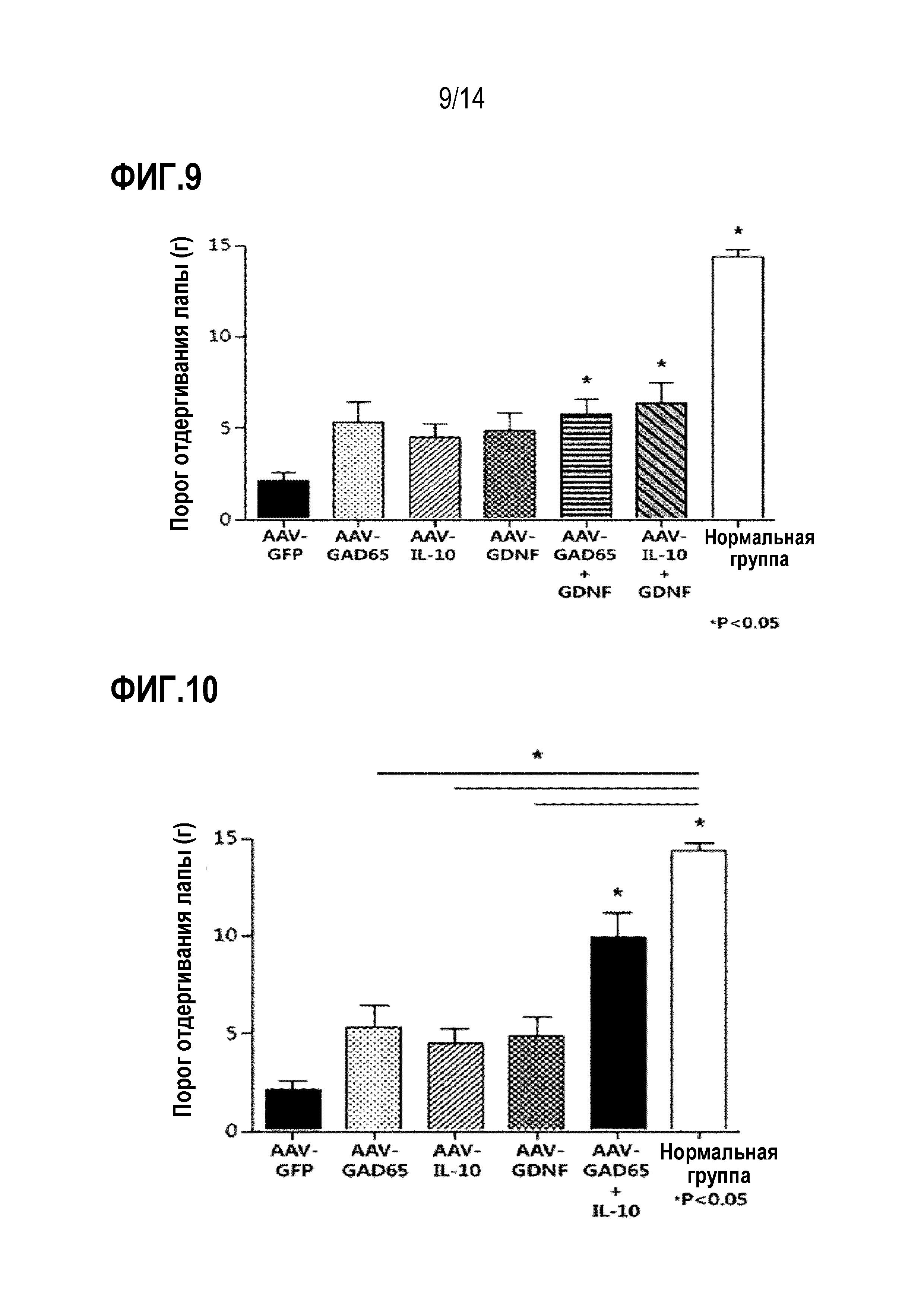
На фиг.9 представлен график, показывающий результаты сравнения анальгетической эффективности при введении вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности и совместном введении вирусов AAV-GAD65 и AAV-GDNF или вирусов AAV-IL-10 и AAV-GDNF.
На фиг.10 представлен график, показывающий результаты сравнения анальгетической эффективности при введении вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности и совместном введении вирусов AAV-GAD65 и AAV-IL-10.
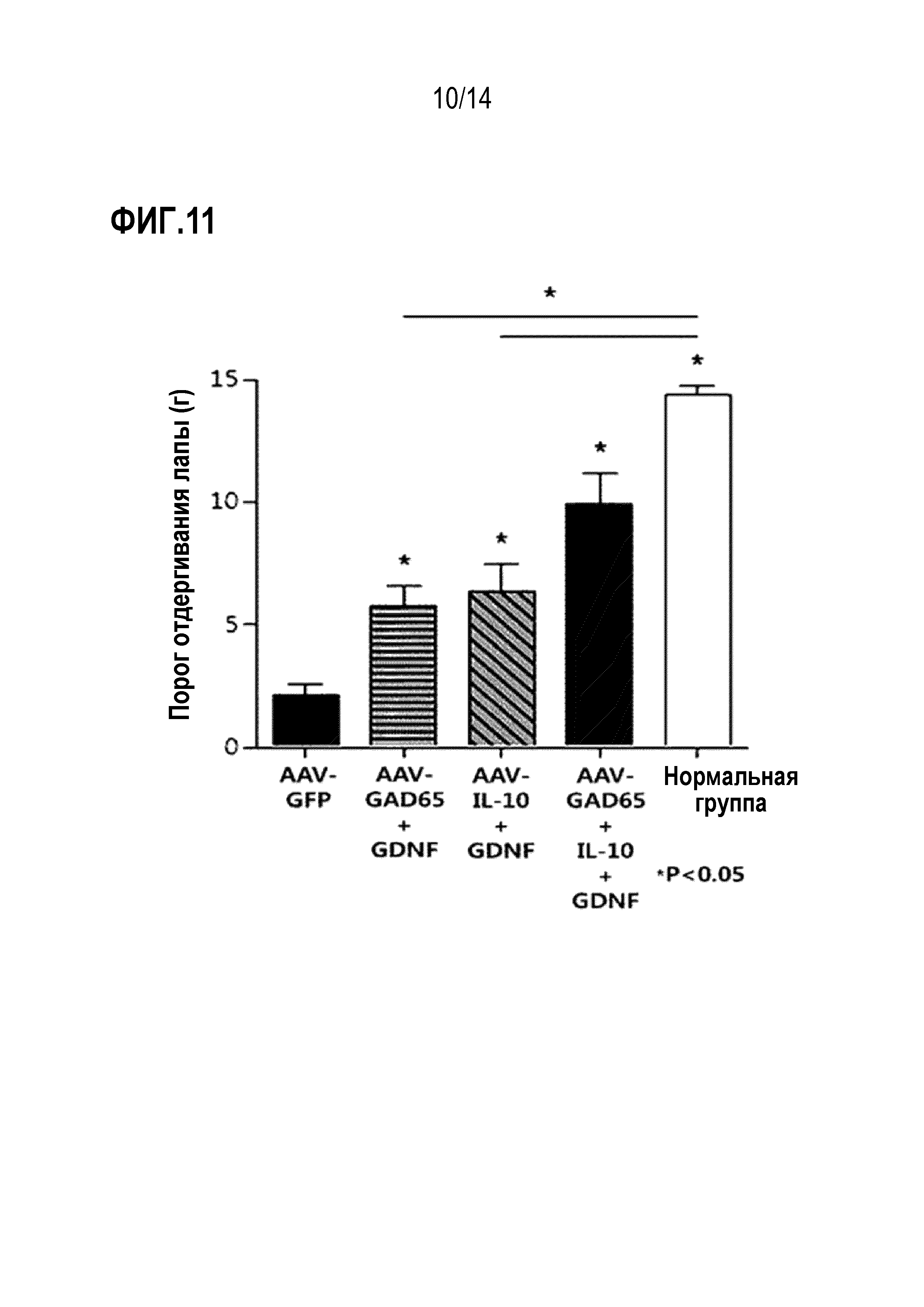
На фиг. 11 представлен график, показывающий результаты сравнения анальгетической эффективности при совместном введении вирусов AAV-GAD65 и AAV-GDNF или вирусов AAV-IL-10 и AAV-GDNF и совместном введении всех вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF.
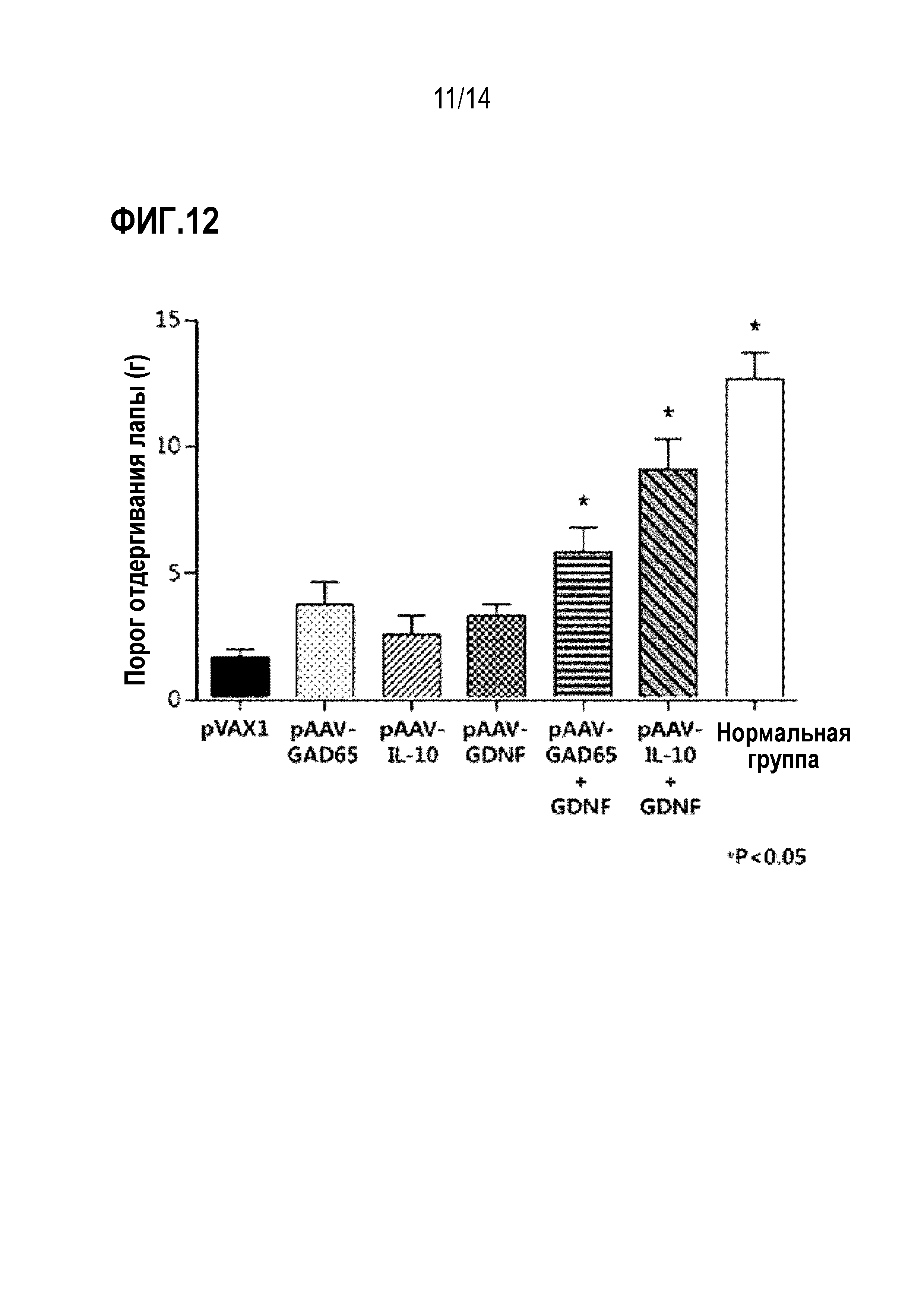
На фиг. 12 представлен график, показывающий результаты сравнения анальгетической эффективности при введении плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности и совместном введении плазмид pAAV-GAD65 и pAAV-GDNF или плазмид pAAV-IL-10 и pAAV-GDNF.
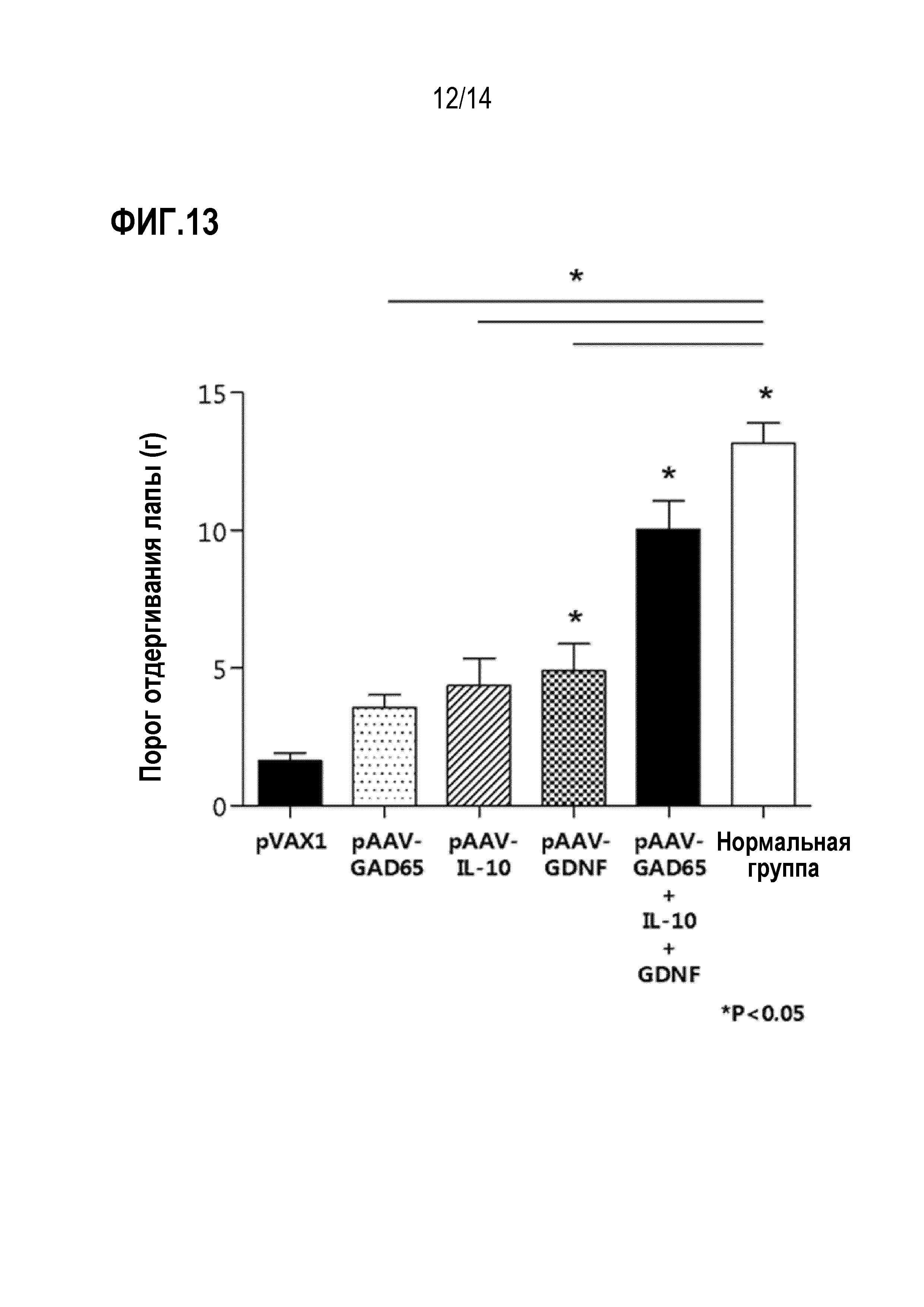
На фиг. 13 представлен график, показывающий результаты сравнения анальгетической эффективности при введении плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности и совместном введении всех плазмид pAAV-GAD65, pAAV-IL-10 и pAAV-GDNF.
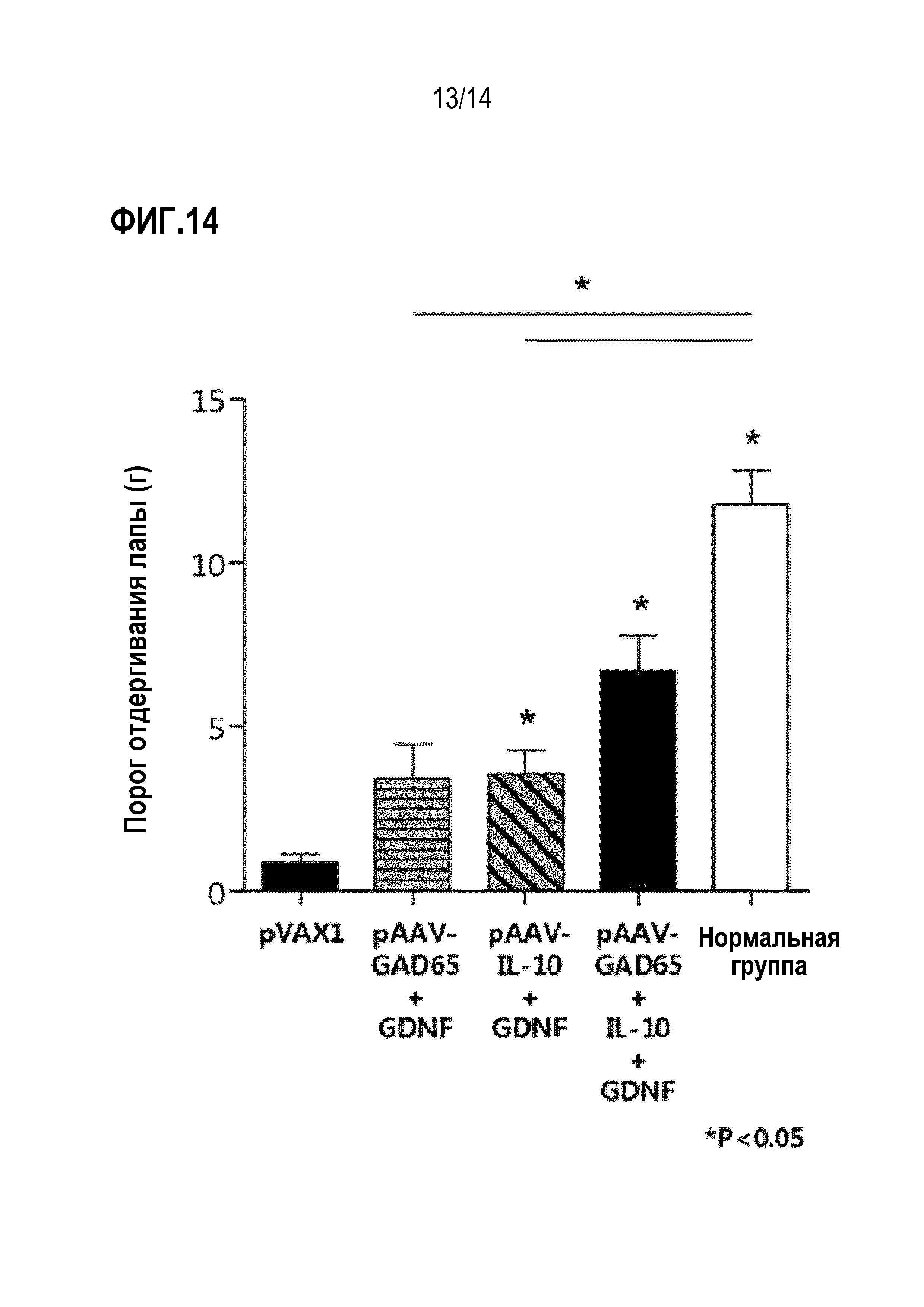
На фиг.14 представлен график, показывающий результаты сравнения анальгетической эффективности при совместном введении плазмид pAAV-GAD65 и pAAV-GDNF или плазмид pAAV-IL-10 и pAAV-GDNF и совместном введении всех плазмид pAAV-GAD65, pAAV-IL-10 и pAAV-GDNF.
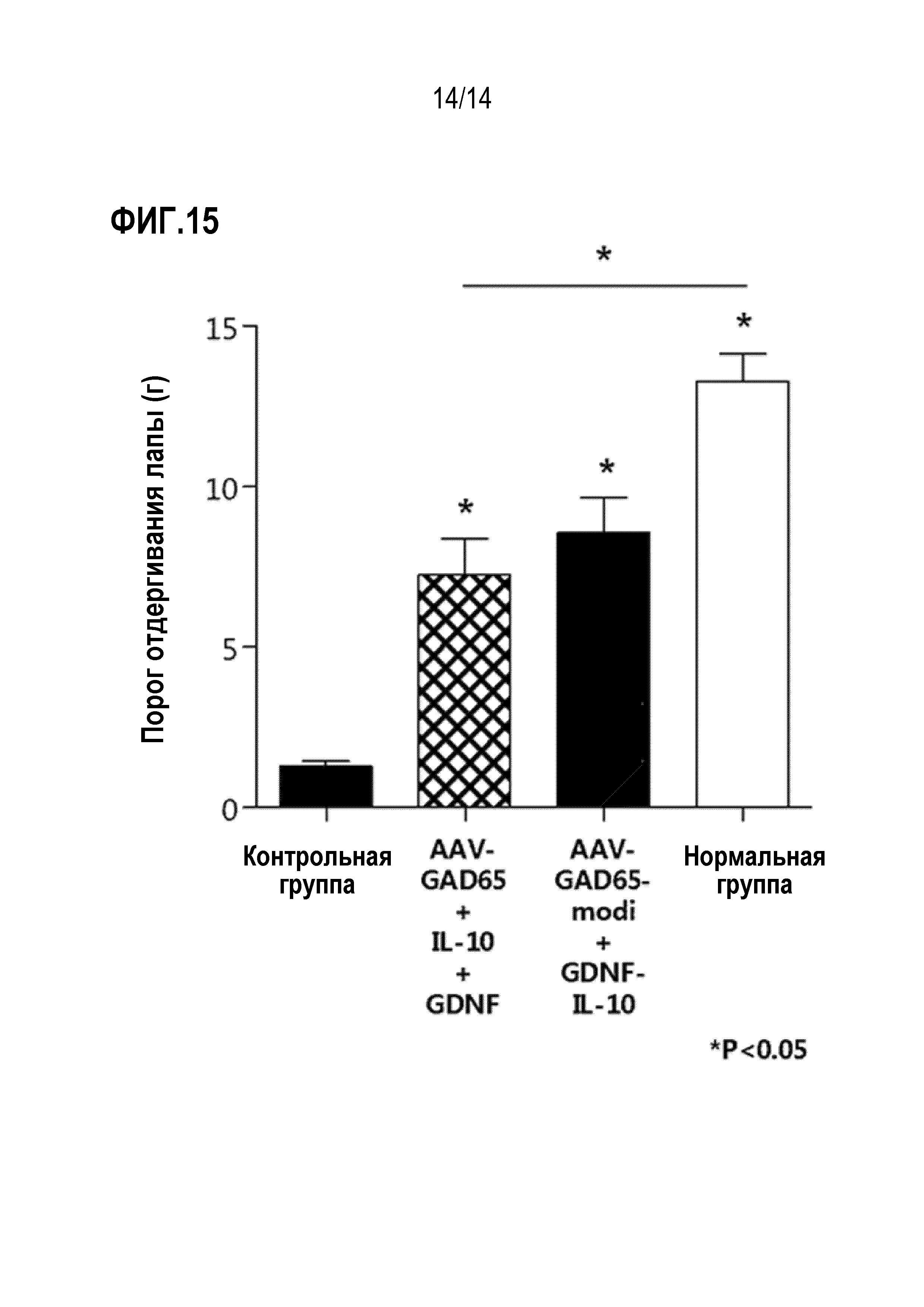
На фиг. 15 представлен график, показывающий результаты сравнения анальгетической эффективности при совместном введении вирусов AAV-GAD65-modi и AAV-GDNF-IL-10 и совместном введении всех вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF.
Наилучший способ осуществления изобретения
Далее настоящее изобретение будет описано подробно.
Настоящее изобретение обеспечивает фармацевтическую композицию для облегчения или лечения боли, содержащую два или более генов, выбранных из группы, состоящей из гена, кодирующего глутаматдекарбоксилазу (GAD), гена, кодирующего интерлейкин-10 (IL-10), и гена, кодирующего глиальный нейротрофический фактор (GDNF).
В одном варианте осуществления комбинация двух или более генов может представлять гены GAD и IL-10, GAD и GDNF, IL-10 и GDNF или GAD, IL-10 и GDNF.
Два или более генов, выбранных из группы, состоящей из гена, кодирующего GAD, гена, кодирующего IL-10, и гена, кодирующего GDNF, могут находиться в форме, будучи включенными в носитель. Здесь носителем может быть вирусный вектор или невирусный вектор, такой как плазмида, липосома и т. д. Кроме того, гены могут быть в форме, в которой некоторые из генов находятся в вирусном векторе, и остальные гены находятся в невирусном векторе.
В одном варианте осуществления гены могут быть в форме, в которой ген GAD находится в вирусном векторе, и ген IL-10 находится в невирусном векторе. Кроме того, гены могут быть в форме, в которой ген GAD находится в вирусном векторе, и ген GDNF находится в невирусном векторе. Кроме того, гены могут быть в форме, в которой ген IL-10 находится в вирусном векторе, и ген GDNF находится в невирусном векторе. Кроме того, гены могут быть в форме, в которой ген GAD находится в вирусном векторе, и гены IL-10 и GDNF находятся в невирусном векторе. Кроме того, гены могут быть в форме, в которой гены GAD и IL-10 находятся в вирусном векторе, и ген GDNF находится в невирусном векторе. Кроме того, гены могут быть в форме, в которой гены GAD и GDNF находятся в вирусном векторе, и ген IL-10 находится в невирусном векторе. Кроме того, гены могут быть в форме, в которой ген IL-10 находится в вирусном векторе, и гены GAD и GDNF находятся в невирусном векторе. Кроме того, гены могут быть в форме, в которой гены IL-10 и GDNF находятся в вирусном векторе, и ген GAD находится в невирусном векторе. Также гены могут быть в форме, в которой ген GDNF находится в вирусном векторе, и гены GAD и IL-10 находятся в невирусном векторе.
Кроме того, ген может быть в форме, будучи операбельно связанным в векторе. В частности, ген может быть в форме, будучи операбельно связанным в вирусном векторе или невирусном векторе.
Вирусный вектор может представлять, по меньшей мере, один, выбранный из группы, состоящей из вектора на основе аденовируса, аденоассоциированного вируса (AAV), вируса простого герпеса, лентивируса, ретровируса, цитомегаловируса, бакуловируса, поксвируса и т.д. В частности, вирусный вектор может быть на основе аденоассоциированного вируса.
В одном варианте осуществления ген, кодирующий GAD, может находиться операбельно связанным в носителе 1 (например, первом векторе), и ген, кодирующий IL-10, может находиться операбельно связанным в носителе 2 (например, втором векторе), и ген, кодирующий GDNF, может находиться операбельно связанным в носителе 3 (например, в третьем векторе). Кроме того, один носитель может содержать два или более генов.
Невирусный вектор может представлять, по меньшей мере, один, выбранный из группы, состоящей из плазмиды, липосомы, катионного полимера, мицеллы, эмульсии и твердых липидных наночастиц.
Как здесь используется, термин «плазмида» относится к циклическому фрагменту ДНК, существующему отдельно от хромосомы бактерий. Плазмиды не имеют генов, необходимых для выживания бактерий, но содержат гены, необходимые для резистентности к некоторым антибиотикам и для обмена генами между бактериями. Кроме того, плазмиды могут реплицироваться независимо от хромосом и содержать селектируемые маркеры.
Как здесь используется, термин «липосома» относится к небольшой везикуле, полученной образованием бислоя за счет наличия гидрофильной части и гидрофобной части, когда молекула, имеющая как гидрофобную часть, так и гидрофильную часть, такая как фосфолипид, суспендируется в водном растворе. Липосома отделена от внешней оболочки мембраной, состоящей из липидного бислоя, и липосомы, содержащие ДНК, мРНК и т. д., можно использовать в качестве переносчиков генетической информации.
Как здесь используется, термин «катионный полимер» относится к катионному липиду или полимерному соединению, которое представляет собой вещество, образующее комплекс посредством ионной связи с анионной ДНК и доставляет ДНК в клетку.
Как здесь используется, термин «мицелла» относится к термодинамически стабильному коллоидному агрегату, образованному из молекул, состоящих из полярной группы и неполярной гидрофобной группы, таких как поверхностно-активные вещества и молекулы липидов, посредством ассоциации с помощью ван-дер-ваальсовых сил или тому подобное в растворе. Кроме того, мицеллы, содержащие ДНК, мРНК и тому подобное, можно использовать в качестве переносчиков генетической информации.
Как здесь используется, термин «эмульсия» означает, что, когда два раствора разных фаз смешиваются, то одна жидкость образует мелкие частицы и диспергируется в другой жидкости. ДНК, мРНК и тому подобное могут находиться в центре частицы в эмульсии, которую можно использовать в качестве переносчиков генетической информации.
Как здесь используется, термин «твердая липидная наночастица» относится к препарату в форме, в которой лекарственное средство содержится в микрочастице наноразмера, полученной из твердого липида вместо жидкого липида.
Носитель 1 (например, первый вектор), содержащий любой один ген, выбранный из группы, состоящей из генов GAD, IL-10 и GDNF, и носитель 2 (например, второй вектор), содержащий любой один ген, выбранный из группы оставшихся генов, не включенных в носитель 1 по настоящему изобретению, может иметь соотношение смешивания на основе титра вируса на единицу объема от 1:1 до 100 или от 1 до 100:1. В частности, соотношение смешивания на основе титра вируса на единицу объема носителя 1 и носителя 2 может составлять от 1:1 до 10 или от 1 до 10:1.
Носитель 1 (например, первый вектор), содержащий ген, кодирующий GAD, носитель 2 (например, второй вектор), содержащий ген, кодирующий IL-10, и носитель 3 (например, третий вектор), содержащий ген, кодирующий GDNF, по настоящему изобретению могут иметь соотношение смешивания на основе титра вируса на единицу объема от 1:0,1 до 10:0,1 до 10.
Как здесь используется, термин «операбельно связанный» означает, что введенный ген связан с регуляторной последовательностью таким образом, что экспрессия может иметь место в клетке-хозяине. Регуляторная последовательность представляет собой последовательность ДНК, которая регулирует экспрессию гена, и может включать разные регуляторные элементы, такие как промоторы и энхансеры, или сигнал полиаденилирования. Кроме того, регуляторная последовательность обеспечивает сайт связывания транскрипционного фактора, который контролирует экспрессию введенного гена, и может влиять на сложную структуру транскрипционным фактором для определения функции транскрипционного фактора.
Как здесь используется, термин «GAD» относится к ферменту, который катализирует декарбоксилирование глутамата с образованием GABA (гамма-аминомасляной кислоты). GAD может представлять GAD65 или GAD67. В частности, GAD65 может происходить от человека, крысы, собаки, кошки или лошади, не ограничиваясь этим. Ген, кодирующий GAD, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, показанную в SEQ ID NO: 1, 4, 32, 34 или 36.
Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 1, может представлять последовательность ДНК, показанную в SEQ ID NO: 2 или 3, и может быть последовательностью мРНК, показанной в NCBI референсной последовательности NCBI:NM_000818.2. Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 4, может представлять нуклеотидную последовательность, которая была кодон-оптимизирована, чтобы подходила для последовательности ДНК, показанной в SEQ ID NO: 5, или гена, кодирующего аминокислотную последовательность, показанную в SEQ ID NO: 4, который может быть последовательностью мРНК, показанной в NCBI референсной последовательности NCBI:NM_000817.2. Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 32, может представлять последовательность ДНК, показанную в SEQ ID NO: 33. Нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 34, может представлять последовательность ДНК, показанную в SEQ ID NO: 35, и нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 36, может представлять последовательность ДНК, показанную в SEQ ID NO: 37.
Кроме того, ген, кодирующий GAD, может представлять нуклеотидную последовательность, кодирующую вариант GAD, который может сохранять активность GAD и продуцировать GABA. Вариант GAD включает все последовательности, которые сохраняют характеристики GAD в отношении продукции GABA. Несмотря на то, что она не ограничивается какой-либо одной последовательностью, нуклеотидная последовательность, кодирующая вариант GAD, предпочтительно может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, или 90% или более, с аминокислотной последовательностью GAD, описанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности на уровне 95% или более.
Кроме того, нуклеотидная последовательность, кодирующая вариант GAD, может представлять нуклеотидную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, или 90% или более с нуклеотидной последовательностью GAD, описанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, имеющую гомологию последовательности на уровне 95% или более.
Термин «% гомологии последовательностей» определяется сравнением областей сравнения в состоянии, в котором две последовательности оптимально выровнены. Кроме того, некоторые из нуклеотидных последовательностей в областях сравнения могут включать добавления или делеции (т. е. гэпы) относительно референсной последовательности (без добавления или делеции) для оптимального выравнивания двух последовательностей.
Как здесь используется, термин «IL-10» относится к противовоспалительному цитокину, относящемуся к цитокину II класса (Renauld, Nat. Rev. Immunol., 2003). IL-10 находится в форме гомодимера, состоящего из двух субъединиц, каждая из которых имеет длину из 178 аминокислот. Он также известен как фактор, ингибирующий синтез цитокинов (CSIF) у человека. IL-10 выполняет функцию ингибирования активности NK-клеток (естественных киллеров) в иммунном ответе и образует комплекс с рецептором IL-10, для участия в трансдукции сигналов. IL-10 может представлять белок, происходящий от человека, крысы, собаки, кошки или лошади, не ограничиваясь этим. Ген, кодирующий IL-10, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, показанную в SEQ ID NO: 6, 9, 38, 40 или 42.
В частности, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 6, может представлять последовательность ДНК, показанную в SEQ ID NO: 7 или 8, и может быть последовательностью мРНК, показанной в NCBI референсной последовательности NCBI:NM_012854.2. Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 9, может представлять последовательность ДНК, показанную в SEQ ID NO: 10 или 14, и может быть последовательностью мРНК, показанной в NCBI референсной последовательности NCBI:NM_000572.2. Нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 38, может представлять последовательность ДНК, показанную в SEQ ID NO: 39. Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 40, может представлять последовательность ДНК, показанную в SEQ ID NO: 41, и нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 42, может представлять ДНК, показанную в SEQ ID NO: 43.
Кроме того, ген, кодирующий IL-10, может представлять нуклеотидную последовательность, кодирующую вариант IL-10, который сохраняет активность IL-10. Нуклеотидная последовательность, кодирующая вариант IL-10, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, 90% или более с аминокислотной последовательностью IL-10, показанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности на уровне 95% или более.
Нуклеотидная последовательность, кодирующая вариант IL-10, может представлять нуклеотидную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, 90% или более с нуклеотидной последовательностью IL-10, показанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, имеющую гомологию последовательности на уровне 95% или более.
Как здесь используется, термин «GDNF» относится к белку, входящему в семейство лигандов GDNF. Семейство лигандов GDNF состоит из GDNF, нейротурина (NRTN), артемина (ARTN) и персефина (PSPN). Кроме того, GDNF представляет белок, который способствует выживаемости многих видов нейронов и передает сигналы через рецептор GFRα1. GDNF может представлять белок, происходящий от человека, крысы, собаки, кошки или лошади, не ограничиваясь этим. Ген, кодирующий GDNF, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, показанную в SEQ ID NO: 11, 44, 46 или 48.
В частности, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 11, может представлять последовательность ДНК, показанную в SEQ ID NO: 12 или 13, и может быть последовательностью мРНК, показанной в NCBI референсной последовательности NCBI:NM_199231.2. Кроме того, нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 44, может представлять последовательность ДНК, показанную в SEQ ID NO: 45. Нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанную в SEQ ID NO: 46, может представлять последовательность ДНК, показанную в SEQ ID NO: 47, и нуклеотидная последовательность, кодирующая аминокислотную последовательность, показанная в SEQ ID NO: 48, может представлять последовательность ДНК, показанную в SEQ ID NO: 49.
Кроме того, ген, кодирующий GDNF, может представлять нуклеотидную последовательность, кодирующую вариант GDNF, который сохраняет активность GDNF. Нуклеотидная последовательность, кодирующая вариант GDNF, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, или 90% или более с аминокислотной последовательностью GDNF, показанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, кодирующую аминокислотную последовательность, имеющую гомологию последовательности на уровне 95% или более.
Кроме того, нуклеотидная последовательность, кодирующая вариант GDNF, может представлять нуклеотидную последовательность, имеющую гомологию последовательности, по меньшей мере, 60% или более, 70% или более, 80% или более, 90% или более с нуклеотидной последовательностью GDNF, показанной выше, и наиболее предпочтительно, может представлять нуклеотидную последовательность, имеющую гомологию последовательности на уровне 95% или более.
GABA, продукт гена GAD, обладает эффектом блокирования трансдукции болевых сигналов, но ее избыточные количества могут вызывать такие симптомы, как зуд, головокружение, сонливость и т. д., а также побочные эффекты, такие как увеличение частоты сердечных сокращений или частоты дыхательных движений (Longo, Am. Fam. Physician, 2000).
Известно, что IL-10 является цитокином, который проявляет противовоспалительное действие, но могут возникнуть побочные эффекты, такие как гриппоподобные симптомы и тому подобное (Friedrich, J. Invest. Dermatol., 2002).
Кроме того, известно, что экспрессия GDNF проявляет анальгетическую эффективность при различных болях, таких как нейропатическая боль и тому подобное, но сообщалось, что в экспериментах на обезьянах избыточное введение вызывало повреждение нейронов головного мозга (Hovland, Toxicol. Pathol., 2007).
Фармацевтическая композиция по настоящему изобретению может проявлять анальгетическое действие с небольшим количеством генов или носителей, содержащих их. Композиция по настоящему изобретению состоит из вектора, содержащего ген, кодирующий GAD, вектора, содержащего ген, кодирующий противовоспалительный цитокин в нервных тканях, и/или вектора, содержащего ген, кодирующий GDNF. Посредством совместного введения веществ, обладающих различными механизмами анальгетического действия, можно достичь аналогичного или лучшего эффекта облегчения или лечения боли в более низкой дозировке, чем при введении по отдельности.
В частности, согласно настоящему изобретению, когда два или более генов, выбранных из группы, состоящей из генов, кодирующих GAD65, IL-10 и GDNF, вводятся совместно, то возникает синергетический анальгетический эффект. Следовательно, фармацевтическая композиция по настоящему изобретению может быть пригодна для облегчения или лечения боли.
Согласно одному варианту осуществления настоящего изобретения первый вектор, второй вектор и/или третий вектор могут представлять векторы на основе аденоассоциированного вируса. Аденоассоциированный вирус не ограничивается конкретным серотипом и предпочтительно может быть любым из AAV1-AAV9.
Боль может быть выбрана из группы, состоящей из ноцицептивной боли, психогенной боли, воспалительной боли, патологической боли, нейропатической боли, боли при раке, послеоперационной боли, боли при невралгии тройничного нерва, идиопатической боли, диабетической нейропатической боли или мигрени. В конкретном примере боль может представлять собой боль при пояснично-крестцовой радикулопатии (LSR).
Воспалительная боль относится к боли, связанной с повреждением тканей и инфильтрацией иммунных клеток. Кроме того, патологическая боль означает болезненное состояние, при котором боль вызвана повреждением нервной ткани или ее аномальной функцией. Кроме того, патологическая боль может быть дисфункциональной болью, такой как фибромиалгия, боль при синдроме раздраженного кишечника или головная боль напряжения.
Кроме того, боль может включать боль в спине, которая может быть анатомически различимой: боль в шее, боль в средней части спины, боль в нижней части спины или боль в копчике. Кроме того, боль может представлять, по меньшей мере, одну, выбранную из группы, состоящей из нейропатической боли, боли при раке, послеоперационной боли, боли при невралгии тройничного нерва, идиопатической боли, диабетической нейропатической боли или мигрени и тому подобного. В конкретном примере боль может представлять собой боль при пояснично-крестцовой радикулопатии.
Нейропатическая боль может быть вызвана повреждением или заболеванием, которое отрицательно влияет на соматосенсорную систему. Нейропатическая боль может представлять аномальное ощущение, называемое аллодинией и дизестезией. Кроме того, общие характеристики нейропатической боли включают чувство жара или холода, покалывание как булавками и иголками, онемение и зуд. Напротив, ноцицептивная боль часто проявляется в виде ноющей боли.
Кроме того, мигрень ассоциирована с рядом симптомов со стороны вегетативной нервной системы и является хроническим заболеванием, которое вызывает головные боли со степенью тяжести от незначительной до сильно выраженной. Известно, что мигрень связана с повышенной возбудимостью коры головного мозга и аномальной регуляцией болевых нейронов в ядре тройничного нерва ствола головного мозга (Noseda, Pain, 2013).
В частности, фармацевтическую композицию по настоящему изобретению можно использовать для облегчения или лечения нейропатической боли и хронической боли при раке.
Как здесь используется, термин «облегчение или лечение» означает любое действие, которое ослабляет или изменяет целебным образом болевые симптомы введением композиции по настоящему изобретению.
Фармацевтическая композиция по настоящему изобретению может дополнительно содержать физиологически приемлемый носитель. Кроме того, фармацевтические композиции по настоящему изобретению могут дополнительно содержать подходящие эксципиенты и разбавители, обычно используемые в приготовлении фармацевтических композиций. Кроме того, композиции по настоящему изобретению можно использовать посредством их приготовления общими способами в виде оральных составов, таких как порошки, гранулы, таблетки, капсулы, суспензии, эмульсии, сиропы, аэрозоли и т. д., составов для наружного применения, суппозиториев или инъекционных составов. Конкретно, фармацевтическая композиция может быть в форме инъекционного препарата. В отношении подходящих составов, известных в данной области, то можно использовать составы, которые приведены в Remington's Pharmaceutical Science (1985).
Кроме того, фармацевтическая композиция может содержать соль (хлорид натрия), лактозу, декстрозу, сахарозу, сорбит, маннит, ксилит, эритрит, мальтит, крахмал, аравийскую камедь, альгинат, желатин, фосфат кальция, силикат кальция, целлюлозу, метилцеллюлозу, микрокристаллическую целлюлозу, поливинилпирролидон, воду, метилгидроксибензоат, пропилгидроксибензоат, тальк, стеарат магния, минеральное масло и т. д., в качестве носителей, эксципиентов и разбавителей. Для формуляции фармацевтической композиции по настоящему изобретению обычно можно использовать разбавители или эксципиенты, такие как наполнители, добавки, связующие вещества, увлажнители, дезинтегрирующие агенты, поверхностно-активные вещества и т.д.
Составы для парентерального введения могут включать стерильные растворы, неводные растворы, суспензии, эмульсии, лиофилизированные составы и суппозитории. Пропиленгликоль, полиэтиленгликоль, растительное масло, такое как оливковое масло, и инъецируемый сложный эфир, такой как этилолеат и т. д., можно использовать для неводных растворов и суспензий. Витепсол, макрогол, твин 61, масло какао, лауриновое масло, глицерожелатин и т. д. можно использовать в качестве основы для суппозиториев.
Настоящее изобретение также относится к способу облегчения или лечения боли, включающему введение фармацевтической композиции, содержащей два или более генов, выбранных из группы, состоящей из генов, кодирующих GAD, IL-10 и GDNF, субъекту, нуждающемуся в этом.
Боль представляет боль, аналогичную описанной выше в отношении фармацевтической композиции.
Субъектом может быть млекопитающее, включая человека, или клетка и/или ткань, выделенная из организма млекопитающего, включая человека. Как здесь используется, термин «животное, отличное от человека» предназначен для включения всех позвоночных животных, которые включают млекопитающих и животных, отличных от млекопитающих, таких как приматы, овцы, собаки, кошки, лошади, коровы, куры, земноводные, рептилии и т.д.
В отношении пути введения, дозы и кратности введения, то фармацевтическую композицию можно вводить субъекту различными путями и в различных дозах, в зависимости от состояния пациента и наличия или отсутствия побочных эффектов, и специалисты в данной области могут соответствующим образом выбрать диапазон оптимальных способов введения, доз и кратностей введения. Кроме того, фармацевтическую композицию можно вводить в комбинации с другим лекарственным средством или физиологически активным веществом, о котором известно, что оно проявляет терапевтическую эффективность при расстройстве, которое лечат. Также фармацевтическая композиция может быть приготовлена в форме комбинированной композиции.
В частности, фармацевтическая композиция по настоящему изобретению может быть обеспечена в форме состава для инъекции. Например, инъекции могут быть включать: подкожную инъекцию, внутримышечную инъекцию, внутривенную инъекцию, эпидуральную инъекцию или интратекальную инъекцию и тому подобное. Конкретно, фармацевтическую композицию можно вводить посредством эпидуральной инъекции или интратекальной инъекции, и более конкретно, ее можно вводить посредством трансфораминальной эпидуральной инъекции или интратекальной инъекции.
Как здесь используется, термин «трансфораминальная эпидуральная инъекция» относится к способу введения лекарственного средства внутрь межпозвонкового отверстия, которое представляет пространство, где нервы выходят из спинного мозга через пространство между позвоночными вырезками и в пространство вне твердой мозговой оболочки, которая окружает спинной мозг и спинномозговые нервы. В одном варианте осуществления, если фармацевтическая композиция по настоящему изобретению приготовлена из вирусов, то лекарственное средство можно вводить внутрь межпозвонкового отверстия субъекта проведением терапии эпидуральной инъекцией.
Как здесь используется, термин «интратекальная инъекция» относится к способу введения путем инъекции лекарственного средства в пространство внутри твердой мозговой оболочки спинномозгового канала. В одном варианте осуществления, если фармацевтическая композиция по настоящему изобретению приготовлена из плазмид, то лекарственное средство может быть введено внутрь спинномозгового канала субъекта проведением терапии интратекальной инъекцией.
В частности, если фармацевтическая композиция приготовлена из вирусных векторов, то ее можно вводить взрослому человеку в количестве от 1,0 × 106 до 1,0 × 1014 vg. Кроме того, когда нужно вводить два типа вирусов, то каждый тип вирусов можно вводить в количестве от 5,0 × 105 до 5,0 × 1013 vg. Если необходимо ввести три типа вирусов, то каждый тип вирусов можно вводить в количестве от 3,0 × 105 до 3,0 × 1013 vg.
Кроме того, если фармацевтическая композиция приготовлена из невирусных векторов, то ее можно вводить взрослому человеку в концентрации от 0,1 до 10 мг/мл. Также, если фармацевтическая композиция приготовлена из плазмидных векторов, то дозировка может составлять 0,1 мл, 1 мл, 2 мл, 3 мл, 4 мл, 5 мл, 6 мл, 7 мл, 8 мл, 9 мл, 10 мл или более, включая все значения и диапазоны между ними.
В отношении кратности введения вирусного вектора, то его можно вводить один или более раз, или от 1 до 10 раз. Также его можно вводить с интервалом от 1 суток до 1 месяца или от 1 месяца до 1 года в случае повторного введения. Кроме того, если фармацевтическая композиция приготовлена из невирусных векторов, то ее можно вводить 1 или более раз, или от 1 до 10 раз. Кроме того, ее можно вводить с интервалом от 12 до 24 ч или от 1 до 14 суток в случае многократного введения.
Настоящее изобретение относится к применению фармацевтической композиции по настоящему изобретению для облегчения или лечения боли.
Настоящее изобретение обеспечивает применение фармацевтической композиции по настоящему изобретению для получения терапевтического агента для облегчения или лечения боли.
Способы осуществления изобретения
Далее настоящее изобретение будет подробно описано со ссылкой на примеры. Однако следующие примеры приведены только для иллюстративных целей и не предназначены для ограничения объема настоящего изобретения.
Пример 1. Приготовление и анализ свойств рекомбинантного аденоассоциированного вируса
Аденоассоциированные вирусы, необходимые для настоящего изобретения, конструировали и получали на основе безхелперной системы AAV (Agilent).
Пример 1.1. Конструирование плазмиды pAAV-GAD65
Для конструирования плазмиды pAAV-GAD65, показанной на фиг. 1, промоторную область CMV в pJDK-rGAD65 (Lee, Gene Ther., 2005) амплифицировали с использованием ПЦР, и затем полученный продукт вводили в pGEM-T (Promega) для получения pGEM-Т-СMV. Последовательности праймеров, использованные для амплификации промотора CMV, являются следующими:
F-JDK (SEQ ID NO: 15): 5'-TTCGGCCGTCGAGGAGCTTGGCCCATTG-3'
R-JDK (SEQ ID NO: 16): 5'-GACGTCGACCTAGCTAGCGAATTCGGGGCCGCGGAG-3'.
В отношении гена GAD65, то ген с последовательностью, показанной SEQ ID NO: 3, конструировали кодон-оптимизацией, чтобы он подходил для человека, на основе человеческой GAD65 (NCBI NM_000818.2), показанной аминокислотной последовательностью SEQ ID NO: 1, и передавали в Bioneer для синтеза гена. Ген hGAD65, введенный в pGEM-T, обрабатывали NheI и SalI с получением фрагмента ДНК размером 1,7 т.п.н. Затем его подвергали лигированию с фрагментом ДНК размером 3,7 т.п.н., полученным обработкой pGEM-T-CMV NheI и SalI, для завершения конструирования pGEM-T-CMV-hGAD65.
SV40pA амплифицировали проведением ПЦР с использованием pCI (Invitrogen) в качестве матрицы, и затем полученный продукт обрабатывали ClaI и SalI с получением фрагмента ДНК размером 222 п.н. Фрагмент ДНК подвергали лигированию с фрагментом ДНК размером 5,4 т.п.н., полученным разрезанием pGEM-T-CMV-hGAD65 с использованием ClaI и SalI, с получением окончательной конструкции pGEM-T-CMV-hGAD65-SV40pA. Последовательности праймеров, использованные для амплификации SV40pA, являются следующими:
F-SV40pA (SEQ ID NO: 17): 5'-CCATCGATCAGACATGATAAGATACATTGATGAG-3'
R-SV40pA (SEQ ID NO: 18): 5'-GACGTCGACGCGGCCGCTACCACATTTGTAGAGGTTTTACTTG-3'.
Для конструирования аденоассоциированного вирусного вектора ген резистентности к ампициллину в pAAV-MCS (Agilent) заменяли геном резистентности к канамицину. Ген резистентности к канамицину амплифицировали с помощью ПЦР с использованием pET-28 (а) (Novagen) в качестве матрицы. Амплифицированный ген резистентности к канамицину длиной 816 п.н. подвергали лигированию с pGEM-T для получения pGEM-T-Kan'. Последовательности праймеров, использованные для амплификации гена резистентности к канамицину, являются следующими:
F-Kan (SEQ ID NO: 19): 5'-AGGCGCCATGAGCCATATTCAACGGGAA-3'
R-Kan (SEQ ID NO: 20): 5'-TTCATGATTAGAAAAACTCATCGAGCATC-3'.
Для вставки гена резистентности к канамицину были соответственно созданы сайты узнавания рестриктаз SpeI и EcoRV мутагенезом выше и ниже гена резистентности к ампициллину в pAAV-MCS, и затем полученную конструкцию снова обрабатывали SpeI и EcoRV. Полученный продукт подвергали лигированию с фрагментом ДНК, полученным разрезанием ранее сконструированной pGEM-T-Kanr с помощью NheI и EcoRV, с получением pAAV-MCS-Kanr.
Сконструированную pAAV-MCS-Kanr обрабатывали NotI и BamHI, и затем подвергали лигированию с фрагментом ДНК размером 2,7 т.п.н., полученным путем разрезания pGEM-T-CMV-hGAD65-SV40pA с помощью EagI и PvuI, с получением pssAAV-GAD65.
Для введения экспрессионной кассеты GAD65 в pVAX1 (Invitrogen), генерировали сайт узнавания рестриктазы BamHI мутагенезом ниже bGHpA. Затем полученный продукт разрезали с помощью MluI и NheI с получением фрагментов ДНК. Промоторные области LITR и CMV амплифицировали с помощью ПЦР с использованием pssaAV-GAD65 в качестве матрицы и клонировали в pGEM-T easy (Promega). Затем полученную конструкцию разрезали с помощью AscI и NheI и подвергали лигированию с предварительно приготовленным вектором pVAX1, для конструирования pVAX1-LITR-CMV. Последовательности праймеров, использованные для амплификации промоторной области LITR и CMV, являются следующими:
F-ITR (SEQ ID NO: 21): 5'-ATGGCGCGCCCCTGGCCTTTTGCTGGCC-3',
R-JDK (SEQ ID NO: 16): 5'-GACGTCGACCTAGCTAGCGAATTCGGGGCCGCGGAG-3'.
pVAX1-LITR-CMV снова разрезали NotI и NheI с получением фрагментов ДНК. PSSAAV-GAD65 разрезали EagI и NheI и подвергали лигированию с предварительно приготовленными фрагментами ДНК для конструирования pVAX1-LITR-CMV-hGAD65-SV40pA.
PVAX1-LITR-CMV-hGAD65-SV40pA разрезали HpaI и BamHI с получением фрагментов ДНК. Кроме того, psA-SV40pA-RITR, которая ранее была получена амплификацией ПЦР с использованием pssaAV-GAD65 в качестве матрицы и клонированием в pGEM-T easy, обрабатывали HpaI и BamHI с получением фрагментов ДНК. Два фрагмента ДНК лигировали для завершения конструирования pVAX1-LITR-CMV-hGAD65-SV40pA-RITR (в дальнейшем сокращенно обозначенной как «pAAV-GAD65»). Последовательности праймеров, использованные для амплификации области SV40pA и RITR, являются следующими:
F-SV40pA (SEQ ID NO: 17): 5'-CCATCGATCAGACATGATAAGATACATTGATGAG-3'
R-ITR (SEQ ID NO: 22): 5'-ATGGATCCGCTAGTAAATACCGCATCAG-3'.
Схематическое изображение плазмиды pAAV-GAD65 показано на фиг. 1.
Для конструирования модифицированной pAAV-GAD65 проводили следующую процедуру.
Сначала вектор разрезали Nhe1, и затем произвольную случайную нуклеотидную последовательность вставляли между CMV-промотором и геном GAD65 методом инфузии. Вставленные нуклеотидные последовательности являются следующими:
Scramble stuffer (SEQ ID NO: 29):
5'-GTCGACGGTATCGATAAGCTTGATATCGAATTCCTGCAGCCC-3'
Stuffer_scramble_F (SEQ ID NO: 30):
5'-CTAGGTCGACGGTATCGATAAGCTTGATATCGAATTCCTGCAGCCC-3'
Stuffer_scramble_R (SEQ ID NO: 31):
5'-CTAGGGGCTGCAGGAATTCGATATCAAGCTTATCGATACCGTCGAC-3'.
Затем нуклеотидную последовательность WPRE (Schambach, Gene Ther., 2006), из которой была удалена область Х-белка, которая может оказывать онкогенный эффект, амплифицировали с помощью ПЦР и вставляли в конец гена GAD65 с использованием рестриктаз Pac1 и Hpa1. В то же время некоторый участок SV40pA удаляли для получения модифицированной SV40pA. Последовательности праймеров, использованные для амплификации WPRE, являются следующими:
WPRE_Pac1_F (SEQ ID NO: 25):
5'-GGTGGTTTAATTAAAATCAACCTCTGGATTACAAAATTTG-3'
WPRE_modi_Hpa1_R (SEQ ID NO: 26): 5'-GGTGGTGTTAACGACAACACCACGGAATTG-3'.
Окончательно модифицированная плазмида представляла pVAX1-LITR-CMV-scramble stuffer-hGAD65-WPRE (modi)-SV40pA (modi)-RITR (в дальнейшем сокращенно обозначенная как «pAAV-GAD65-modi»), и ее схематическое изображение приведено на фиг. 1.
Пример 1.2. Конструирование плазмиды pAAV-IL-10
Плазмиду pAAV-IL-10 конструировали аналогично способу, описанному в примере 1.1. В отношении гена крысиного IL-10, то ген, представленный нуклеотидной последовательностью SEQ ID NO: 8, конструировали кодон-оптимизацией, чтобы он подходил для крыс, на основе человеческого IL-10 (NCBI NM-012854), представленного аминокислотной последовательностью, показанной в SEQ ID NO: 6, и передавали в Bioneer для синтеза гена. Ген rIL-10 амплифицировали проведением ПЦР с использованием крысиного гена IL-10, вставленного в pGEM-T easy в качестве матрицы, и затем полученный продукт обрабатывали NheI и SalI с получением фрагмента ДНК размером 0,5 т.п.н. Кроме того, PGEM-T-CMV обрабатывали NheI и SalI с получением фрагмента ДНК размером 3,7 т.п.н. Два фрагмента ДНК лигировали с получением pGEM-T-CMV-rIL-10. Последовательности праймеров, использованные для амплификации rIL-10, являются следующими:
F-rIL-10 (SEQ ID NO: 23): 5'-CCGCTAGCGCCACCATGCCT-3'
R-rIL-10 (SEQ ID NO: 24): 5'-GACGTCGACGCCATCGATGGCTTAATTAATCAATTCTTC-3'.
В отношении SV40pA, то ген амплифицировали проведением ПЦР с использованием pCI в качестве матрицы, и затем обрабатывали NotI и SalI с получением фрагмента ДНК размером 222 п.н. Кроме того, pGEM-T-CMV-rIL-10 обрабатывали ClaI и SalI с получением фрагмента ДНК размером 4,2 т.п.н. Два фрагмента ДНК лигировали для конструирования pGEM-T-CMV-rIL-10-SV40pA. Последовательности праймеров, использованные для амплификации SV40pA, являются следующими:
F-SV40pA (SEQ ID NO: 17): 5'-CCATCGATCAGACATGATAAGATACATTGATGAG-3'
R-SV40pA (SEQ ID NO: 18): 5'-GACGTCGACGCGGCCGCTACCACATTTGTAGAGGTTTTACTTG-3'.
pGEM-T-CMV-rIL-10-SV40pA обрабатывали EagI с получением фрагмента ДНК размером 1,6 т.п.н. Кроме того, pAAV-MCS-Kanr обрабатывали NotI и BamHI с получением фрагментов ДНК. Затем два фрагмента ДНК лигировали для конструирования pssAAV-CMV-rIL-10-SV40pA (далее сокращенно обозначенной как «pAAV-IL-10»). Схематическое изображение плазмиды pAAV-IL-10 приведено на фиг. 2.
Пример 1.3. Конструирование плазмиды pAAV-GDNF
В отношении гена человеческого GDNF, то ген, представленный SEQ ID NO: 13, конструировали кодон-оптимизацией, чтобы он подходил для человека, на основе человеческого GDNF (NCBI NM_199231.2), представленного аминокислотной последовательностью SEQ ID NO: 11, и передавали в Bioneer для синтеза гена. Ген hGDNF, вставленный в плазмиду pGEM-B1, обрабатывали NheI и PacI с получением фрагмента ДНК размером примерно 0,6 т.п.н. Плазмиду pGEM-T-CMV-rIL-10-SV40pA обрабатывали NheI и PacI с получением фрагмента размером 2,8 т.п.н., из которого был удален ген rIL-10. Два фрагмента ДНК лигировали с получением плазмиды pGEM-T-CMV-hGDNF-SV40pA.
Затем полученную плазмиду pGEM-T-CMV-hGDNF-SV40pA обрабатывали EagI с получением фрагмента ДНК размером 1,5 т.п.н. Кроме того, pAAV-MCS-Kanr обрабатывали NotI и BamHI с получением фрагмента ДНК размером 1,8 т.п.н. Два фрагмента ДНК лигировали для конструирования pssAAV-CMV-hGDNF-SV40pA-Kanr (далее сокращенно обозначенной как «pAAV-GDNF»). Схематическое изображение плазмиды pAAV-GDNF приведено на фиг. 3.
Пример 1.4. Конструирование плазмиды pAAV-GDNF-IL-10
Промотор CAG (цитомегаловирусный энхансер, промотор гена β-актина курицы и поли(А)-сигнал гена β-глобина кролика) подвергали ПЦР-амплификации с использованием pAxCAwtit2, входящей в набор двойной экспрессии аденовируса (Takara), и обрабатывали ApaI и XbaI для повышения экспрессии, тем самым удаляя примерно 80% области β-актина курицы в промоторе CAG с получением короткого промотора CAG (sCAG) (Fagoe, Gene Ther., 2014). В отношении гена человеческого IL-10, то ген, кодирующий SEQ ID NO: 14, конструировали кодон-оптимизацией гена, кодирующего SEQ ID NO: 9, для того, чтобы он подходил для человека, и передавали в Bioneer для синтеза гена. Затем фрагмент ДНК поли(A)-сигнала гена бычьего гормона роста (bGH) получали с помощью ПЦР-амплификации. PVAX1/sCAG-hIL-10-bGHpA конструировали с использованием pVAX1 (Invitrogen) для включения промотора и поли(А)-сигнала гена человеческого IL-10.
Затем pVAX1/CMV-hGDNF-SV40pA получали аналогично способу, описанному в примере 1.1., с использованием гена человеческого GDNF из примера 1.3. Затем генную кассету SV40pA-hGDNF-CMV амплифицировали проведением ПЦР с использованием pVAX1/CMV-hGDNF-SV40pA в качестве матрицы и получали фрагмент ДНК размером 1,5 т.п.н. Последовательности праймеров, использованные для амплификации генной кассеты, являются следующими:
SV40-CMV-sCAG-bGHpA-Infu-F (SEQ ID NO: 27):
5'-CCTGCGGCCGGTCGACTACCACATTTGTAGAGGTTTTACTTGC-3'
SV40-CMV-sCAG-bGHpA-Infu-R (SEQ ID NO: 28):
5'-AATAATCAATGTCGACTCGAGGAGCTTGGCCCATT-3'
Затем pVAX1/sCAG-hIL-10-bGHpA обрабатывали SalI с получением фрагмента ДНК размером примерно 3,9 т.п.н., и фрагмент ДНК размером 1,5 т.п.н., описанный выше, вставляли в фрагмент ДНК размером 3,9 т.п.н., используя набор для клонирования ДНК In-Fusion HD (Clontech), с получением pVAX1/SV40pA-hGDNF-CMV-sCAG-hIL-10-bGHpA (в дальнейшем сокращенно обозначенной как «pAAV-GDNF-IL-10»). Плазмида pAAV-GDNF-IL-10 схематически показана на фиг.4.
Экспериментальный пример 1. Подтверждение экспрессии плазмид pAAV-GAD65, pAAV-IL-10, pAAV-GDNF и pAAV-GDNF-IL-10
Плазмидами pAAV-GAD65, pAAV-IL-10, pAAV-GDNF или pAAV-GDNF-IL-10, полученными соответственно в примерах 1.1.-1.4., трансфицировали клетки линии эмбриональной почки человека 293Т с использованием jetPRIME (Polyplus). Трансфицированные клетки культивировали в термостате при 37°С в течение 48 ч. Затем собирали клеточную культуральную среду или культивированные клетки. Клетки растворяли в растворителе, и полученные образцы обрабатывали каждым из антител к GAD65 (Merck Millipore), IL-10 (Santa Cruz) и GDNF (R & D systems) и подвергали анализу вестерн-блоттингом.
В частности, в случае pAAV-GAD65 клетки линии эмбриональной почки человека 293T обрабатывали 2 мкг плазмиды pAAV-GAD65 и культивировали в течение 48 ч. После этого культивированные клетки растворяли и экспрессию GAD65 в клетках подтверждали вестерн-блоттингом.
В случае плазмид pAAV-IL-10 и pAAV-GDNF клетки линии эмбриональной почки человека 293Т обрабатывали 1 мкг плазмиды pAAV-IL-10 или плазмиды pAAV-GDNF и культивировали в течение 48 ч. После этого собирали культуральную среду и экспрессию IL-10 или GDNF в среде подтверждали вестерн-блоттингом.
В случае плазмиды pAAV-GDNF-IL-10 клетки линии эмбриональной почки человека 293Т обрабатывали 1 мкг плазмиды pAAV-GDNF-IL-10 и культивировали в течение 48 ч. После этого культивированные клетки растворяли и экспрессию внутриклеточных IL-10 и GDNF подтверждали вестерн-блоттингом.
В результате было подтверждено, что трансфицированные плазмиды pAAV-GAD65, pAAV-IL-10, pAAV-GDNF или pAAV-GDNF-IL-10 экспрессировались (фиг. 5 и 6).
Пример 2. Получение рекомбинантного аденоассоциированного вируса
Вирус AAV-IL-10, использованный в эксперименте, получали и очищали UNC Vector Core. Способ получения заключается в следующем. Клетки линии эмбриональной почки человека 293Т трансфицировали PVax-rIL-10, pHelper и pRC5. После этого полученный продукт подвергали очистке колоночной хроматографией для защиты вируса AAV5-IL-10. Титр продуцированного вируса измеряли с использованием кПЦР.
Вирусы AAV-GAD65 и AAV-GDNF получали и очищали KRcrogen. Способ получения заключается в следующем. AAV-трансгенами (плазмидами pAAV-GAD65 и pAAV-GDNF) соответственно трансфицировали клетки линии эмбриональной почки человека 293Т с использованием кальций-фосфатного метода вместе с pHelper и pRC. В случае GAD65 использовали pRC5, введенную с капсидным геном серотипа 5. В случае GDNF использовали pRC1, введенную с капсидным геном AAV серотипа 1. Трансфицированные клетки культивировали в термостате при 37°С, и клетки собирали через 48 ч.
Затем выделяли и очищали только полосы, содержащие вирусы, методом высокоскоростного центрифугирования в градиенте плотности хлорида цезия для защиты вирусов AAV5-GAD65 и AAV1-GDNF. Титры продуцированных вирусов измеряли с использованием кПЦР.
Вирус AAV-GDNF-IL-10 получали и очищали Cdmogen. Способ получения заключается в следующем. AAV-трансгеном (плазмидой pAAV-GDNF-IL-10) трансфицировали клетки линии эмбриональной почки человека 293Т с использованием кальций-фосфатного метода вместе с pHelper и pRC5. Трансфицированные клетки культивировали в термостате при 37°С, и клетки собирали через 48 ч.
Затем выделяли и очищали только полосы, содержащие вирусы, методом высокоскоростного центрифугирования в градиенте плотности хлорида цезия для защиты вируса AAV5-GDNF-IL-10. Титр продуцированного вируса измеряли с использованием кПЦР.
Вирус AAV-GAD65-modi получали и очищали Cdmogen. Способ получения заключается в следующем. AAV-трансгеном (плазмидой pAAV-hGAD65-modi) трансфицировали клетки линии эмбриональной почки человека 293Т с использованием кальций-фосфатного метода вместе с pHelper и pRC5. Трансфицированные клетки культивировали в термостате при 37°С, и клетки собирали через 48 ч.
Затем выделяли и очищали только полосы, содержащие вирусы, методом высокоскоростного центрифугирования в градиенте плотности хлорида цезия для защиты вируса AAV5-GAD65-modi. Титр продуцированного вируса измеряли с использованием КПЦР.
Экспериментальный пример 2. Анализ свойств рекомбинантного аденоассоциированного вируса
Для изучения экспрессии белков рекомбинантного аденоассоциированного вируса, доставленного в клетку, клетки линии эмбриональной почки человека 293Т или клетки HeLa обрабатывали вирусами AAV-GAD65, AAV-GAD65-modi, AAV-IL-10 или AAV-GDNF, полученными, как описано выше, и экспрессию белков анализировали вестерн-блоттингом. В частности, клетки 293T или HeLa высевали из расчета 5 × 105 клеток/лунку в 6-луночный планшет. На следующие сутки клетки соответственно обрабатывали 3 типами вирусов из расчета 10000 vg/лунку и затем культивировали в термостате при 37°С. Через 48 ч клетки собирали и растворяли в растворителе, и культуральную среду концентрировали с использованием системы Амикон (Merck Millipore). Затем полученные образцы соответственно обрабатывали антителами к GAD65 (Cell signaling), IL-10 (Santa Cruz) и GDNF (системы R & D) и подвергали анализу вестерн-блоттингом.
В результате было подтверждено, что каждый целевой белок экспрессировался в клеточном лизате клеток линии эмбриональной почки человека 293T или клеток линии HeLa, обработанных вирусами AAV-GAD65, AAV-GAD65-modi, AAV-IL-10 или AAV-GDNF (фиг. 7). Таким образом, было подтверждено, что аномалии в структурах и свойствах рекомбинантных аденоассоциированных вирусов, использованных в эксперименте, отсутствовали.
Кроме того, для подтверждения того, что GAMA продуцируется вирусом AAV-GAD65 или AAV-GAD65-modi, культуральную среду клеток, обработанных вирусами AAV-GAD65 или AAV-GAD65-modi, собирали и подвергали анализу GABA ELISA (LDN). Для каждой экспериментальной группы готовили два идентичных образца по отдельности для проведения анализа, и гистограмма показывает значение для каждого образца.
В результате было подтверждено, что GABA секретируется в культуральную среду GAD65, введенной в клетки вирусом AAV-GAD65 или AAV-GAD65-modi (фиг. 8).
Экспериментальный пример 3. Сравнение анальгетической эффективности при введении вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности и совместном введении вирусов AAV-GAD65 и AAV-GDNF или AAV-IL-10 и AAV-GDNF
Экспериментальный пример 3.1. Подготовка образцов для введения
Вирусы, полученные в примере 2, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Вирусы AAV-GAD65, AAV-IL-10, AAV-GDNF и AAV-GFP разводили в PBS с получением титров, показанных в таблице 1. Вирус AAV-GFP вводили в том же количестве, что и другие рекомбинантные аденоассоциированные вирусы. GFP представляет собой белок, не обладающий анальгетической эффективностью. Нужные вирусы смешивали в соответствии с титрами, указанными в таблице 1, и вводили в количестве 9,0 × 108 vg/5 мкл на животное (vg: вирусный геном).
|
Экспериментальный пример 3.2. Получение крыс с индуцированной нейропатической болью и введение образцов
Самцов крыс SD с массой 150-200 г подвергали ингаляционной анестезии. Затем верхнюю часть голени надрезали и, и оба конца общего малоберцового нерва и большеберцового нерва связывали, и узлы делали с интервалом от 0,5 до 1 см наложением шовного материала 7-0. Области между узлами двух нервных пучков разрезали ножницами, и место разреза зашивали. После этого крысам давали восстановиться от наркоза и возвращали в клетку. Через две недели проводили тест с волосками фон Фрея для исследования индукции боли, и затем соответственно вводили образцы, приготовленные в примере 2.1 (Decosterd, Pain, 2000).
Образцы вводили методом трансфораминальной эпидуральной инъекции в участок, смежный с ганглием дорсальных корешков (DRG). Крысу с индуцированной болью подвергали ингаляционной анестезии, и позвонки обнажали прямым надрезом на спине крысы на уровне поясничных позвонков от L3 до L5. На стороне открытого пространства делали видимым поперечный отросток L4, одна из проекций позвоночника. Крысу помещали в боковое положение, чтобы ее латеральная сторона была видна сверху, и было видно межпозвоночное отверстие L4.
Затем иглу, прикрепленную к катетеру, вводили в подготовленный образец, и шприц Гамильтона соединяли с противоположным концом катетера и вытягивали до отметки 5 мкл для введения образца в катетер. Шприц Гамильтона отсоединяли от катетера, и затем катетер фиксировали удерживанием на расстоянии 1 см от кончика иглы с помощью зажима Halsted-Mosquito. Затем, удерживая и вытягивая позвонок L4 вверх с помощью пинцета, кончик иглы, зафиксированный зажимом Halsted-Mosquito, другой рукой проводили вокруг межпозвонкового отверстия L4. Кончик иглы вводили в изогнутую область внутри межпозвонкового отверстия, пространство которого было закреплено. Затем удерживаемую иглу высвобождали.
После подтверждения того, что игла была зафиксирована, к катетеру, соединенному с противоположной стороной иглы, подсоединяли шприц на 1 мл. Осторожно нажимая на поршень шприца, образец медленно вводили вокруг ганглия дорсальных корешков крысы. После этого место разреза зашивали. Через 4 недели после введения образца наблюдали за болевыми реакциями с использованием теста с волосками фон Фрея.
Экспериментальный пример 3.3. Наблюдение за болевыми реакциями с использованием теста с волосками фон Фрея
Наблюдение за болевыми реакциями проводили с помощью теста с волосками фон Фрея. Метод заключается в том, чтобы рассчитать пороговое значение в соответствии с заранее определенным паттерном болевой реакции, с использованием набора из восьми волосков с массой 0,4; 0,6; 1; 2; 4; 6; 8 и 15 г.
Области, индуцирующие боль, исследовали по изменению положения от начального участка наружной части стопы до подушечки стопы, где генерировалась боль. Когда крысы внезапно отдергивали подушечку стопы и сжимали или облизывали подушечки стопы ртом при возникновении боли, то можно было обнаружить области, вызывающие боль. Если реакции имели место три или более раз, когда окружающую область пять раз прокалывали волоском на каждой стадии, то это рассматривалось в качестве проявления болевой реакции. Тестирование продолжали заменой волоска на следующей стадии. Таким образом, регистрировали болевой паттерн на каждой стадии.
Болевые паттерны регистрировали на основе таблицы паттернов, установленных S.R. Chaplan, и пороговые значения рассчитывали с использованием болевых паттернов (Chaplan, J. Neurosci. Methods, 1994). В отношении поведенческого анализа, то группы животных обследовались «вслепую» в течение определенного времени, и по меньшей, 3 эксперта проводили наблюдение, и результаты зарегистрированных паттернов статистически обрабатывали, чтобы провести анализ результатов по болевым реакциям.
В результате было установлено, что анальгетический эффект был выше, когда вирусы AAV-GAD65 и AAV-GDNF или AAV-IL-10 и AAV-GDNF комбинировали и вводили совместно по сравнению с введением вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности (фиг. 9).
Экспериментальный пример 4. Сравнение анальгетической эффективности при введении вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности и совместном введении вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF
Вирусы, полученные в примере 2, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Вирусы AAV-GAD65, AAV-IL-10, AAV-GDNF и AAV-GFP разводили в PBS с получением титров, показанных в таблице 2. Вирус AAV-GFP вводили в том же количестве, что и другие рекомбинантные аденоассоциированные вирусы. GFP представляет собой белок, об анальгетической эффективности которого не сообщалось. Вирусы смешивали в соответствии с титрами, указанными в таблице 2, и вводили в количестве 9,0 × 108 vg/5 мкл на животное.
|
Животным, которых использовали в качестве модели боли, полученным аналогично методу, описанному в экспериментальном примере 3.2, вводили образцы, приготовленные согласно титрам вирусов, указанным в таблице 2. Затем проводили тест с волосками фон Фрея аналогично методу, описанному в экспериментальном примере 3.3., для наблюдения за болевыми реакциями.
В результате было установлено, что анальгетический эффект был выше, когда все вирусы AAV-GAD65, AAV-IL-10 и AAV-GDNF вводили совместно по сравнению с введением вирусов AAV-GAD65, AAV-IL-10 или AAV-GDNF по отдельности (фиг. 10).
Экспериментальный пример 5. Сравнение анальгетической эффективности при совместном введении вирусов AAV-GAD65 и AAV-GDNF или вирусов AAV-IL-10 и AAV-GDNF и совместном введении вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF
Аденоассоциированные вирусы, полученные в примере 2, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Вирусы AAV-GAD65, AAV-IL-10, AAV-GDNF и AAV-GFP разводили в PBS для получения титров, указанных в таблице 3. AAV-GFP вводили в том же количестве, что и другие рекомбинантные аденоассоциированные вирусы. GFP представляет собой белок, который не обладает анальгетической эффективностью. Нужные вирусы смешивали в соответствии с титрами, указанными в таблице 3, и вводили в количестве 9,0 × 108 vg/5 мкл на животное.
|
Животным, которых использовали в качестве модели боли, полученным аналогично методу, описанному в экспериментальном примере 3.2, вводили образцы, приготовленные согласно титрам вирусов, указанным в таблице 3. Затем проводили тест с волосками фон Фрея аналогично методу, описанному в экспериментальном примере 3.3., для наблюдения за болевыми реакциями.
В результате было установлено, что анальгетический эффект был выше, когда все вирусы AAV-GAD65, AAV-IL-10 и AAV-GDNF вводили совместно по сравнению с введением вирусов AAV-GAD65, AAV-IL -10 или AAV-GDNF по отдельности (фиг. 11).
Экспериментальный пример 6. Сравнение анальгетической эффективности при введении плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности и совместном введении плазмид pAAV-GAD65 и pAAV-GDNF, или pAAV-IL-10 и pAAV-GDNF
Экспериментальный пример 6.1. Подготовка образцов для введения
Плазмиды, полученные в примере 1, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Плазмиды pAAV-GAD65, pAAV-IL-10, pAAV-GDNF и pVAX1 разводили в Трис-ЭДТА буфере для получения концентраций, указанных в таблице 4. Плазмиду pVAX1 вводили в том же количестве, что и другие плазмиды. Сообщения о том, что плазмида pVAX1 обладает анальгетической эффективностью, отсутствуют. Нужные плазмиды смешивали в соответствии с содержанием, указанным в таблице 4, и вводили в количестве 30 мкг/50 мкл на животное.
|
Экспериментальный пример 6.2. Получение животной модели нейропатической боли и введение образцов
Самцов крыс SD с массой 150-200 г подвергали ингаляционной анестезии. Затем верхнюю часть голени надрезали и, и оба конца общего малоберцового нерва и большеберцового нерва связывали, и узлы делали с интервалом от 0,5 до 1 см наложением шовного материала 7-0. Области между узлами двух нервных пучков разрезали ножницами, и место разреза зашивали. После этого крысам давали восстановиться от наркоза и возвращали в клетку. Через две недели проводили тест с волосками фон Фрея для изучения индукции боли, и затем соответственно вводили образцы, приготовленные в примере 6.1 (Decosterd, Pain, 2000).
Образцы вводили методом интратекальной инъекции. Крысу с индуцированной болью подвергали ингаляционной анестезии, и позвонки обнажали прямым надрезом на спине крысы на уровне поясничного позвонка L5. Пробирку емкостью 50 мл помещали под крысу, чтобы расширить пространство между L5 и L6, и вставляли иглу размером 27 G × 13 мм. После этого шприц на 1 мл, заполненный 50 мкл образца, соединяли с иглой. Образец медленно вводили, слегка надавливая на поршень шприца. После этого разрез зашивали, и эту стадию завершали. Через один сутки после введения образцов наблюдали за болевыми реакциями с использованием теста с волосками фон Фрея.
Экспериментальный пример 6.3. Наблюдение за болевыми реакциями с помощью теста с волосками фон Фрея
Тест с волосками фон Фрея проводили аналогично методу, описанному в экспериментальном примере 3.3. В результате было обнаружено, что анальгетический эффект был выше, когда плазмиды pAAV-GAD65 и pAAV-GDNF или pAAV-IL-10 и pAAV-GDNF вводили совместно по сравнению с введением плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности (фиг. 12).
Экспериментальный пример 7. Сравнение анальгетической эффективности при введении плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности и совместном введении плазмид pAAV-GAD65, pAAV-IL-10 и pAAV-GDNF
Плазмиды, полученные в примере 1, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Плазмиды pAAV-GAD65, pAAV-IL-10, pAAV-GDNF и pVAX1 разводили в Трис-ЭДТА буфере для получения концентраций, указанных в таблице 5. Плазмиду pVAX1 вводили в том же количестве, что и другие плазмиды. Сообщения о том, что плазмида pVAX1 обладает анальгетической эффективностью, отсутствуют. Нужные плазмиды смешивали в соответствии с содержанием, указанным в таблице 5, и вводили в количестве 30 мкг/50 мкл на животное.
|
Животным, которых использовали в качестве модели боли, полученным аналогично методу, описанному в экспериментальном примере 6.2, вводили образцы, приготовленные согласно титрам вирусов, указанным в таблице 5. Затем проводили тест с волосками фон Фрея аналогично методу, описанному в экспериментальном примере 3.3., для наблюдения за болевыми реакциями.
В результате было установлено, что анальгетический эффект был выше, когда все плазмиды pAAV-GAD65, pAAV5-IL-10 и pAAV5-GDNF вводили совместно по сравнению с введением плазмид pAAV-GAD65, pAAV-IL-10 или pAAV-GDNF по отдельности (фиг. 13).
Экспериментальный пример 8. Сравнение анальгетической эффективности при совместном введении плазмид pAAV-GAD65 и pAAV-GDNF, или pAAV-IL-10 и pAAV-GDNF и совместном введении плазмид pAAV-GAD65, pAAV-IL-10 и pAAV-GDNF
Плазмиды, полученные в примере 1, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Плазмиды pAAV-GAD65, pAAV-IL-10, pAAV-GDNF и pVAX1 разводили в Трис-ЭДТА буфере для получения концентраций, указанных в таблице 6. Плазмиду pVAX1 вводили в той же концентрации и количестве, что и другие плазмиды. Сообщения о том, что плазмида pVAX1 обладает анальгетической эффективностью, отсутствуют. Нужные плазмиды смешивали в соответствии с содержанием, указанным в таблице 6, и вводили в количестве 30 мкг/50 мкл на животное.
|
Животным, которых использовали в качестве модели боли, полученным аналогично методу, описанному в экспериментальном примере 6.2, вводили образцы, приготовленные согласно титрам вирусов, указанным в таблице 6. Затем проводили тест с волосками фон Фрея аналогично методу, описанному в экспериментальном примере 6.3., для наблюдения за болевыми реакциями.
В результате было установлено, что анальгетический эффект был выше, когда плазмиды pAAV-GAD65, pAAV-IL-10 и pAAV-GDNF вводили совместно по сравнению с совместным введением плазмид pAAV-GAD65 и pAAV-GDNF или pAAV-IL-10 и pAAV-GDNF (фиг. 14).
Экспериментальный пример 9. Сравнение анальгетической эффективности при совместном введении вирусов AAV-GAD65-modi и AAV-GDNF-IL-10 и совместном введении вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF
Аденоассоциированные вирусы, полученные в примере 2, использовали для теста. За 30 мин до проведения эксперимента с введением животным реагенты, хранившиеся при -80°С, оттаивали при комнатной температуре и готовили перемешиванием на вортексе. Вирусы AAV-GAD65-modi, AAV-GDNF-IL-10, AAV-GAD65, AAV-IL-10 и AAV-GDNF разводили в PBS для получения титров, указанных в таблице 7. Нужные вирусы смешивали в соответствии с титрами, указанными в таблице 7, и вводили в количестве 1,0 × 109 vg/5 мкл или 1,5 × 109 vg/5 мкл на животное.
|
Животным, которых использовали в качестве модели боли, полученным аналогично методу, описанному в экспериментальном примере 3.2, вводили образцы, приготовленные согласно титрам вирусов, указанным в таблице 7. Затем проводили тест с волосками фон Фрея аналогично методу, описанному в экспериментальном примере 3.3., для наблюдения за болевыми реакциями.
В результате было установлено, что анальгетический эффект был выше, когда вирусы AAV-GAD65-modi и AAV-GDNF-IL-10 вводили совместно по сравнению с совместным введением всех вирусов AAV-GAD65, AAV-IL-10 и AAV-GDNF (фиг. 15).
--->
Список последовательностей
<110> KOLON LIFE SCIENCE, INC.
<120> КОМПОЗИЦИЯ ДЛЯ ОБЛЕГЧЕНИЯ ИЛИ ЛЕЧЕНИЯ БОЛИ
<130> PCB706093KLS/PCT
<150> KR 2016/0143519
<151> 2016-10-31
<160> 49
<170> KopatentIn 2.0
<210> 1
<211> 585
<212> Белок
<213> Homo sapiens
<400> 1
Met Ala Ser Pro Gly Ser Gly Phe Trp Ser Phe Gly Ser Glu Asp Gly
1 5 10 15
Ser Gly Asp Ser Glu Asn Pro Gly Thr Ala Arg Ala Trp Cys Gln Val
20 25 30
Ala Gln Lys Phe Thr Gly Gly Ile Gly Asn Lys Leu Cys Ala Leu Leu
35 40 45
Tyr Gly Asp Ala Glu Lys Pro Ala Glu Ser Gly Gly Ser Gln Pro Pro
50 55 60
Arg Ala Ala Ala Arg Lys Ala Ala Cys Ala Cys Asp Gln Lys Pro Cys
65 70 75 80
Ser Cys Ser Lys Val Asp Val Asn Tyr Ala Phe Leu His Ala Thr Asp
85 90 95
Leu Leu Pro Ala Cys Asp Gly Glu Arg Pro Thr Leu Ala Phe Leu Gln
100 105 110
Asp Val Met Asn Ile Leu Leu Gln Tyr Val Val Lys Ser Phe Asp Arg
115 120 125
Ser Thr Lys Val Ile Asp Phe His Tyr Pro Asn Glu Leu Leu Gln Glu
130 135 140
Tyr Asn Trp Glu Leu Ala Asp Gln Pro Gln Asn Leu Glu Glu Ile Leu
145 150 155 160
Met His Cys Gln Thr Thr Leu Lys Tyr Ala Ile Lys Thr Gly His Pro
165 170 175
Arg Tyr Phe Asn Gln Leu Ser Thr Gly Leu Asp Met Val Gly Leu Ala
180 185 190
Ala Asp Trp Leu Thr Ser Thr Ala Asn Thr Asn Met Phe Thr Tyr Glu
195 200 205
Ile Ala Pro Val Phe Val Leu Leu Glu Tyr Val Thr Leu Lys Lys Met
210 215 220
Arg Glu Ile Ile Gly Trp Pro Gly Gly Ser Gly Asp Gly Ile Phe Ser
225 230 235 240
Pro Gly Gly Ala Ile Ser Asn Met Tyr Ala Met Met Ile Ala Arg Phe
245 250 255
Lys Met Phe Pro Glu Val Lys Glu Lys Gly Met Ala Ala Leu Pro Arg
260 265 270
Leu Ile Ala Phe Thr Ser Glu His Ser His Phe Ser Leu Lys Lys Gly
275 280 285
Ala Ala Ala Leu Gly Ile Gly Thr Asp Ser Val Ile Leu Ile Lys Cys
290 295 300
Asp Glu Arg Gly Lys Met Ile Pro Ser Asp Leu Glu Arg Arg Ile Leu
305 310 315 320
Glu Ala Lys Gln Lys Gly Phe Val Pro Phe Leu Val Ser Ala Thr Ala
325 330 335
Gly Thr Thr Val Tyr Gly Ala Phe Asp Pro Leu Leu Ala Val Ala Asp
340 345 350
Ile Cys Lys Lys Tyr Lys Ile Trp Met His Val Asp Ala Ala Trp Gly
355 360 365
Gly Gly Leu Leu Met Ser Arg Lys His Lys Trp Lys Leu Ser Gly Val
370 375 380
Glu Arg Ala Asn Ser Val Thr Trp Asn Pro His Lys Met Met Gly Val
385 390 395 400
Pro Leu Gln Cys Ser Ala Leu Leu Val Arg Glu Glu Gly Leu Met Gln
405 410 415
Asn Cys Asn Gln Met His Ala Ser Tyr Leu Phe Gln Gln Asp Lys His
420 425 430
Tyr Asp Leu Ser Tyr Asp Thr Gly Asp Lys Ala Leu Gln Cys Gly Arg
435 440 445
His Val Asp Val Phe Lys Leu Trp Leu Met Trp Arg Ala Lys Gly Thr
450 455 460
Thr Gly Phe Glu Ala His Val Asp Lys Cys Leu Glu Leu Ala Glu Tyr
465 470 475 480
Leu Tyr Asn Ile Ile Lys Asn Arg Glu Gly Tyr Glu Met Val Phe Asp
485 490 495
Gly Lys Pro Gln His Thr Asn Val Cys Phe Trp Tyr Ile Pro Pro Ser
500 505 510
Leu Arg Thr Leu Glu Asp Asn Glu Glu Arg Met Ser Arg Leu Ser Lys
515 520 525
Val Ala Pro Val Ile Lys Ala Arg Met Met Glu Tyr Gly Thr Thr Met
530 535 540
Val Ser Tyr Gln Pro Leu Gly Asp Lys Val Asn Phe Phe Arg Met Val
545 550 555 560
Ile Ser Asn Pro Ala Ala Thr His Gln Asp Ile Asp Phe Leu Ile Glu
565 570 575
Glu Ile Glu Arg Leu Gly Gln Asp Leu
580 585
<210> 2
<211> 2824
<212> ДНК
<213> Homo sapiens
<400> 2
gcggccgccc gcacttcccg cctctggctc gcccgaggac gcgctggcac gcctcccacc 60
ccctcactct gactccagct ggcgtgcatg gtctgcctcg catcctcacg actcagctcc 120
ctccctctct cgtgtttttt tcctccgccg ccccctcatt catccccact gggctccctt 180
tccctcaaat gctctggggc tctccgcgct ttcctgagtc cgggctccga ggacccttag 240
gtagtcccgg tctcttttaa agctccccgg cttccaaagg gttgccacgt ccctaaaccc 300
tgtctccagc tcgcatacac acacgcacag acacgcacgt tttctgttcc tgcgtgacac 360
ccgccctcgc cgctcggccc cgccggtccc cgcgcggtgc cctcctcccg ccacacgggc 420
acgcacgcgc gcgcagggcc aagcccgagg cagctcgccc gcagctcgca ctcgcaggcg 480
acctgctcca gtctccaaag ccgatggcat ctccgggctc tggcttttgg tctttcgggt 540
cggaagatgg ctctggggat tccgagaatc ccggcacagc gcgagcctgg tgccaagtgg 600
ctcagaagtt cacgggcggc atcggaaaca aactgtgcgc cctgctctac ggagacgccg 660
agaagccggc ggagagcggc gggagccaac ccccgcgggc cgccgcccgg aaggccgcct 720
gcgcctgcga ccagaagccc tgcagctgct ccaaagtgga tgtcaactac gcgtttctcc 780
atgcaacaga cctgctgccg gcgtgtgatg gagaaaggcc cactttggcg tttctgcaag 840
atgttatgaa cattttactt cagtatgtgg tgaaaagttt cgatagatca accaaagtga 900
ttgatttcca ttatcctaat gagcttctcc aagaatataa ttgggaattg gcagaccaac 960
cacaaaattt ggaggaaatt ttgatgcatt gccaaacaac tctaaaatat gcaattaaaa 1020
cagggcatcc tagatacttc aatcaacttt ctactggttt ggatatggtt ggattagcag 1080
cagactggct gacatcaaca gcaaatacta acatgttcac ctatgaaatt gctccagtat 1140
ttgtgctttt ggaatatgtc acactaaaga aaatgagaga aatcattggc tggccagggg 1200
gctctggcga tgggatattt tctcccggtg gcgccatatc taacatgtat gccatgatga 1260
tcgcacgctt taagatgttc ccagaagtca aggagaaagg aatggctgct cttcccaggc 1320
tcattgcctt cacgtctgaa catagtcatt tttctctcaa gaagggagct gcagccttag 1380
ggattggaac agacagcgtg attctgatta aatgtgatga gagagggaaa atgattccat 1440
ctgatcttga aagaaggatt cttgaagcca aacagaaagg gtttgttcct ttcctcgtga 1500
gtgccacagc tggaaccacc gtgtacggag catttgaccc cctcttagct gtcgctgaca 1560
tttgcaaaaa gtataagatc tggatgcatg tggatgcagc ttggggtggg ggattactga 1620
tgtcccgaaa acacaagtgg aaactgagtg gcgtggagag ggccaactct gtgacgtgga 1680
atccacacaa gatgatggga gtccctttgc agtgctctgc tctcctggtt agagaagagg 1740
gattgatgca gaattgcaac caaatgcatg cctcctacct ctttcagcaa gataaacatt 1800
atgacctgtc ctatgacact ggagacaagg ccttacagtg cggacgccac gttgatgttt 1860
ttaaactatg gctgatgtgg agggcaaagg ggactaccgg gtttgaagcg catgttgata 1920
aatgtttgga gttggcagag tatttataca acatcataaa aaaccgagaa ggatatgaga 1980
tggtgtttga tgggaagcct cagcacacaa atgtctgctt ctggtacatt cctccaagct 2040
tgcgtactct ggaagacaat gaagagagaa tgagtcgcct ctcgaaggtg gctccagtga 2100
ttaaagccag aatgatggag tatggaacca caatggtcag ctaccaaccc ttgggagaca 2160
aggtcaattt cttccgcatg gtcatctcaa acccagcggc aactcaccaa gacattgact 2220
tcctgattga agaaatagaa cgccttggac aagatttata ataaccttgc tcaccaagct 2280
gttccacttc tctagagaac atgccctcag ctaagccccc tactgagaaa cttcctttga 2340
gaattgtgcg acttcacaaa atgcaaggtg aacaccactt tgtctctgag aacagacgtt 2400
accaattatg gagtgtcacc agctgccaaa atcgtaggtg ttggctctgc tggtcactgg 2460
agtagttgct actcttcaga atatggacaa agaaggcaca ggtgtaaata tagtagcagg 2520
atgaggaacc tcaaactggg tatcattttg cacgtgctct tctgttctca aatgctaaat 2580
gcaaacactg tgtatttatt agttaggtgt gccaaactac cgttcccaaa ttggtgtttc 2640
tgaatgacat caacattccc ccaacattac tccattacta aagacagaaa aaaataaaaa 2700
cataaaatat acaaacatgt ggcaacctgt tcttcctacc aaatataaac ttgtgtatga 2760
tccaagtatt ttatctgtgt tgtctctcta aacccaaata aatgtgtaaa tgtggacaca 2820
tctc 2824
<210> 3
<211> 1758
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Оптимизированный ген человеческой GAD65
<400> 3
atggcatctc cgggctccgg cttttggtcc ttcgggtcgg aagatggctc aggggattcc 60
gagaatcccg gcacagcgcg ggcctggtgt caagtggctc agaagttcac gggcggcatc 120
ggaaacaaac tgtgtgccct gctctacggc gacgccgaga agcccgcaga gagcggcggg 180
agccaacccc cgcgggccgc cgcccggaag gccgcctgcg cctgtgacca gaagccctgc 240
tcatgcagca aggtagatgt caactacgcg tttctccatg ccacagatct gctgccggct 300
tgcgacggtg aaaggcccac tttggccttt ctgcaggatg ttatgaacat tctgctgcag 360
tacgtggtga aaagtttcga ccggtcaacc aaagtgatcg actttcacta tcctaatgaa 420
cttctccagg agtacaattg ggagctggct gaccagccac agaacctgga ggaaatcttg 480
atgcattgcc aaactactct aaaatatgca attaaaacag gccatcctag atacttcaac 540
cagctttcta ccggtttgga tatggtgggg ctggcagccg actggctgac atccaccgca 600
aataccaaca tgttcaccta tgagatcgct cctgtcttcg tgcttttgga atacgtcacc 660
ctaaagaaga tgcgtgaaat cattggctgg ccaggaggct ctggtgatgg tatattttct 720
cccggcggcg cgatctctaa catgtatgcc atgatgatcg cacgctttaa gatgttccca 780
gaagtcaagg agaaaggaat ggctgctctt cccaggctca ttgccttcac gagtgaacac 840
agtcactttt ccctcaagaa gggggctgcc gccttaggga tcggaacaga cagcgtgatt 900
ctgataaagt gcgacgagag agggaaaatg attccatctg atcttgagag aaggattctt 960
gaagccaaac agaaagggtt tgtccctttc ctcgtgagtg ccacagctgg aaccaccgtg 1020
tacggcgcat ttgaccccct cttagctgtc gcggatatat gtaagaagta taagatctgg 1080
atgcacgtgg atgctgcttg gggtggggga ttactgatgt ccaggaaaca caagtggaaa 1140
ctgtctggcg tggagcgcgc caacagcgtg acgtggaatc cacacaaaat gatgggagtc 1200
cctttgcagt gctctgctct cctggttcga gaagagggac tgatgcagaa ttgcaaccaa 1260
atgcatgcct cctacctctt tcagcaggat aaacattatg acctgtctta cgacactggt 1320
gacaaggccc tgcagtgtgg gcgccacgtt gatgtattca agctatggct gatgtggagg 1380
gcaaagggga ctaccggttt tgaagcccat gttgacaaat gtctggagtt ggcagagtat 1440
ttatacaata tcataaaaaa ccgagaagga tatgagatgg tgtttgatgg caagcctcag 1500
cacacaaatg tctgcttctg gtacatccct cccagcctac gtactctgga ggacaacgaa 1560
gagagaatga gtcgcctctc gaaggtggct ccagtgatta aagccagaat gatggagtat 1620
ggaaccacaa tggtcagcta ccaacccttg ggggacaagg taaatttctt ccgcatggtc 1680
atctcaaacc cagcggcaac tcaccaagac attgatttcc tgattgaaga gatcgagcgg 1740
ctcggccagg atctgtga 1758
<210> 4
<211> 594
<212> Белок
<213> Homo sapiens
<400> 4
Met Ala Ser Ser Thr Pro Ser Ser Ser Ala Thr Ser Ser Asn Ala Gly
1 5 10 15
Ala Asp Pro Asn Thr Thr Asn Leu Arg Pro Thr Thr Tyr Asp Thr Trp
20 25 30
Cys Gly Val Ala His Gly Cys Thr Arg Lys Leu Gly Leu Lys Ile Cys
35 40 45
Gly Phe Leu Gln Arg Thr Asn Ser Leu Glu Glu Lys Ser Arg Leu Val
50 55 60
Ser Ala Phe Lys Glu Arg Gln Ser Ser Lys Asn Leu Leu Ser Cys Glu
65 70 75 80
Asn Ser Asp Arg Asp Ala Arg Phe Arg Arg Thr Glu Thr Asp Phe Ser
85 90 95
Asn Leu Phe Ala Arg Asp Leu Leu Pro Ala Lys Asn Gly Glu Glu Gln
100 105 110
Thr Val Gln Phe Leu Leu Glu Val Val Asp Ile Leu Leu Asn Tyr Val
115 120 125
Arg Lys Thr Phe Asp Arg Ser Thr Lys Val Leu Asp Phe His His Pro
130 135 140
His Gln Leu Leu Glu Gly Met Glu Gly Phe Asn Leu Glu Leu Ser Asp
145 150 155 160
His Pro Glu Ser Leu Glu Gln Ile Leu Val Asp Cys Arg Asp Thr Leu
165 170 175
Lys Tyr Gly Val Arg Thr Gly His Pro Arg Phe Phe Asn Gln Leu Ser
180 185 190
Thr Gly Leu Asp Ile Ile Gly Leu Ala Gly Glu Trp Leu Thr Ser Thr
195 200 205
Ala Asn Thr Asn Met Phe Thr Tyr Glu Ile Ala Pro Val Phe Val Leu
210 215 220
Met Glu Gln Ile Thr Leu Lys Lys Met Arg Glu Ile Val Gly Trp Ser
225 230 235 240
Ser Lys Asp Gly Asp Gly Ile Phe Ser Pro Gly Gly Ala Ile Ser Asn
245 250 255
Met Tyr Ser Ile Met Ala Ala Arg Tyr Lys Tyr Phe Pro Glu Val Lys
260 265 270
Thr Lys Gly Met Ala Ala Val Pro Lys Leu Val Leu Phe Thr Ser Glu
275 280 285
Gln Ser His Tyr Ser Ile Lys Lys Ala Gly Ala Ala Leu Gly Phe Gly
290 295 300
Thr Asp Asn Val Ile Leu Ile Lys Cys Asn Glu Arg Gly Lys Ile Ile
305 310 315 320
Pro Ala Asp Phe Glu Ala Lys Ile Leu Glu Ala Lys Gln Lys Gly Tyr
325 330 335
Val Pro Phe Tyr Val Asn Ala Thr Ala Gly Thr Thr Val Tyr Gly Ala
340 345 350
Phe Asp Pro Ile Gln Glu Ile Ala Asp Ile Cys Glu Lys Tyr Asn Leu
355 360 365
Trp Leu His Val Asp Ala Ala Trp Gly Gly Gly Leu Leu Met Ser Arg
370 375 380
Lys His Arg His Lys Leu Asn Gly Ile Glu Arg Ala Asn Ser Val Thr
385 390 395 400
Trp Asn Pro His Lys Met Met Gly Val Leu Leu Gln Cys Ser Ala Ile
405 410 415
Leu Val Lys Glu Lys Gly Ile Leu Gln Gly Cys Asn Gln Met Cys Ala
420 425 430
Gly Tyr Leu Phe Gln Pro Asp Lys Gln Tyr Asp Val Ser Tyr Asp Thr
435 440 445
Gly Asp Lys Ala Ile Gln Cys Gly Arg His Val Asp Ile Phe Lys Phe
450 455 460
Trp Leu Met Trp Lys Ala Lys Gly Thr Val Gly Phe Glu Asn Gln Ile
465 470 475 480
Asn Lys Cys Leu Glu Leu Ala Glu Tyr Leu Tyr Ala Lys Ile Lys Asn
485 490 495
Arg Glu Glu Phe Glu Met Val Phe Asn Gly Glu Pro Glu His Thr Asn
500 505 510
Val Cys Phe Trp Tyr Ile Pro Gln Ser Leu Arg Gly Val Pro Asp Ser
515 520 525
Pro Gln Arg Arg Glu Lys Leu His Lys Val Ala Pro Lys Ile Lys Ala
530 535 540
Leu Met Met Glu Ser Gly Thr Thr Met Val Gly Tyr Gln Pro Gln Gly
545 550 555 560
Asp Lys Ala Asn Phe Phe Arg Met Val Ile Ser Asn Pro Ala Ala Thr
565 570 575
Gln Ser Asp Ile Asp Phe Leu Ile Glu Glu Ile Glu Arg Leu Gly Gln
580 585 590
Asp Leu
<210> 5
<211> 1784
<212> ДНК
<213> Homo sapiens
<400> 5
atggcgtctc gaccccatct tcgtccgcaa cctcctcgaa cgcgggagcg gaccccaata 60
ccactaacct gcgccccaca acgtacgata cctggtgcgg cgtggcccat ggatgcacca 120
gaaaactggg gctcaagatc tgcggcttct tgcaaaggac caacagcctg gaagagaaga 180
gtcgccttgt gagtgccttc aaggagaggc aatcctccaa gaacctgctt tcctgtgaaa 240
acagcgaccg ggatgcccgc ttccggcgca cagagactga cttctctaat ctgtttgcta 300
gagatctgct tccggctaag aacggtgagg agcaaaccgt gcaattcctc ctggaagtgg 360
tggacatact cctcaactat gtccgcaaga catttgatcg ctccaccaag gtgctggact 420
ttcatcaccc acaccagttg ctggaaggca tggagggctt caacttggag ctctctgacc 480
accccgagtc cctggagcag atcctggttg actgcagaga caccttgaag tatggggttc 540
gcacaggtca tcctcgattt ttcaaccagc tctccactgg attggatatt attggcctag 600
ctggagaatg gctgacatca acggccaata ccaacatgtt tacatatgaa attgcaccag 660
tgtttgtcct catggaacaa ataacactta agaagatgag agagatagtt ggatggtcaa 720
gtaaagatgg tgatgggata ttttctcctg ggggcgccat atccaacatg tacagcatca 780
tggctgctcg ctacaagtac ttcccggaag ttaagacaaa gggcatggcg gctgtgccta 840
aactggtcct cttcacctca gaacagagtc actattccat aaagaaagct ggggctgcac 900
ttggctttgg aactgacaat gtgattttga taaagtgcaa tgaaaggggg aaaataattc 960
cagctgattt tgaggcaaaa attcttgaag ccaaacagaa gggatatgtt cccttttatg 1020
tcaatgcaac tgctggcacg actgtttatg gagcttttga tccgatacaa gagattgcag 1080
atatatgtga gaaatataac ctttggttgc atgtcgatgc tgcctgggga ggtgggctgc 1140
tcatgtccag gaagcaccgc cataaactca acggcataga aagggccaac tcagtcacct 1200
ggaaccctca caagatgatg ggcgtgctgt tgcagtgctc tgccattctc gtcaaggaaa 1260
agggtatact ccaaggatgc aaccagatgt gtgcaggata cctcttccag ccagacaagc 1320
agtatgatgt ctcctacgac accggggaca aggcaattca gtgtggccgc cacgtggata 1380
tcttcaagtt ctggctgatg tggaaagcaa agggcacagt gggatttgaa aaccagatca 1440
acaaatgcct ggaactggct gaatacctct atgccaagat taaaaacaga gaagaatttg 1500
agatggtttt caatggcgag cctgagcaca caaacgtctg tttttggtat attccacaaa 1560
gcctcagggg tgtgccagac agccctcaac gacgggaaaa gctacacaag gtggctccaa 1620
aaatcaaagc cctgatgatg gagtcaggta cgaccatggt tggctaccag ccccaagggg 1680
acaaggccaa cttcttccgg atggtcatct ccaacccagc cgctacccag tctgacattg 1740
acttcctcat tgaggagata gaaagactgg gccaggatct gtaa 1784
<210> 6
<211> 178
<212> Белок
<213> rattus norvegicus
<400> 6
Met Pro Gly Ser Ala Leu Leu Cys Cys Leu Leu Leu Leu Ala Gly Val
1 5 10 15
Lys Thr Ser Lys Gly His Ser Ile Arg Gly Asp Asn Asn Cys Thr His
20 25 30
Phe Pro Val Ser Gln Thr His Met Leu Arg Glu Leu Arg Ala Ala Phe
35 40 45
Ser Gln Val Lys Thr Phe Phe Gln Lys Lys Asp Gln Leu Asp Asn Ile
50 55 60
Leu Leu Thr Asp Ser Leu Leu Gln Asp Phe Lys Gly Tyr Leu Gly Cys
65 70 75 80
Gln Ala Leu Ser Glu Met Ile Lys Phe Tyr Leu Val Glu Val Met Pro
85 90 95
Gln Ala Glu Asn His Gly Pro Glu Ile Lys Glu His Leu Asn Ser Leu
100 105 110
Gly Glu Lys Leu Lys Thr Leu Trp Ile Gln Leu Arg Arg Cys His Arg
115 120 125
Phe Leu Pro Cys Glu Asn Lys Ser Lys Ala Val Glu Gln Val Lys Asn
130 135 140
Asp Phe Asn Lys Leu Gln Asp Lys Gly Val Tyr Lys Ala Met Asn Glu
145 150 155 160
Phe Asp Ile Phe Ile Asn Cys Ile Glu Ala Tyr Val Thr Leu Lys Met
165 170 175
Lys Asn
<210> 7
<211> 682
<212> ДНК
<213> rattus norvegicus
<400> 7
catgcctggc tcagcactgc tatgttgcct gctcttactg gctggagtga agaccagcaa 60
aggccattcc atccggggtg acaataactg cacccacttc ccagtcagcc agacccacat 120
gctccgagag ctgagggctg ccttcagtca agtgaagact ttctttcaaa agaaggacca 180
gctggacaac atactgctga cagattcctt actgcaggac tttaagggtt acttgggttg 240
ccaagccttg tcagaaatga tcaagtttta cctggtagaa gtgatgcccc aggcagagaa 300
ccatggccca gaaatcaagg agcatttgaa ttccctggga gagaagctga agaccctctg 360
gatacagctg cgacgctgtc atcgatttct cccctgtgag aataaaagca aggcagtgga 420
gcaggtgaag aatgatttta ataagctcca agacaaaggt gtctacaagg ccatgaatga 480
gtttgacatc ttcatcaact gcatagaagc ctacgtgaca ctcaaaatga aaaattgaac 540
cacccggcat ctactggact gcaggacata aatagagctt ctaaatctga tccagagatc 600
ttagctaacg ggagcaactc cttggaaaac ctcgtttgta cctctctcca aaatatttat 660
tacctctgat acctcagttc cc 682
<210> 8
<211> 537
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Оптимизированный ген крысиного IL-10
<400> 8
atgcctggct cagccctgct atgttgcctt ctcctgctgg cgggagtcaa gacaagcaag 60
ggccattcca tccggggaga taataactgc acccacttcc cagtctctca aacccacatg 120
ttgcgagagc tgagggctgc cttcagtcag gtgaagacgt tcttccagaa gaaggaccag 180
ctggacaaca ttctgctgac tgacagcctg ctgcaggatt tcaagggtta tttggggtgt 240
caagccctgt ctgaaatgat caagttttac ctggtagaag tgatgcccca ggcagagaat 300
catggccccg agatcaagga gcacctcaac tccctggggg agaagctgaa gaccctgtgg 360
attcagctga ggcgctgcca cagatttctc ccctgtgaaa acaagagcaa ggcagtggag 420
caggtgaaga acgattttaa taagctccag gacaagggcg tctacaaggc catgaacgag 480
ttcgacatct ttatcaactg catagaagct tacgttacac tcaagatgaa gaattga 537
<210> 9
<211> 178
<212> Белок
<213> Homo sapiens
<400> 9
Met His Ser Ser Ala Leu Leu Cys Cys Leu Val Leu Leu Thr Gly Val
1 5 10 15
Arg Ala Ser Pro Gly Gln Gly Thr Gln Ser Glu Asn Ser Cys Thr His
20 25 30
Phe Pro Gly Asn Leu Pro Asn Met Leu Arg Asp Leu Arg Asp Ala Phe
35 40 45
Ser Arg Val Lys Thr Phe Phe Gln Met Lys Asp Gln Leu Asp Asn Leu
50 55 60
Leu Leu Lys Glu Ser Leu Leu Glu Asp Phe Lys Gly Tyr Leu Gly Cys
65 70 75 80
Gln Ala Leu Ser Glu Met Ile Gln Phe Tyr Leu Glu Glu Val Met Pro
85 90 95
Gln Ala Glu Asn Gln Asp Pro Asp Ile Lys Ala His Val Asn Ser Leu
100 105 110
Gly Glu Asn Leu Lys Thr Leu Arg Leu Arg Leu Arg Arg Cys His Arg
115 120 125
Phe Leu Pro Cys Glu Asn Lys Ser Lys Ala Val Glu Gln Val Lys Asn
130 135 140
Ala Phe Asn Lys Leu Gln Glu Lys Gly Ile Tyr Lys Ala Met Ser Glu
145 150 155 160
Phe Asp Ile Phe Ile Asn Tyr Ile Glu Ala Tyr Met Thr Met Lys Ile
165 170 175
Arg Asn
<210> 10
<211> 1600
<212> ДНК
<213> Homo sapiens
<400> 10
aaaccacaag acagacttgc aaaagaaggc atgcacagct cagcactgct ctgttgcctg 60
gtcctcctga ctggggtgag ggccagccca ggccagggca cccagtctga gaacagctgc 120
acccacttcc caggcaacct gcctaacatg cttcgagatc tccgagatgc cttcagcaga 180
gtgaagactt tctttcaaat gaaggatcag ctggacaact tgttgttaaa ggagtccttg 240
ctggaggact ttaagggtta cctgggttgc caagccttgt ctgagatgat ccagttttac 300
ctggaggagg tgatgcccca agctgagaac caagacccag acatcaaggc gcatgtgaac 360
tccctggggg agaacctgaa gaccctcagg ctgaggctac ggcgctgtca tcgatttctt 420
ccctgtgaaa acaagagcaa ggccgtggag caggtgaaga atgcctttaa taagctccaa 480
gagaaaggca tctacaaagc catgagtgag tttgacatct tcatcaacta catagaagcc 540
tacatgacaa tgaagatacg aaactgagac atcagggtgg cgactctata gactctagga 600
cataaattag aggtctccaa aatcggatct ggggctctgg gatagctgac ccagcccctt 660
gagaaacctt attgtacctc tcttatagaa tatttattac ctctgatacc tcaaccccca 720
tttctattta tttactgagc ttctctgtga acgatttaga aagaagccca atattataat 780
ttttttcaat atttattatt ttcacctgtt tttaagctgt ttccataggg tgacacacta 840
tggtatttga gtgttttaag ataaattata agttacataa gggaggaaaa aaaatgttct 900
ttggggagcc aacagaagct tccattccaa gcctgaccac gctttctagc tgttgagctg 960
ttttccctga cctccctcta atttatcttg tctctgggct tggggcttcc taactgctac 1020
aaatactctt aggaagagaa accagggagc ccctttgatg attaattcac cttccagtgt 1080
ctcggaggga ttcccctaac ctcattcccc aaccacttca ttcttgaaag ctgtggccag 1140
cttgttattt ataacaacct aaatttggtt ctaggccggg cgcggtggct cacgcctgta 1200
atcccagcac tttgggaggc tgaggcgggt ggatcacttg aggtcaggag ttcctaacca 1260
gcctggtcaa catggtgaaa ccccgtctct actaaaaata caaaaattag ccgggcatgg 1320
tggcgcgcac ctgtaatccc agctacttgg gaggctgagg caagagaatt gcttgaaccc 1380
aggagatgga agttgcagtg agctgatatc atgcccctgt actccagcct gggtgacaga 1440
gcaagactct gtctcaaaaa ataaaaataa aaataaattt ggttctaata gaactcagtt 1500
ttaactagaa tttattcaat tcctctggga atgttacatt gtttgtctgt cttcatagca 1560
gattttaatt ttgaataaat aaatgtatct tattcacatc 1600
<210> 11
<211> 185
<212> Белок
<213> Homo sapiens
<400> 11
Met Lys Leu Trp Asp Val Val Ala Val Cys Leu Val Leu Leu His Thr
1 5 10 15
Ala Ser Ala Phe Pro Leu Pro Ala Ala Asn Met Pro Glu Asp Tyr Pro
20 25 30
Asp Gln Phe Asp Asp Val Met Asp Phe Ile Gln Ala Thr Ile Lys Arg
35 40 45
Leu Lys Arg Ser Pro Asp Lys Gln Met Ala Val Leu Pro Arg Arg Glu
50 55 60
Arg Asn Arg Gln Ala Ala Ala Ala Asn Pro Glu Asn Ser Arg Gly Lys
65 70 75 80
Gly Arg Arg Gly Gln Arg Gly Lys Asn Arg Gly Cys Val Leu Thr Ala
85 90 95
Ile His Leu Asn Val Thr Asp Leu Gly Leu Gly Tyr Glu Thr Lys Glu
100 105 110
Glu Leu Ile Phe Arg Tyr Cys Ser Gly Ser Cys Asp Ala Ala Glu Thr
115 120 125
Thr Tyr Asp Lys Ile Leu Lys Asn Leu Ser Arg Asn Arg Arg Leu Val
130 135 140
Ser Asp Lys Val Gly Gln Ala Cys Cys Arg Pro Ile Ala Phe Asp Asp
145 150 155 160
Asp Leu Ser Phe Leu Asp Asp Asn Leu Val Tyr His Ile Leu Arg Lys
165 170 175
His Ser Ala Lys Arg Cys Gly Cys Ile
180 185
<210> 12
<211> 558
<212> ДНК
<213> Homo sapiens
<400> 12
atgaagttat gggatgtcgt ggctgtctgc ctggtgctgc tccacaccgc gtccgccttc 60
ccgctgcccg ccgcaaatat gccagaggat tatcctgatc agttcgatga tgtcatggat 120
tttattcaag ccaccattaa aagactgaaa aggtcaccag ataaacaaat ggcagtgctt 180
cctagaagag agcggaatcg gcaggctgca gctgccaacc cagagaattc cagaggaaaa 240
ggtcggagag gccagagggg caaaaaccgg ggttgtgtct taactgcaat acatttaaat 300
gtcactgact tgggtctggg ctatgaaacc aaggaggaac tgatttttag gtactgcagc 360
ggctcttgcg atgcagctga gacaacgtac gacaaaatat tgaaaaactt atccagaaat 420
agaaggctgg tgagtgacaa agtagggcag gcatgttgca gacccatcgc ctttgatgat 480
gacctgtcgt ttttagatga taacctggtt taccatattc taagaaagca ttccgctaaa 540
aggtgtggat gtatctga 558
<210> 13
<211> 558
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Оптимизированный ген человеческого GDNF
<400> 13
atgaaacttt gggacgtggt ggctgtctgc ctggtgctcc tccacaccgc cagtgcgttt 60
ccgctgcccg ccgctaacat gccagaggat tatcctgatc agttcgatga tgttatggac 120
ttcattcaag ccacaatcaa gcggctgaaa cgatcaccag ataaacagat ggcagtgctt 180
cctcgccgcg agcgtaatcg gcaggctgca gcagccaatc ccgagaattc ccgaggaaaa 240
gggcgcaggg gtcagagggg caagaaccgg gggtgtgtcc tgactgcaat acatttaaac 300
gtgactgact tgggtctggg ctatgagacc aaggaagaac tcattttcag gtactgcagc 360
ggctcttgcg atgccgcgga aacaacgtac gacaaaatct tgaagaacct ctccagaaac 420
agaaggctgg tgagtgacaa ggtaggacag gcctgttgca gacccatcgc ctttgacgac 480
gatctgagct ttctggatga caatctggtt taccacatcc tacggaagca ttctgctaaa 540
agatgtggat gtatttga 558
<210> 14
<211> 537
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Оптимизированный ген человеческого IL-10
<400> 14
atgcacagct cagcactgct gtgctgcctg gtcctgctga caggggtgag ggcaagccca 60
ggccagggaa cccaatctga gaacagctgc acccacttcc ctggcaatct gcctaacatg 120
ctgcgcgacc tccgagatgc cttcagcaga gtgaagactt ttttccagat gaaggatcag 180
ctggacaacc tgctgctgaa ggagtccctc ctggaggact ttaagggcta cctgggatgc 240
caggccctgt ctgagatgat ccaattctac ctggaagaag ttatgcccca ggctgagaac 300
caggacccag acattaaggc ccatgtcaac tccctggggg aaaatctgaa gaccctcagg 360
ctgcggctac ggcgctgtca ccgttttctg ccctgtgaga ataagagcaa ggctgtggag 420
caggtgaaga acgccttcaa taagctccag gagaagggta tctacaaagc gatgagtgaa 480
tttgatatct tcattaatta tatagaagct tatatgacaa tgaaaatcag aaactga 537
<210> 15
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации CMV промотора (F-JDK)
<400> 15
ttcggccgtc gaggagcttg gcccattg 28
<210> 16
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации CMV промотора (F-JDK)
<400> 16
gacgtcgacc tagctagcga attcggggcc gcggag 36
<210> 17
<211> 34
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации SV40pA промотора (F-SV40pA)
<400> 17
ccatcgatca gacatgataa gatacattga tgag 34
<210> 18
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации SV40pA промотора (R-SV40pA)
<400> 18
gacgtcgacg cggccgctac cacatttgta gaggttttac ttg 43
<210> 19
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации гена резистентности к канамицину (F-Kan)
<400> 19
aggcgccatg agccatattc aacgggaa 28
<210> 20
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации гена резистентности к канамицину (F-Kan)
<400> 20
ttcatgatta gaaaaactca tcgagcatc 29
<210> 21
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации LITR и CMV (F-ITR)
<400> 21
atggcgcgcc cctggccttt tgctggcc 28
<210> 22
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для амплификации SV40pA и RITR (R-ITR)
<400> 22
atggatccgc tagtaaatac cgcatcag 28
<210> 23
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для амплификации rIL-10 (F-rIL-10)
<400> 23
ccgctagcgc caccatgcct 20
<210> 24
<211> 39
<212> ДНК
<213> Искусственная последовательность<220>
<223> Обратный праймер для амплификации rIL-10 (R-rIL-10)
<400> 24
gacgtcgacg ccatcgatgg cttaattaat caattcttc 39
<210> 25
<211> 40
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для WPRE_Pac1_F
<400> 25
ggtggtttaa ttaaaatcaa cctctggatt acaaaatttg 40
<210> 26
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для WPRE_modi_Hpa1_R
<400> 26
ggtggtgtta acgacaacac cacggaattg 30
<210> 27
<211> 43
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для SV40-CMV-sCAG-bGHpA-Infu-F
<400> 27
cctgcggccg gtcgactacc acatttgtag aggttttact tgc 43
<210> 28
<211> 35
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Обратный праймер для SV40-CMV-sCAG-bGHpA-Infu-R
<400> 28
aataatcaat gtcgactcga ggagcttggc ccatt 35
<210> 29
<211> 42
<212> ДНК
<213> Искусственная последовательность
<220>
<223> нуклеотидная последовательность Stuffer scramble
<400> 29
gtcgacggta tcgataagct tgatatcgaa ttcctgcagc cc 42
<210> 30
<211> 46
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для Stuffer_scramble_F
<400> 30
ctaggtcgac ggtatcgata agcttgatat cgaattcctg cagccc 46
<210> 31
<211> 46
<212> ДНК
<213> Искусственная последовательность
<220>
<223> Прямой праймер для Stuffer_scramble_R
<400> 31
ctaggggctg caggaattcg atatcaagct tatcgatacc gtcgac 46
<210> 32
<211> 585
<212> Белок
<213> Canis lupus
<400> 32
Met Ala Ser Pro Gly Ser Gly Phe Trp Ser Phe Gly Ser Glu Asp Gly
1 5 10 15
Ser Gly Asp Pro Glu Asn Pro Ser Thr Ala Arg Ala Trp Cys Gln Val
20 25 30
Ala Gln Lys Phe Thr Gly Gly Ile Gly Asn Lys Leu Cys Ala Leu Leu
35 40 45
Tyr Gly Asp Ala Glu Lys Pro Ala Glu Ser Gly Gly Ser Glu Pro Pro
50 55 60
Arg Ala Thr Ser Arg Lys Ala Ala Cys Ala Cys Asn Gln Lys Pro Cys
65 70 75 80
Ser Cys Pro Lys Ala Glu Val Asn Tyr Ala Phe Leu His Ala Thr Asp
85 90 95
Leu Leu Pro Ala Cys Asp Gly Glu Arg Pro Thr Leu Ala Phe Leu Gln
100 105 110
Asp Val Met Asp Ile Leu Leu Gln Tyr Val Val Lys Ser Phe Asp Arg
115 120 125
Ser Thr Lys Val Ile Asp Phe His Tyr Pro Asn Glu Leu Leu Gln Glu
130 135 140
Tyr Asn Trp Glu Leu Ala Asp Gln Pro Gln Asn Leu Glu Glu Ile Leu
145 150 155 160
Met His Cys Gln Thr Thr Leu Lys Tyr Ala Ile Lys Thr Gly His Pro
165 170 175
Arg Tyr Phe Asn Gln Leu Ser Thr Gly Leu Asp Met Val Gly Leu Ala
180 185 190
Ala Asp Trp Leu Thr Ser Thr Ala Asn Thr Asn Met Phe Thr Tyr Glu
195 200 205
Ile Ala Pro Val Phe Val Leu Leu Glu Tyr Val Thr Leu Lys Lys Met
210 215 220
Arg Glu Ile Ile Gly Trp Pro Gly Gly Ser Gly Asp Gly Ile Phe Ser
225 230 235 240
Pro Gly Gly Ala Ile Ser Asn Met Tyr Ala Met Leu Ile Ala Arg Phe
245 250 255
Lys Met Phe Pro Glu Val Lys Glu Lys Gly Met Ala Ala Val Pro Arg
260 265 270
Leu Ile Ala Phe Thr Ser Glu His Ser His Phe Ser Leu Lys Lys Gly
275 280 285
Ala Ala Ala Leu Gly Ile Gly Thr Asp Ser Val Ile Leu Ile Lys Cys
290 295 300
Asp Glu Arg Gly Lys Met Val Pro Ser Asp Leu Glu Arg Arg Ile Leu
305 310 315 320
Glu Ala Lys Gln Lys Gly Phe Val Pro Phe Leu Val Ser Ala Thr Ala
325 330 335
Gly Thr Thr Val Tyr Gly Ala Phe Asp Pro Leu Leu Ala Val Ala Asp
340 345 350
Ile Cys Lys Lys Tyr Lys Ile Trp Met His Val Asp Ala Ala Trp Gly
355 360 365
Gly Gly Leu Leu Met Ser Arg Lys His Lys Trp Lys Leu Ser Gly Val
370 375 380
Glu Arg Ala Asn Ser Val Thr Trp Asn Pro His Lys Met Met Gly Val
385 390 395 400
Pro Leu Gln Cys Ser Ala Leu Leu Val Arg Glu Glu Gly Leu Met Gln
405 410 415
Ser Cys Asn Gln Met His Ala Ser Tyr Leu Phe Gln Gln Asp Lys His
420 425 430
Tyr Asp Leu Ser Tyr Asp Thr Gly Asp Lys Ala Leu Gln Cys Gly Arg
435 440 445
His Val Asp Val Phe Lys Leu Trp Leu Met Trp Arg Ala Lys Gly Thr
450 455 460
Thr Gly Phe Glu Ala His Ile Asp Lys Cys Leu Glu Leu Ala Glu Tyr
465 470 475 480
Leu Tyr Ser Ile Ile Lys Asn Arg Glu Gly Tyr Glu Met Val Phe Asp
485 490 495
Gly Lys Pro Gln His Thr Asn Val Cys Phe Trp Tyr Val Pro Pro Ser
500 505 510
Leu Arg Val Leu Glu Asp Asn Glu Glu Arg Met Asn Arg Leu Ser Lys
515 520 525
Val Ala Pro Val Ile Lys Ala Arg Met Met Glu Tyr Gly Thr Thr Met
530 535 540
Val Ser Tyr Gln Pro Leu Gly Asp Lys Val Asn Phe Phe Arg Met Val
545 550 555 560
Ile Ser Asn Pro Ala Ala Thr His Gln Asp Ile Asp Phe Leu Ile Glu
565 570 575
Glu Ile Glu Arg Leu Gly Gln Asp Leu
580 585
<210> 33
<211> 1758
<212> ДНК
<213> Canis lupus
<400> 33
atggcatctc caggctctgg cttctggtcc ttcgggtctg aagatggctc cggggatccc 60
gagaacccca gcacagcgag agcctggtgt caggtggccc agaagttcac gggcggcatc 120
ggaaacaagc tgtgcgccct gctctacgga gatgccgaga agcccgcgga gagtggcggg 180
agcgagcccc cgcgcgccac ctccaggaag gccgcctgcg cttgtaatca gaagccttgc 240
agctgcccca aagcggaggt caactatgcg tttctacacg caacagacct gctgccagcc 300
tgtgatggag aaaggcccac gttggcgttt ctgcaagatg ttatggacat tttgcttcag 360
tatgttgtga aaagtttcga tagatcaacc aaagtgattg atttccatta ccctaatgag 420
ctccttcaag agtataactg ggaattggca gaccaaccac aaaatttgga ggaaattttg 480
atgcattgcc aaacgactct aaaatatgca attaaaacag ggcatcccag atatttcaat 540
cagctttcca ctggactgga tatggttgga ttagcagcag actggctgac atcaacagca 600
aacacaaaca tgttcaccta tgaaattgct ccagtatttg tgctcttgga atatgtcaca 660
ctaaagaaaa tgagagaaat cattggctgg ccgggaggct ctggcgatgg gatattttct 720
cctggtggcg ctatttctaa catgtatgcc atgctgatcg cacgctttaa gatgttccca 780
gaagtcaagg agaaaggaat ggctgcggtt cccaggctca ttgccttcac atctgagcat 840
agtcactttt ctctcaagaa gggagctgca gctttgggga ttggaacaga cagcgtgatt 900
ctgattaaat gtgatgagag ggggaaaatg gtcccatctg atcttgaaag aaggatcctt 960
gaagccaaac aaaaaggatt tgttcctttc cttgtgagcg ccacagctgg gaccaccgtg 1020
tatggagcat tcgaccccct cttagcagtt gctgacattt gtaaaaagta caagatctgg 1080
atgcatgtgg atgctgcttg gggtggggga ttactgatgt cccggaagca caaatggaag 1140
ctgagcggcg tggagagggc caactctgtg acatggaacc cacacaagat gatgggcgtc 1200
cctttacagt gctccgctct cctggttaga gaagagggat tgatgcagag ttgcaaccag 1260
atgcatgcct cctacctctt ccagcaagat aaacactatg acctgtccta tgatactggg 1320
gataaggcct tacagtgtgg acgccacgtt gatgttttta aattatggct aatgtggagg 1380
gcaaagggca ccactgggtt tgaagcacat attgataagt gcctggagct ggctgagtat 1440
ttatacagta tcataaaaaa ccgagaagga tacgaaatgg tgtttgatgg aaagcctcag 1500
cacacaaatg tctgcttctg gtacgtgcct ccaagtttgc gtgtcctgga agacaatgaa 1560
gagagaatga accgcctctc aaaggtggcc ccagtgatta aagcccgaat gatggagtat 1620
gggaccacaa tggtcagcta tcagcccttg ggagacaagg tcaatttctt ccgcatggtt 1680
atctcaaatc ccgcagcaac tcaccaagac atcgacttcc tgattgaaga aatagaacgc 1740
cttggacaag atttataa 1758
<210> 34
<211> 585
<212> Белок
<213> Felis catus
<400> 34
Met Ala Thr Pro Gly Ser Gly Phe Trp Ser Phe Gly Ser Glu Asp Gly
1 5 10 15
Ser Gly Asp Pro Glu Asn Pro Gly Thr Ala Arg Ala Trp Cys Gln Val
20 25 30
Ala Gln Lys Phe Thr Gly Gly Ile Gly Asn Lys Leu Cys Ala Leu Leu
35 40 45
Tyr Gly Asp Ser Glu Lys Pro Ala Glu Ser Gly Gly Ser Gln Pro Ala
50 55 60
Arg Ala Thr Ser Arg Lys Ala Thr Cys Ala Cys Asn Gln Lys Pro Cys
65 70 75 80
Ser Cys Pro Lys Ala Asp Val Asn Tyr Ala Phe Leu His Ala Thr Asp
85 90 95
Leu Leu Pro Ala Cys Asp Gly Glu Arg Pro Thr Leu Ala Phe Leu Gln
100 105 110
Asp Val Met Gly Ile Leu Leu Gln Tyr Val Val Lys Ser Phe Asp Arg
115 120 125
Ser Thr Lys Val Ile Asp Phe His Tyr Pro Asn Glu Leu Leu Gln Glu
130 135 140
Tyr Asn Trp Glu Leu Ala Asp Gln Pro Gln Asn Leu Glu Glu Ile Leu
145 150 155 160
Met His Cys Gln Thr Thr Leu Lys Tyr Ala Ile Lys Thr Gly His Pro
165 170 175
Arg Tyr Phe Asn Gln Leu Ser Thr Gly Leu Asp Met Val Gly Leu Ala
180 185 190
Ala Asp Trp Leu Thr Ser Thr Ala Asn Thr Asn Met Phe Thr Tyr Glu
195 200 205
Ile Ala Pro Val Phe Val Leu Leu Glu Tyr Val Thr Leu Lys Lys Met
210 215 220
Arg Glu Ile Ile Gly Trp Pro Gly Gly Ser Gly Asp Gly Ile Phe Ser
225 230 235 240
Pro Gly Gly Ala Ile Ser Asn Met Tyr Ala Met Leu Ile Ala Arg Phe
245 250 255
Lys Met Phe Pro Glu Val Lys Glu Lys Gly Met Ala Ala Val Pro Arg
260 265 270
Leu Ile Ala Phe Thr Ser Glu His Ser His Phe Ser Leu Lys Lys Gly
275 280 285
Ala Ala Ala Leu Gly Ile Gly Thr Asp Ser Val Ile Leu Ile Lys Cys
290 295 300
Asp Glu Arg Gly Lys Met Ile Pro Ser Asp Leu Glu Arg Arg Ile Leu
305 310 315 320
Glu Ala Lys Gln Lys Gly Phe Val Pro Phe Leu Val Ser Ala Thr Ala
325 330 335
Gly Thr Thr Val Tyr Gly Ala Phe Asp Pro Leu Leu Ala Val Ala Asp
340 345 350
Ile Cys Lys Lys Tyr Lys Ile Trp Met His Val Asp Ala Ala Trp Gly
355 360 365
Gly Gly Leu Leu Met Ser Arg Lys His Lys Trp Lys Leu Ser Gly Val
370 375 380
Glu Arg Ala Asn Ser Val Thr Trp Asn Pro His Lys Met Met Gly Val
385 390 395 400
Pro Leu Gln Cys Ser Ala Leu Leu Val Arg Glu Glu Gly Leu Met Gln
405 410 415
Ser Cys Asn Gln Met His Ala Ser Tyr Leu Phe Gln Gln Asp Lys His
420 425 430
Tyr Asp Leu Ser Tyr Asp Thr Gly Asp Lys Ala Leu Gln Cys Gly Arg
435 440 445
His Val Asp Val Phe Lys Leu Trp Leu Met Trp Arg Ala Lys Gly Thr
450 455 460
Thr Gly Phe Glu Ala His Ile Asp Lys Cys Leu Glu Leu Ala Glu Tyr
465 470 475 480
Leu Tyr Asn Ile Ile Lys Asn Arg Glu Gly Tyr Glu Met Val Phe Asp
485 490 495
Gly Lys Pro Gln His Thr Asn Val Cys Phe Trp Tyr Val Pro Pro Ser
500 505 510
Leu Arg Val Leu Glu Asp Asn Glu Glu Arg Met Ser Arg Leu Ser Lys
515 520 525
Val Ala Pro Val Ile Lys Ala Arg Met Met Glu Tyr Gly Thr Thr Met
530 535 540
Val Ser Tyr Gln Pro Leu Gly Asp Lys Val Asn Phe Phe Arg Met Val
545 550 555 560
Ile Ser Asn Pro Ala Ala Thr His Gln Asp Ile Asp Phe Leu Ile Glu
565 570 575
Glu Ile Glu Arg Leu Gly Gln Asp Leu
580 585
<210> 35
<211> 1758
<212> ДНК
<213> Felis catus
<400> 35
atggcaactc caggctcagg cttttggtcc ttcgggtctg aagatggctc cggggatccc 60
gagaaccccg gcacagcgag agcctggtgt caggtggccc agaagttcac gggcggcatc 120
ggaaacaagc tgtgcgccct gctctacggg gattcagaga agccggcaga gagtggaggg 180
agccagcccg cgcgggccac ctcccggaag gccacctgtg cctgtaacca gaagccttgc 240
agctgcccca aagcggatgt caactatgcg tttctacacg caacagacct gctgccagcc 300
tgtgatggag aaaggcccac tttggcgttt ctgcaagatg taatgggcat tttgcttcag 360
tatgtggtga aaagtttcga cagatcaacc aaagtgattg atttccatta ccctaatgag 420
ctcctgcaag agtataactg ggaattggca gaccaaccac aaaatttgga ggaaattttg 480
atgcattgcc aaacgactct aaaatatgca ataaaaacag ggcatcccag gtacttcaat 540
caactttcca cgggactgga tatggttgga ttagcagcag actggctgac atcaacagca 600
aacactaata tgttcaccta tgaaattgct ccagtatttg tgctcttgga atatgtcaca 660
ctgaaaaaaa tgagagaaat cattggctgg cctgggggct ccggcgatgg gatattttct 720
cctggtggcg ctatatctaa catgtatgcc atgctgattg cacgctttaa gatgttccca 780
gaagtcaagg agaaaggaat ggctgctgtt cccaggctca ttgccttcac atccgagcat 840
agtcattttt ctctcaagaa gggagctgca gctctgggga ttggaacaga cagcgtgatt 900
ctgattaaat gcgatgagag agggaaaatg atcccatctg atcttgaaag aaggatcctt 960
gaagccaaac agaaaggatt tgttcctttc cttgtgagtg ccacagctgg gaccactgtg 1020
tatggagcat ttgaccccct cttggcggtc gctgacattt gcaaaaagta caagatctgg 1080
atgcatgtgg atgcagcttg gggtggggga ttactgatgt cccggaaaca caagtggaaa 1140
ctgagcggcg tggagagggc caactctgtg acatggaacc cacacaagat gatgggcgtc 1200
cccttacagt gctctgctct cctggttaga gaagaggggt tgatgcagag ttgcaaccag 1260
atgcatgctt cctacctttt ccagcaagat aaacactacg acctgtccta cgacactgga 1320
gacaaggcct tacagtgtgg acgccatgtc gatgttttta aattatggct aatgtggagg 1380
gcaaagggca ccactgggtt tgaagcacat attgataagt gcttggagct ggcagaatat 1440
ttatacaata tcataaaaaa ccgagaagga tatgaaatgg tgtttgatgg aaagcctcag 1500
cacacaaatg tctgcttctg gtacgtgcct ccaagtttgc gagtcctgga agacaatgaa 1560
gagagaatga gccgcctctc aaaggtggcc ccagtgatta aagccagaat gatggagtat 1620
gggaccacaa tggtcagcta tcagcccttg ggagacaagg tcaatttctt ccgcatggtc 1680
atctcaaatc ccgcagcaac tcaccaagac attgacttcc tgattgaaga aatagaacgc 1740
cttggacaag atttataa 1758
<210> 36
<211> 585
<212> Белок
<213> Equus caballus
<400> 36
Met Ala Ser Pro Gly Ser Gly Phe Trp Ser Phe Gly Ser Glu Asp Gly
1 5 10 15
Ser Gly Asp Pro Glu Asn Pro Gly Thr Ala Arg Ala Trp Cys Gln Val
20 25 30
Ala Gln Lys Phe Thr Gly Gly Ile Gly Asn Lys Leu Cys Ala Leu Leu
35 40 45
Tyr Gly Asp Ala Glu Lys Ala Ala Glu Ser Gly Gly Ser Glu Pro Pro
50 55 60
Arg Ala Thr Ser Arg Lys Ala Ala Cys Ser Cys Asn Gln Lys Pro Cys
65 70 75 80
Ser Cys Ser Lys Ala Asp Val Asn Tyr Ala Phe Leu His Ala Thr Asp
85 90 95
Leu Leu Pro Ala Cys Asp Gly Glu Arg Pro Thr Leu Ala Phe Leu Gln
100 105 110
Asp Val Met Asp Ile Leu Leu Gln Tyr Val Val Lys Ser Phe Asp Arg
115 120 125
Ser Thr Lys Val Ile Asp Phe His Tyr Pro Asn Glu Leu Leu Gln Glu
130 135 140
Tyr Asn Trp Glu Leu Ala Asp Gln Pro Gln Asn Leu Glu Glu Ile Leu
145 150 155 160
Met His Cys Gln Thr Thr Leu Lys Tyr Ala Ile Lys Thr Gly His Pro
165 170 175
Arg Tyr Phe Asn Gln Leu Ser Thr Gly Leu Asp Met Val Gly Leu Ala
180 185 190
Ala Asp Trp Leu Thr Ser Thr Ala Asn Thr Asn Met Phe Thr Tyr Glu
195 200 205
Ile Ala Pro Val Phe Val Leu Leu Glu Tyr Val Thr Leu Lys Lys Met
210 215 220
Arg Glu Ile Ile Gly Trp Pro Gly Gly Ser Gly Asp Gly Ile Phe Ser
225 230 235 240
Pro Gly Gly Ala Ile Ser Asn Met Tyr Ala Met Leu Ile Ala Arg Phe
245 250 255
Lys Met Phe Pro Glu Val Lys Glu Lys Gly Met Ala Ala Val Pro Arg
260 265 270
Leu Ile Ala Phe Thr Ser Glu His Ser His Phe Ser Leu Lys Lys Gly
275 280 285
Ala Ala Ala Leu Gly Ile Gly Thr Asp Ser Val Ile Leu Ile Arg Cys
290 295 300
Asp Glu Arg Gly Lys Met Ile Pro Ser Asp Leu Glu Arg Arg Ile Leu
305 310 315 320
Glu Ala Lys Gln Lys Gly Phe Val Pro Phe Leu Val Ser Ala Thr Ala
325 330 335
Gly Thr Thr Val Tyr Gly Ala Phe Asp Pro Leu Leu Ala Val Ala Asp
340 345 350
Ile Cys Lys Lys Tyr Lys Ile Trp Met His Val Asp Ala Ala Trp Gly
355 360 365
Gly Gly Leu Leu Met Ser Arg Lys His Lys Trp Lys Leu Ser Gly Val
370 375 380
Glu Arg Ala Asn Ser Val Thr Trp Asn Pro His Lys Met Met Gly Val
385 390 395 400
Pro Leu Gln Cys Ser Ala Leu Leu Val Arg Glu Glu Gly Leu Met Gln
405 410 415
Ser Cys Asn Gln Met His Ala Ser Tyr Leu Phe Gln Gln Asp Lys His
420 425 430
Tyr Asp Leu Ser Tyr Asp Thr Gly Asp Lys Ala Leu Gln Cys Gly Arg
435 440 445
His Val Asp Val Phe Lys Leu Trp Leu Met Trp Arg Ala Lys Gly Thr
450 455 460
Thr Gly Phe Glu Ala His Ile Asp Lys Cys Leu Glu Leu Ala Glu Tyr
465 470 475 480
Leu Tyr Asn Ile Ile Lys Asn Arg Glu Gly Tyr Glu Met Val Phe Asp
485 490 495
Gly Lys Pro Gln His Thr Asn Val Cys Phe Trp Tyr Val Pro Pro Ser
500 505 510
Leu Arg Val Leu Glu Asp Asn Glu Glu Arg Met Ser Arg Leu Ser Lys
515 520 525
Val Ala Pro Val Ile Lys Ala Arg Met Met Glu Tyr Gly Thr Thr Met
530 535 540
Val Ser Tyr Gln Pro Leu Gly Asp Lys Val Asn Phe Phe Arg Met Val
545 550 555 560
Ile Ser Asn Pro Ala Ala Thr His Gln Asp Ile Asp Phe Leu Ile Glu
565 570 575
Glu Ile Glu Arg Leu Gly Gln Asp Leu
580 585
<210> 37
<211> 1758
<212> ДНК
<213> Equus caballus
<400> 37
atggcatctc ccggctccgg cttttggtcc tttgggtctg aagatggctc cggggatccc 60
gagaaccctg gcacagcgag agcctggtgt caggtggccc agaagttcac cggcggcatc 120
ggaaacaagc tatgcgccct gctctacgga gacgccgaga aggcggcgga gagcggcggg 180
agcgagcccc cgcgggccac ctcccggaag gccgcctgct cctgcaacca gaagccctgc 240
agctgctcca aagccgatgt caactatgcg tttctacacg caacagactt gctgccagct 300
tgtgacggag aaagacccac tttggcgttt ctgcaagatg ttatggacat tttgcttcag 360
tatgtggtga aaagtttcga tagatcaacc aaagtgattg acttccatta ccctaatgag 420
ctccttcaag agtataattg ggaattggca gaccaaccac aaaatctgga ggaaattttg 480
atgcattgcc aaacaacttt aaaatatgca attaaaacag ggcatcctag atatttcaat 540
caactttcca ctggactgga tatggttgga ttagcagcag actggctgac atcaacagca 600
aacaccaaca tgttcaccta tgaaattgct ccagtattcg tgcttttgga atatgtcaca 660
ttaaagaaaa tgagagaaat cattggctgg ccaggaggct ctggcgatgg aatattttct 720
cctggtggcg ccatatctaa catgtatgcc atgctgattg cacgctttaa gatgttccca 780
gaagtcaagg agaaaggaat ggccgctgtt cccaggctca ttgccttcac gtctgagcat 840
agtcattttt ctctcaagaa gggagctgca gccttgggga ttggaacaga cagcgtaatt 900
ctgattagat gtgatgagag ggggaaaatg atcccatcgg atcttgaaag aagaatcctt 960
gaagccaaac aaaaaggatt tgtccctttt cttgtgagtg ccacggctgg gaccaccgtg 1020
tatggagcat tcgatcccct cttagctgtc gctgacattt gcaaaaagta caagatctgg 1080
atgcatgtgg atgcagcttg gggcggggga ttactgatgt cccggaaaca caagtggaaa 1140
ctgagtggcg tggagagggc caactctgtg acatggaatc cacacaagat gatgggtgtc 1200
cctttgcagt gctctgctct cctggttaga gaagagggat tgatgcagag ttgcaaccag 1260
atgcatgcct cctacctctt tcagcaagat aaacactatg acctgtccta tgacactgga 1320
gacaaggcct tgcagtgcgg acgccacgtg gatgttttta agttatggct catgtggagg 1380
gcaaagggaa caactgggtt tgaagcacat attgataagt gtttggagtt ggcggagtat 1440
ttatacaata tcataaaaaa ccgagaagga tatgaaatgg tgtttgacgg aaagcctcag 1500
cacaccaatg tctgcttctg gtatgtacct ccgagtctgc gtgttctaga agacaatgaa 1560
gagagaatga gccgcctctc aaaggtggcc ccggtgatta aagccagaat gatggagtat 1620
gggaccacaa tggtcagcta ccagcccttg ggagacaagg tcaatttctt ccgcatggtc 1680
atctcaaatc ccgcagcaac tcaccaagac attgacttcc tgattgaaga aatagaacgc 1740
cttggacaag atttataa 1758
<210> 38
<211> 181
<212> Белок
<213> Canis lupus
<400> 38
Met His Gly Ser Ala Leu Leu Cys Cys Cys Leu Val Leu Leu Ala Gly
1 5 10 15
Val Gly Ala Ser Arg His Gln Ser Thr Leu Leu Glu Asp Asp Cys Thr
20 25 30
His Phe Pro Ala Ser Leu Pro His Met Leu Arg Glu Leu Arg Ala Ala
35 40 45
Phe Gly Arg Val Lys Ile Phe Phe Gln Met Lys Asp Lys Leu Asp Asn
50 55 60
Ile Leu Leu Thr Gly Ser Leu Leu Glu Asp Phe Lys Ser Tyr Leu Gly
65 70 75 80
Cys Gln Ala Leu Ser Glu Met Ile Gln Phe Tyr Leu Glu Glu Val Met
85 90 95
Pro Arg Ala Glu Asn His Asp Pro Asp Ile Lys Asn His Val Asn Ser
100 105 110
Leu Gly Glu Lys Leu Lys Thr Leu Arg Leu Arg Leu Arg Leu Arg Arg
115 120 125
Cys His Arg Phe Leu Pro Cys Glu Asn Lys Ser Lys Ala Val Glu Gln
130 135 140
Val Lys Ser Ala Phe Ser Lys Leu Gln Glu Lys Gly Val Tyr Lys Ala
145 150 155 160
Met Ser Glu Phe Asp Ile Phe Ile Asn Tyr Ile Glu Thr Tyr Met Thr
165 170 175
Met Arg Met Lys Ile
180
<210> 39
<211> 546
<212> ДНК
<213> Canis lupus
<400> 39
atgcatggct cagcactgct ctgttgctgc ctggtcctcc tggccggggt gggagccagc 60
cgacaccaga gcaccctact tgaggacgac tgcacccact tcccagccag cctgccccac 120
atgctccgag agctccgagc tgccttcggg agggtgaaga tcttctttca aatgaaggac 180
aagctggaca acatactgct gaccgggtcc ctgctggagg actttaagag ttacctgggt 240
tgccaagccc tgtcggagat gatccagttt tacttggagg aggtgatgcc ccgggctgag 300
aaccacgacc cagacatcaa gaaccacgtg aactccctgg gagagaagct caagaccctc 360
aggctgagac tgaggctgcg acgctgtcac cgatttcttc cctgtgagaa taagagcaag 420
gcggtggagc aggtgaagag cgcatttagt aagctccagg agaaaggtgt ctacaaagcc 480
atgagtgagt ttgacatctt catcaactac atagaaacct acatgacaat gaggatgaaa 540
atctga 546
<210> 40
<211> 178
<212> Белок
<213> Felis catus
<400> 40
Met His Ser Ser Ala Leu Leu Cys Phe Leu Val Phe Leu Ala Gly Val
1 5 10 15
Gly Ala Ser Arg His Gln Ser Thr Leu Ser Glu Asp Asn Cys Thr His
20 25 30
Phe Ser Val Ser Leu Pro His Met Leu Arg Glu Leu Arg Ala Ala Phe
35 40 45
Gly Lys Val Lys Thr Phe Phe Gln Thr Lys Asp Glu Leu His Ser Ile
50 55 60
Leu Leu Thr Arg Ser Leu Leu Glu Asp Phe Lys Gly Tyr Leu Gly Cys
65 70 75 80
Gln Ala Leu Ser Glu Met Ile Gln Phe Tyr Leu Glu Glu Val Met Pro
85 90 95
Gln Ala Glu Asn Glu Asp Pro Asp Ile Lys Gln His Val Asn Ser Leu
100 105 110
Gly Glu Lys Leu Lys Thr Leu Arg Leu Arg Leu Arg Arg Cys His Arg
115 120 125
Phe Leu Pro Cys Glu Asn Lys Ser Lys Val Val Glu Gln Val Lys Ser
130 135 140
Thr Phe Ser Lys Leu Gln Glu Lys Gly Val Tyr Lys Ala Met Gly Glu
145 150 155 160
Phe Asp Ile Phe Ile Asn Tyr Ile Glu Ala Tyr Met Thr Met Lys Met
165 170 175
Lys Ile
<210> 41
<211> 537
<212> ДНК
<213> Felis catus
<400> 41
atgcacagct cagcacttct gtgtttcctg gtcttcctgg ccggggtagg agccagccga 60
caccagagca ccctgtctga ggacaactgc acccacttct cagtcagcct gccccacatg 120
ctccgagagc tccgagctgc cttcggcaag gtgaagactt tctttcaaac caaggacgag 180
ctgcacagca tattgttgac caggtccttg ctggaggact ttaagggtta cctgggttgc 240
caagccttgt ccgagatgat ccagttttat ttggaggagg tgatgcccca ggctgagaac 300
gaggacccag acatcaaaca gcacgtgaac tccctgggag aaaagctgaa gaccctccgg 360
ctgagactgc ggcgctgtca tcgatttctg ccctgtgaaa acaagagcaa ggtggtggag 420
caggtgaaga gtacctttag taagctccaa gagaaaggtg tctacaaagc catgggtgag 480
tttgacatct tcatcaacta catagaagct tacatgacaa tgaagatgaa aatctga 537
<210> 42
<211> 178
<212> Белок
<213> Equus caballus
<400> 42
Met His Ser Ser Ala Leu Leu Cys Tyr Leu Val Phe Leu Ala Gly Val
1 5 10 15
Gly Ala Ser Arg Asp Arg Gly Thr Gln Ser Glu Asn Ser Cys Thr His
20 25 30
Phe Pro Thr Ser Leu Pro His Met Leu His Glu Leu Arg Ala Ala Phe
35 40 45
Ser Arg Val Lys Thr Phe Phe Gln Met Lys Asp Gln Leu Asp Asn Met
50 55 60
Leu Leu Asn Gly Ser Leu Leu Glu Asp Phe Lys Gly Tyr Leu Gly Cys
65 70 75 80
Gln Ala Leu Ser Glu Met Ile Gln Phe Tyr Leu Glu Glu Val Met Pro
85 90 95
Gln Ala Glu Asn His Gly Pro Asp Ile Lys Glu His Val Asn Ser Leu
100 105 110
Gly Glu Lys Leu Lys Thr Leu Arg Val Arg Leu Arg Arg Cys His Arg
115 120 125
Phe Leu Pro Cys Glu Asn Lys Ser Lys Ala Val Glu Gln Val Lys Ser
130 135 140
Ala Phe Ser Lys Leu Gln Glu Lys Gly Val Tyr Lys Ala Met Ser Glu
145 150 155 160
Phe Asp Ile Phe Ile Asn Tyr Ile Glu Ala Tyr Met Thr Thr Lys Met
165 170 175
Lys Asn
<210> 43
<211> 537
<212> ДНК
<213> Equus caballus
<400> 43
atgcacagct cagcactgct atgttacctg gtcttcctgg ccggggtggg agccagccga 60
gaccggggca cccagtctga gaacagctgc acccacttcc caaccagcct gccccacatg 120
ctccatgagc tccgagccgc cttcagcagg gtgaagactt tctttcaaat gaaggaccag 180
ctggacaaca tgttgttgaa cgggtccctg ctggaggact ttaagggtta cctgggttgc 240
caagccttgt cggagatgat ccagttttac ctggaggagg tgatgcccca ggctgagaac 300
cacggcccag acatcaagga gcacgtgaac tccctggggg aaaagctgaa gaccctccga 360
gtgaggctgc ggcgctgtca tcgatttctg ccctgtgaaa ataagagcaa ggcagtggag 420
caggtgaaga gtgccttcag taagctccaa gagaaaggtg tctacaaagc catgagtgag 480
tttgacatct tcatcaacta catagaagcc tatatgacaa cgaagatgaa aaactga 537
<210> 44
<211> 185
<212> Белок
<213> Canis lupus
<400> 44
Met Lys Leu Trp Asp Val Val Ala Val Cys Leu Val Leu Leu His Thr
1 5 10 15
Ala Ser Ala Leu Pro Leu Pro Ala Ala Asn Val Pro Glu Asp Tyr Ser
20 25 30
Asp Gln Phe Asp Asp Val Met Asp Phe Ile Gln Ala Thr Ile Arg Arg
35 40 45
Leu Lys Arg Ser Pro Glu Lys Gln Met Ala Val Pro Ala Arg Arg Glu
50 55 60
Arg Asn Arg Gln Ala Ala Ala Ala Gly Pro Glu His Ser Arg Gly Lys
65 70 75 80
Gly Arg Arg Gly Pro Arg Gly Arg Asn Arg Gly Cys Val Leu Thr Ala
85 90 95
Ile His Leu Asn Val Thr Asp Leu Gly Leu Gly Tyr Glu Thr Lys Glu
100 105 110
Glu Leu Ile Phe Arg Tyr Cys Ser Gly Ser Cys Asp Ala Ala Glu Thr
115 120 125
Met Tyr Asp Lys Ile Leu Lys Asn Leu Ser Lys Ser Arg Arg Leu Ala
130 135 140
Ser Asp Lys Ala Gly Gln Ala Cys Cys Arg Pro Ile Ala Tyr Asp Asp
145 150 155 160
Asp Leu Ser Phe Leu Asp Asp Asn Leu Val Tyr His Ile Leu Arg Lys
165 170 175
His Ser Ala Lys Arg Cys Gly Cys Ile
180 185
<210> 45
<211> 558
<212> ДНК
<213> Canis lupus
<400> 45
atgaagttat gggatgtcgt ggctgtctgc ctggtgctgc tccacaccgc gtccgccctc 60
ccgctgcccg ccgcaaacgt gccggaggac tattctgatc agtttgatga cgtcatggat 120
tttattcagg ccaccatcag aaggctgaaa aggtcacccg agaaacaaat ggccgtgcca 180
gcgagacgag agcggaatcg tcaggccgcg gccgccggcc cggaacattc cagggggaag 240
gggcggcgag gcccgagggg cagaaaccgg ggttgtgtct tgactgcgat acatttaaac 300
gtcactgacc tgggcttggg ctacgaaacc aaggaggaac tgatttttag gtactgcagc 360
ggctcctgcg acgcggccga gaccatgtac gacaaaatat taaaaaactt atccaaaagt 420
agaaggctgg cgagtgacaa agcagggcag gcttgctgca gacccatcgc ctacgatgac 480
gacctgtcgt ttttagatga caacctggtt taccatattc taagaaagca ttccgctaaa 540
aggtgtggat gtatctga 558
<210> 46
<211> 211
<212> Белок
<213> Felis catus
<400> 46
Met Lys Leu Trp Asp Val Val Ala Val Cys Leu Val Leu Leu His Thr
1 5 10 15
Ala Ser Ala Phe Pro Leu Pro Ala Gly Lys Arg Pro Pro Glu Ala Pro
20 25 30
Ala Glu Asp Arg Ser Leu Gly Arg Arg Arg Ala Pro Phe Ala Leu Ser
35 40 45
Ser Asp Ser Asn Met Pro Glu Asp Tyr Pro Asp Gln Phe Asp Asp Val
50 55 60
Met Asp Phe Ile Gln Ala Thr Ile Arg Arg Leu Lys Arg Ser Pro Glu
65 70 75 80
Lys Gln Met Ala Leu Pro Pro Arg Arg Glu Arg Asn Arg Gln Ala Ala
85 90 95
Ala Ala Asn Pro Glu Asn Ser Arg Gly Lys Gly Arg Arg Gly Gln Arg
100 105 110
Gly Arg Asn Arg Gly Cys Val Leu Thr Ala Ile His Leu Asn Val Thr
115 120 125
Asp Leu Gly Leu Gly Tyr Glu Thr Lys Glu Glu Leu Ile Phe Arg Tyr
130 135 140
Cys Ser Gly Ser Cys Asp Ala Ala Glu Thr Met Tyr Asp Lys Ile Leu
145 150 155 160
Lys Asn Leu Ser Lys Asn Arg Arg Leu Val Ser Asp Lys Val Gly Gln
165 170 175
Ala Cys Cys Arg Pro Ile Ala Tyr Asp Asp Asp Leu Ser Phe Leu Asp
180 185 190
Asp Asn Leu Val Tyr His Ile Leu Arg Lys His Ser Ala Lys Arg Cys
195 200 205
Gly Cys Ile
210
<210> 47
<211> 636
<212> ДНК
<213> Felis catus
<400> 47
atgaagttat gggatgtcgt ggctgtctgc ctggtgctgc tccacaccgc gtccgccttc 60
ccgctgcccg ccggtaagag gcctcccgag gcgcccgccg aagaccgctc cctcggccgc 120
cgccgcgcgc ccttcgcgct gagcagtgac tcaaatatgc cagaggatta tcctgatcag 180
tttgacgacg tcatggattt tattcaagct accatcagaa gactgaaaag gtcacccgag 240
aaacaaatgg ccttgccgcc tagaagagag cggaatcggc aggcggcggc cgccaacccg 300
gagaattcca gagggaaagg tcggcgaggc cagaggggca gaaatcgggg ttgtgtctta 360
actgcgatac atttgaacgt caccgacctg ggtttgggct acgaaaccaa ggaggaactg 420
atttttaggt actgcagcgg ctcctgtgat gcagctgaga caatgtacga caaaatatta 480
aaaaacttat ccaaaaacag aaggctggtg agtgacaaag tcgggcaggc atgttgcaga 540
cccatcgcct atgacgacga cctgtcgttt ttagatgaca acctggttta ccatattcta 600
agaaagcatt ccgctaaaag gtgtggatgt atctga 636
<210> 48
<211> 185
<212> Белок
<213> Equus caballus
<400> 48
Met Lys Leu Trp Asp Val Val Ala Val Cys Leu Val Leu Leu His Thr
1 5 10 15
Ala Ser Ala Phe Pro Leu Pro Ala Ala Asn Met Pro Glu Asp Tyr Pro
20 25 30
Asp Gln Phe Asp Asp Val Met Asp Phe Ile Gln Ala Thr Ile Lys Arg
35 40 45
Leu Lys Arg Ser Pro Asp Lys Gln Met Ala Val Leu Pro Arg Arg Glu
50 55 60
Arg Asn Arg Gln Ala Ala Ala Ala Asn Pro Glu Asn Ser Arg Arg Lys
65 70 75 80
Gly Gln Arg Gly Gln Arg Gly Lys Asn Arg Gly Cys Val Leu Thr Ala
85 90 95
Ile His Leu Asn Val Thr Asp Leu Gly Leu Gly Tyr Glu Thr Lys Glu
100 105 110
Glu Leu Ile Phe Arg Tyr Cys Ser Gly Ser Cys Glu Ala Ala Glu Thr
115 120 125
Met Tyr Asp Lys Ile Leu Lys Asn Leu Ser Lys Asn Arg Arg Leu Val
130 135 140
Ser Asp Lys Val Gly Gln Ala Cys Cys Arg Pro Ile Ala Phe Asp Asp
145 150 155 160
Asp Leu Ser Phe Leu Asp Asp Asn Leu Val Tyr His Ile Leu Arg Lys
165 170 175
His Ser Ala Lys Arg Cys Gly Cys Ile
180 185
<210> 49
<211> 558
<212> ДНК
<213> Equus caballus
<400> 49
atgaagttat gggatgtcgt ggctgtctgc ctggtgctgc tccacaccgc gtccgccttc 60
ccgctgcccg ccgcaaatat gccagaggat tatcctgatc agtttgatga tgtcatggat 120
tttattcaag ccaccattaa aagactgaaa aggtcaccag ataaacaaat ggcagtgctt 180
cctagaagag agcggaatcg gcaggctgca gctgccaacc cggagaattc cagaaggaaa 240
ggtcagcgag gccagagggg caaaaaccgg ggttgtgtct taaccgcgat acatttaaat 300
gtcactgact tgggtttggg ctacgaaacc aaggaggaac tgatttttag gtactgcagt 360
ggctcctgcg aggcagccga gacaatgtac gacaaaatat taaaaaactt atccaaaaat 420
agaaggctgg tgagtgacaa agtagggcag gcatgttgca gacccatcgc cttcgatgac 480
gacctgtcat ttttagatga taacttggtt taccatattc taagaaagca ttccgctaaa 540
aggtgtggat gtatctga 558
<---