ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ ОБНАРУЖЕНИЯ И РАСПОЗНАВАНИЯ УСТОЙЧИВОГО К МЕТИЦИЛЛИНУ ЗОЛОТИСТОГО СТАФИЛОКОККА (STAPHYLOCOCCUS AUREUS) (MRSA) С MREJ ТИПА XXI
Вид РИД
Изобретение
[0001] Настоящая заявка подана вместе с Перечнем последовательностей в электронном виде. Перечень последовательностей предоставлен в виде файла, названного GENOM.114A.txt, который последний раз сохранили 14 марта 2013 г., который имеет размер 85,6 килобайт. Указанные в нем сведения полностью включены в данную заявку посредством ссылки.
УРОВЕНЬ ТЕХНИКИ
Область техники
[0002] Варианты реализации, описанные в данной заявке, относятся к средствам молекулярной диагностики для обнаружения устойчивого к метициллину Staphylococcus aureus.
Предшествующий уровень техники
[0003] Экспрессирующие коагулазу виды Staphylococcus aureus (S. aureus) являются условно-патогенными для человека микроорганизмами, что доказано документально (Murray и др. ред., 1999 г., Manual of Clinical Microbiology, 7oe изд., ASM Press, Вашингтон, округ Колумбия). Внутрибольничные инфекции, вызванные S. aureus, являются основной причиной заболеваемости и смертности. Некоторые из наиболее распространенных инфекций, вызываемых S. aureus, поражают кожу, и они включают фурункулы или нарывы, панникулит, импетиго и послеоперационные инфекции ран в различных областях тела. Среди некоторых наиболее тяжелых инфекций, вызванных S. aureus, можно назвать бактериемию, пневмонию, остеомиелит, острый эндокардит, миокардит, перикардит, энцефалит, менингит, синдром ошпаренной кожи и различные абсцессы. Пищевое отравление, опосредованное стафилококковыми энтеротоксинами, представляет собой еще один важный синдром, связанный с S. aureus. Синдром токсического шока, внебольничное заболевание, также объясняют инфекцией или колонизацией токсикогенным S. aureus.
[0004] Устойчивый к метициллину S. aureus (MRSA) стал в 1980-х основной клинической и эпидемиологической проблемой в больницах (Oliveira и др. (2002), Lancet Infect Dis. 2:180-9). MRSA устойчив ко всем β-лактамным соединениям, включая пенициллины, цефалоспорины, карбапенемы и монобактамы, которые представляют собой наиболее часто применяемые антибиотики для лечения инфекций S. aureus.
[0005] Инфекции MRSA поддаются лечению только токсичными и более дорогостоящими антибиотиками, которые обычно используют как последнюю линию защиты. Поскольку MRSA могут легко распространяться от пациента к пациенту через персонал, больницы по всему миру сталкиваются с проблемой контролирования MRSA.
[0006] Еще совсем недавно единственным способом выявления устойчивости штамма было проведение тестирования на чувствительность к противомикробным средствам вручную. Тестирование на чувствительность к противомикробным средствам имеет множество недостатков, включая длительное время (по меньшей мере 48 часов) до получения результатов, недостаточную воспроизводимость, отсутствие стандартизации данного процесса и ошибки персонала. Следовательно, существует потребность в разработке быстрых и простых скрининговых или диагностических тестов для обнаружения и/или распознавания MRSA, с целью уменьшить его распространение и улучшить диагностику и лечение инфицированных пациентов.
[0007] Существует потребность в композициях и способах для быстрого и чувствительного обнаружения MRSA.
КРАТКОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0008] Варианты реализации, описанные в данной заявке, основаны, отчасти, на обнаружении того, что некоторые штаммы Staphylococcus aureus, включая те, которые несут ген, гомологичный mecA, mecALGA251, имеют одну и ту же последовательность, расположенную на правом конце области SCCmec нуклеиновых кислот MRSA, а именно полиморфную область соединения на правом конце. В данной заявке предложены способы и композиции, которые можно применять для обнаружения данных штаммов MRSA, которые до настоящего времени невозможно было обнаружить с помощью обычных коммерчески доступных систем молекулярного анализа. Также в данной заявке предложены композиции и способы, которые обеспечивают возможность дополнительного (например, либо одновременного, либо последовательного) обнаружения Staphylococcus aureus в принципе, и/или дополнительного обнаружения генов mecA и/или mtcALGA251, дополнительно к штаммам MRSA.
[0009] Соответственно, в данной заявке предложены способы и композиции для обнаружения MRSA, который содержит последовательность MREJ типа xxi. В некоторых вариантах реализации предложена композиция для обнаружения устойчивого к метициллину Staphylococcus aureus (MRSA), имеющего нуклеиновые кислоты MREJ типа xxi. MRSA может содержать элемент стафилококковой хромосомной кассеты mec (SCCmec), включая гомолог mecA (mecALGA251, или mecC), при этом кассета SCCmec встроена в хромосомную ДНК S. aureus, что тем самым приводит к образованию полиморфной последовательности типа xxi области соединения на правом конце (MREJ), которая включает полиморфные последовательности на правом конце и хромосомную ДНК, примыкающую к полиморфным последовательностям на правом конце. Композиция может содержать первый праймер для амплификации, который имеет длину от 10 до 45 нуклеотидов и который специфично гибридизуется при стандартных условиях ПЦР с полиморфными последовательностями на правом конце нуклеиновых кислот MREJ типа xxi. В некоторых вариантах реализации, указанный первый праймер для амплификации специфично гибридизуется с последовательностью нуклеиновой кислоты SEQ ID NO: 1 или с комплементарной ей последовательностью при указанных стандартных условиях ПЦР.
[0010] Композиции, описанные в данной заявке, могут дополнительно содержать второй праймер для амплификации, который имеет длину от 10 до 45 нуклеотидов и который специфично гибридизуется при стандартных условиях ПЦР с хромосомными последовательностями S. aureus, расположенными в пределах 1 т.п.н. от точки встраивания элемента SCCmec в хромосомную ДНК. Предпочтительно, указанные первый и второй праймеры для амплификации совместно образуют ампликон области соединения на правом конце нуклеиновых кислот MREJ типа xxi при стандартных условиях ПЦР в случае присутствия MRSA, включающего нуклеиновые кислоты MREJ типа xxi. Соответственно, в некоторых вариантах реализации, второй праймер для амплификации специфично гибридизуется при стандартных условиях ПЦР с orfX. Композиции, описанные в данной заявке, могут дополнительно содержать зонд, например, олигонуклеотидный зонд, который содержит молекулу источника флуоресценции и молекулу гасителя флуоресценции, специфично гибридизующуюся с ампликоном нуклеиновых кислот MREJ типа xxi при стандартных условиях ПЦР.
[0011] Композиции, описанные в данной заявке, могут содержать один или более дополнительных праймеров для амплификации, имеющих длину от 10 до 45 нуклеотидов, которые специфично гибридизуются с одной или более полиморфными последовательностями на правом конце SCCmec из MRSA с i по xx типа MREJ, т.е. с одной или более полиморфными последовательностями на правом конце SCCmec, выбранными из группы, состоящей из последовательностей SEQ ID NO: 5-29.
[0012] В указанных способах и композициях также можно применять дополнительные олигонуклеотиды, т.е. которые сконструированы таким образом, чтобы специфично амплифицировать последовательности mecA и/или mecALGA251/mecC, и/или которые сконструированы таким образом, чтобы специфично амплифицировать характерные для Staphylococcus aureus последовательности.
[0013] Соответственно, в некоторых вариантах реализации предложены композиции, в которых указанный первый праймер для амплификации по меньшей мере на 80% идентичен последовательности SEQ ID NO: 2. В некоторых вариантах реализации, композиции могут включать второй праймер для амплификации, который по меньшей мере на 80% идентичен последовательности SEQ ID NO: 3. В некоторых вариантах реализации, композиция может содержать зонд, при этом указанный зонд по меньшей мере на 80% идентичен последовательности SEQ ID NO: 4 или 82.
[0014] В некоторых вариантах реализации, композиции, описанные в данной заявке, обеспечивают в безводной форме, например, в лиофилизированной или тому подобной форме.
[0015] Также в данной заявке предложены способы обнаружения устойчивого к метициллину Staphylococcus aureus (MRSA), включающего нуклеиновые кислоты MREJ типа xxi, и способы обнаружения нуклеиновых кислот MREJ типа xxi, при этом S. aureus содержит элемент стафилококковой хромосомной кассеты mec (SCCmec), включая гомолог mecA (mecALGA251 или mecC). Кассета SCCmec может быть встроена в хромосомную ДНК S. aureus, что тем самым приводит к образованию полиморфной последовательности типа xxi области соединения на правом конце (MREJ), которая включает полиморфные последовательности на правом конце (последовательности MREP) и хромосомную ДНК, примыкающую к полиморфным последовательностям на правом конце. Соответственно, в некоторых вариантах реализации, указанные способы могут включать этапы обеспечения тестируемого образца; осуществления контакта указанно образца с первым праймером для амплификации, имеющим длину от 10 до 45 нуклеотидов, который специфичным образом гибридизуется при стандартных условиях ПЦР с полиморфными последовательностями на правом конце последовательности MREJ типа xxi; при этом указанное осуществление контакта проводят при условиях, когда в случае, если в указанном образце присутствует S. aureus, включающий нуклеиновые кислоты MREJ типа xxi, то образуется ампликон области соединения на правом конце mec нуклеиновых кислот MREJ типа xxi. Указанный способ также может включать этап определения того, образовался или нет ампликон нуклеиновых кислот MREJ типа xxi после указанного этапа осуществления контакта. В некоторых вариантах реализации, первый праймер для амплификации специфично гибридизуется с последовательностью нуклеиновых кислот SEQ ID NO: 1 при указанных стандартных условиях ПЦР.
[0016] В некоторых вариантах реализации, указанный способ может включать осуществление контакта образца со вторым праймером для амплификации, имеющим длину от 10 до 45 нуклеотидов, который гибридизуется при стандартных условиях ПЦР с геном orfX S. aureus, при этом указанные первый и второй праймеры для амплификации совместно приводят к образованию ампликона последовательности области соединения на правом конце SCCmec (MREJ) MRSA при стандартных условиях ПЦР в случае присутствия нуклеиновых кислот MREJ типа xxi.
[0017] Способ по п. 15 дополнительно включает осуществление контакта образца с зондом, при этом указанный зонд специфично гибридизуется с ампликоном последовательности области соединения на правом конце SCCmec (MREJ) нуклеиновых кислот MREJ типа xxi при стандартных условиях ПЦР. В некоторых вариантах реализации, зонд содержит молекулу источника флуоресценции и молекулу гасителя флуоресценции.
[0018] В некоторых вариантах реализации, этап осуществления контакта также включает осуществление контакта образца с одним или более дополнительными праймерами для амплификации, при этом указанные один или более дополнительных праймеров для амплификации имеют длину от 10 до 45 нуклеотидов, которые специфично гибридизуются с одной из полиморфных последовательностей на правом конце SCCmec из MRSA с MREJ типа с i по хх. В некоторых вариантах реализации, этап осуществления контакта также включает осуществление контакта указанного образца с одним или более дополнительными праймерами для амплификации, имеющими длину от 10 до 45 нуклеотидов, которые специфично гибридизуются и сконструированы таким образом, чтобы образовывать ампликон последовательностей mecA, последовательностей mecC и/или специфичных для Staphylococcus aureus последовательностей. Некоторые частные примеры специфичных для S. aureus последовательностей включают, но не ограничены перечисленными: последовательности nuc, последовательности рибосомной РНК (рРНК), последовательности femB, последовательности Sa442 и тому подобные последовательности. Для квалифицированного специалиста должно быть очевидно, тем не менее, что любую последовательность, которая уникальна для Staphylococcus aureus, можно применять в вариантах реализации, описанных в данной заявке.
[0019] Соответственно, этап осуществления контакта согласно способам, описанным в данной заявке, может включать проведение реакции амплификации нуклеиновых кислот, такой как ПЦР, амплификации с замещением цепи (SDA) (например, амплификации с множественным замещением цепи (MDA)), изотермической амплификации с формированием петель (LAMP), лигазной цепной реакции (ЛЦР), иммунологической амплификации, амплификации, основанной на последовательности нуклеиновых кислот (NASBA), самоподдерживающейся репликации последовательностей (3SR) или амплификации по типу катящегося кольца. В некоторых предпочтительных вариантах реализации, способ включает осуществление мультиплексной ПЦР. В некоторых предпочтительных вариантах реализации, способ включает осуществление ПЦР в реальном времени.
КРАТКОЕ ОПИСАНИЕ ФИГУР
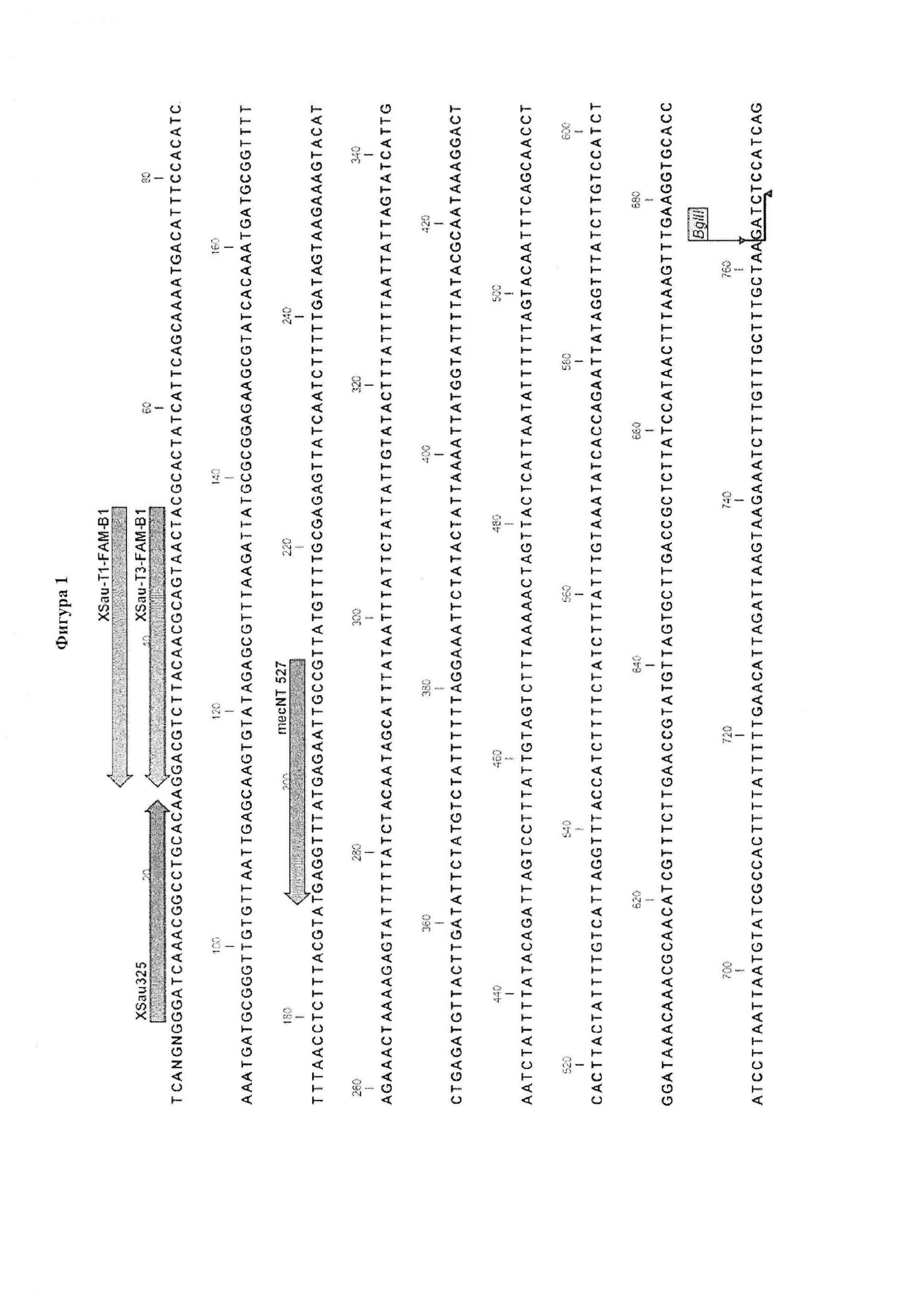
[0020] На Фигуре 1 представлена последовательность области MREJ типа xxi. Также показано расположение различных праймеров и зондов, использованных в вариантах реализации, описанных в данной заявке.
ПОДРОБНОЕ ОПИСАНИЕ ИЗОБРЕТЕНИЯ
[0021] Следует понимать, что как предшествующее краткое описание, так и последующее подробное описание приведены исключительно в качестве примера и объяснения и не предполагают ограничение объема настоящего изобретения. В данном описании, использование форм единственного числа включает формы множественного числа, если не указано иначе. Также, использование терминов "состоят из", "содержат" и "включают", или их модификаций с однокоренными словами, например, но не ограничиваясь перечисленными, "состоит из", "содержал" и "включая", не предполагается ограничивающим. Использование термина "или" означает "и/или", если не указано иначе. Термин "и/или" означает, что термины перед и после него можно брать в совокупности или отдельно. Для целей иллюстрирования, но не в качестве ограничения, "X и/или Y" может означать "X" или "Y" или "X и Y".
[0022] Всякий раз, когда в данной заявке предусмотрен диапазон значений, диапазон считают включающим начальное значение и конечное значение, и любое значение или диапазон значений между ними, если конкретно не указано иное. Например, "от 0,2 до 0,5" означает 0,2, 0,3, 0,4, 0,5; диапазоны между ними, такие как 0,2-0,3, 0,3-0,4, 0,2-0,4; значения между ними с учетом меньшего порядка, такие как 0,25, 0,35, 0,225, 0,335, 0,49; диапазоны значений между ними с учетом меньшего порядка, такие как 0,26-0,39; и тому подобное.
[0023] Заголовки разделов используются в данной заявке исключительно с целью организации и не должны истолковываться как ограничивающие каким-либо образом описанный объект изобретения. Вся литература и аналогичные материалы, цитируемые в данном описании, включая, но не ограничиваясь перечисленными, патенты, заявки на патент, статьи, книги, трактаты и страницы в сети Интернет, независимо от формата такой литературы и аналогичных материалов, в явно выраженной форме включены посредством ссылки с любой целью. В случае, когда в одном или более из включенных литературных источников и аналогичных материалов термин имеет определение или используется таким образом, что это противоречит определению данного термина в настоящем описании, настоящая заявка имеет преимущественную силу. Хотя настоящая идея описана вместе с различными вариантами реализации, не предполагается, что настоящая идея ограничена такими вариантами реализации. Напротив, в объем настоящей идеи входят различные альтернативы, модификации и эквиваленты, что должно быть очевидно для специалистов в данной области.
[0024] В данной заявке предложены композиции и способы улучшенного обнаружения устойчивого к метициллину Staphylococcus aureus (MRSA) и, в частности, способы обнаружения S. aureus, несущего ген, гомологичный mecA, такого как штамм S. aureus СС130 или сс130. Устойчивость к метициллину у Staphylococcus aureus обусловлена продуктом гена mecA, кодирующего пенициллин-связывающий белок 2а (РВР-2а), β-лактам-устойчивую транспептидазу. MecA отсутствует у метициллин-чувствительного S. aureus, но широко распространен среди других видов стафилококков, включая коагулаза-отрицательного стафилококка (CoNS), такие как Staphylococcus epidermidis, Staphylococcus haemolyticus, Staphylococcus capitis, S. saprophyticus, S. lentus, S. hominus, S. cohnii, S. delphini, S. xylosus, S. muscae, S. schleiferi, S. coagulans и другие. Ген mecA высоко консервативен (Ubukata и др., 1990, Antimicrob. Agents Chemother. 34:170-172). У устойчивых к метициллину стафилококков mecA присутствует в генетическом элементе, названном стафилококковой хромосомной кассетой mec (SCCmec), которая встроена в хромосому стафилококков.
[0025] Кассеты SCCmec имеют длину в диапазоне от 20 т.п.н. до более чем 60 т.п.н., и дополнительно к гену mecA включают гены сайт-специфической рекомбиназы и мобильные генетические элементы. Кассеты SCCmec встроены в фиксированное положение, названное "attBscc", в пределах хромосомной ДНК Staphylococcus aureus, которое находится на 3'-конце открытой рамки считывания, названной ʺorfXʺ. Huletsky и др. (2004), J. Clin. Microbiol. 42(5):1875-1884. Штаммы MRSA классифицированы на основании организации кассет SCCmec (что получило название "типирование SCCmec"). Различные SCCmec классифицировали в соответствии с их генами рекомбиназы, и генетической организацией генов mecI и mecR, которые представляют собой регуляторы mecA.
[0026] Много подходов молекулярного анализа в целях обнаружения и распознавания MRSA основаны на обнаружении гена mecA. Поскольку mecA также обнаружен в CoNS, то обнаружения только лишь mecA не достаточно для определения присутствия MRSA, так как устойчивый к метициллину CoNS также будет давать положительный результат теста. Чтобы решить данную проблему, были разработаны анализы, в которых детектируют специфичные для S. aureus гены, дополнительно к mecA. См., Schuenck и др., Res. Microbiol. (2006), в печати, Shittu и др. (2006), Diagn Microbiol Infect Dis. 17 июля, Grisold и др. (2006), Methods Mol. Biol, 345: 79-89, Costa и др. (2005), Diag. Microbiol, and Infect. Dis, 51: 13-17, Mc Donald и др. (2005), J. Clin. Microbiol., 43: 6147-6149, Zhang и др. (2005), J. Clin. Microbiol. 43: 5026-5033, Hagen и др. (2005), Int J Med Microbiol. 295:77-86, Maes, и др. (2002), J. Clin. Microbiol. 40:1514-1517, Saito и др. (1995), J. Clin. Microbiol. 33:2498-2500; Ubukata и др. (1992), J. Clin. Microbiol. 30:1728-1733; Murakami и др. (1991), J. Clin. Microbiol. 29:2240-2244; Hiramatsu и др. (1992), Microbiol. Immunol. 36:445-453). Дополнительно, у Levi и Towner (2003), J. Clin. Microbiol., 41:3890-3892, и Poulsen и др. (2003), J Antimicrob Chemother., 51:419-421, описано обнаружение устойчивости к метициллину и у коагулаза-отрицательных стафилококков, и у S. aureus с помощью набора для обнаружения MRSA EVIGENE™.
[0027] Тем не менее, так как ген mecA широко распространен как у S. aureus, так и у коагулаза-отрицательных стафилококков, каждый из способов, описанных выше, не позволяет различить образцы, содержащие как метициллин-чувствительные S. aureus ("MSSA"), так и устойчивые к метициллину коагулаза-отрицательные стафилококки, и образцы, которые содержат только MRSA или которые содержат и метициллин-чувствительные S. aureus, и MRSA.
[0028] Чтобы решить данную проблему, Hiramatsu и др. разработали основанный на ПЦР метод анализа, специфично определяющий MRSA, в котором используют праймеры, гибридизующиеся с правыми концами ДНК SCCmec типов I-III, в комбинации со специфичными для хромосомы S. Aureus праймерами, соответствующими последовательности нуклеотидов с правой стороны от сайта интеграции SCCmec (патент США 6156507, далее называемый "патент 507"). Совсем недавно, Zhang и др. (2005), J. Clin. Microbiol. 43: 5026-5033, описали мультиплексный анализ для субтипирования типов с I по V SCCmec MRSA. Последовательности нуклеотидов, окружающие сайт интеграции SCCmec у других видов стафилококков (например, S. epidermidis и S. haemolyticus), отличаются от таковых, обнаруженных у S. aureus, следовательно, тесты с использованием метода мультиплексной ПЦР, в которых применяются олигонуклеотиды, гибридизующиеся с правыми концами SCCmec и хромосомы S. aureus, обеспечивают преимущество специфичного обнаружения MRSA.
[0029] ПЦР-анализ, описанный в патенте 507, также позволил разработать "MREP-типирование" (mec right extremety polymorphism) ДНК SCCmec (Ito и др. (2001), Antimicrob. Agents Chemother. 45:1323-1336; Hiramatsu и др. (1996), J. Infect. Chemother. 2:117-129). В способе MREP-типирования используется тот факт, что среди трех типов SCCmec, последовательности нуклеотидов трех типов MREP отличаются по правым концам ДНК SCCmec, расположенным рядом с сайтом интеграции.
[0030] Термин "MREJ" относится к области соединения на правом конце mec "mec right extremety junction". Последовательности нуклеиновых кислот области MREJ имеют длину приблизительно 1 т.п.н. и включают последовательности из правого конца SCCmec, а также бактериальную хромосомную ДНК справа от сайта интеграции SCCmec. Штаммы, которые классифицировали как MREP типов i-iii, соответствуют MREJ типов i-iii. MREJ типов с iv по хх ранее были описаны в Huletsky и др. (2004), J. Clin. Microbiol. 42:1875-1884; международной заявке на патент РСТ/СА02/00824, заявке на патент Соединенных Штатов 2008/0227087.
[0031] Недавно, Garcia-Alvarez и др. сообщили о бактериальных штаммах, которые согласно анализу рибосомной РНК были идентифицированы как S. aureus, и которые проявляли устойчивость к метициллину при проведении обычного тестирования на чувствительность к антибиотикам. Garcia-Alvarez и др. (2011), Lancet 11:595-603. Тем не менее, данные штаммы неожиданно проявили себя как MRSA-отрицательные при тестировании с применением описанных выше способов анализа, которые основаны на обнаружении mecA или обнаружении известных областей MREJ. Специфичность анализа ограничена определением областей MREJ. Garcia-Alvarez и др. продемонстрировали, что данные штаммы несли новый гомолог mecA, который обладал ограниченной гомологией с известными генами mecA, т.е. 63% идентичностью на уровне аминокислот и 70% идентичностью на уровне ДНК, что объясняет неспособность анализов, основанных на обнаружении гена mecA, детектировать данные MRSA. Гомолог mecA назвали mecALGA251, который в данной заявке также называют mecC. Так как анализы, в которых применяли амплификацию MREJ, были также неспособны обнаружить любой из штаммов mecALGA251 MRSA, штаммы mecALGA251 MRSA были бы неправильно определены как ложноотрицательные. Такая ошибка в диагностике и идентификации MRSA может оказать значительное влияние на результаты лечения пациента.
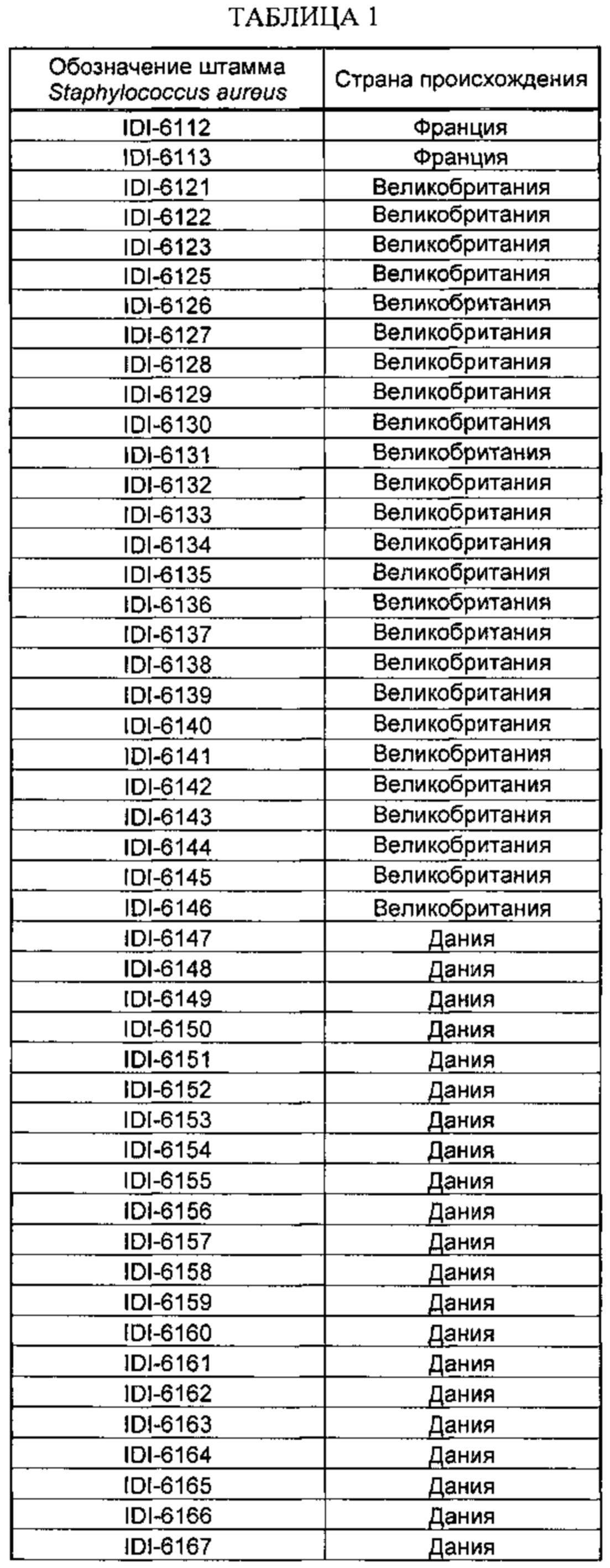
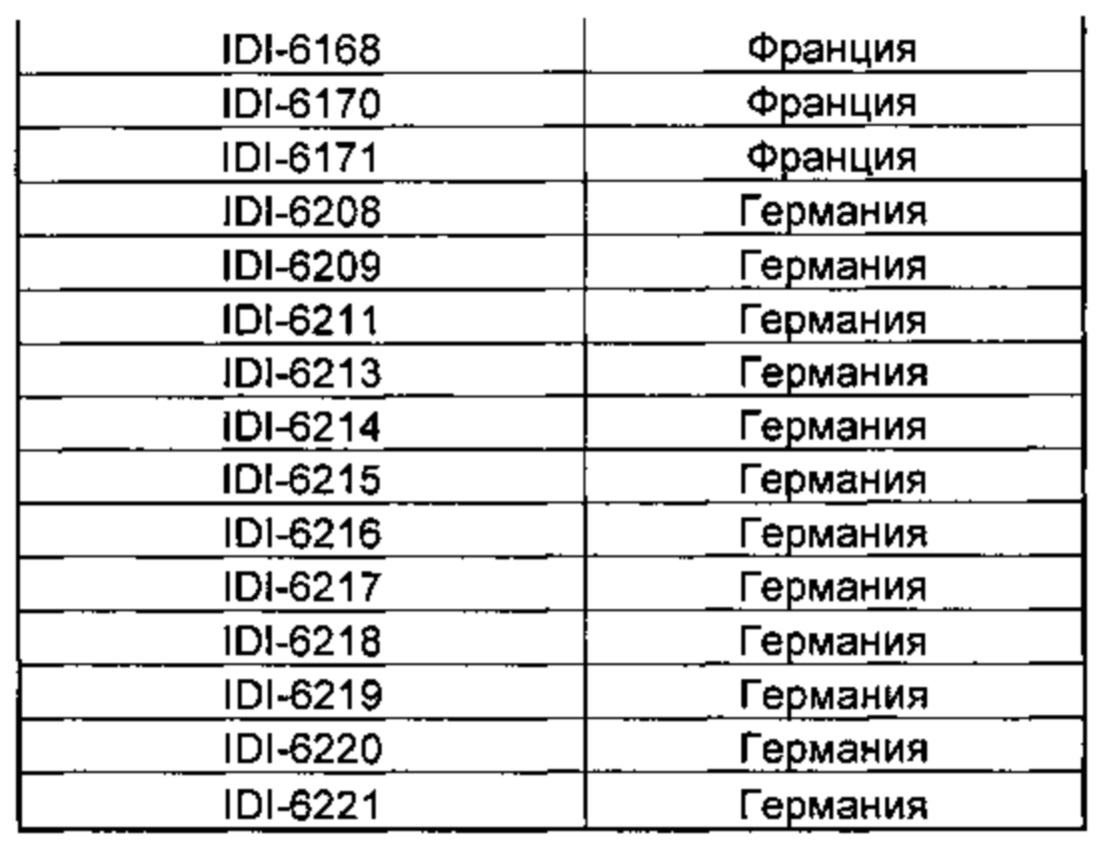
[0032] Несколько штаммов S. aureus, которые по результатам анализа с применением стандартного тестирования чувствительности к антибиотикам оказались устойчивыми к метициллину, но которые не были идентифицированы как MRSA при применении обычных коммерчески доступных систем молекулярного анализа, проанализировали дополнительно. Данные штаммы приведены в таблице 1, ниже.
[0033] В данной заявке описано, что настоящее описание основано, отчасти, на неожиданном наблюдении, состоящем в том, что подавляющее большинство проанализированных штаммов MRSA, которые несут ген, гомологичный mecA, mecALGA251/mecC, несут одинаковую последовательность MREJ. На основании этого неожиданного факта, в данной заявке предложены композиции и способы для улучшенного обнаружения MRSA. Композиции и способы, описанные в данной заявке, обеспечивают преимущество надежного и быстрого обнаружения и распознавания штаммов mecALGA251 MRSA.
Олигонуклеотиды
[0034] Согласно некоторым вариантам реализации, описанным в данной заявке, предложены олигонуклеотиды, например, праймеры для амплификации и/или специфичные к последовательности зонды. В данной заявке, термины "праймер" и "зонд" включают, но не ограничены олигонуклеотидами. Предпочтительно, олигонуклеотидные праймеры и/или зонды, описанные в данной заявке, могут иметь длину между 8 и 45 нуклеотидами. Например, праймеры и или зонды могут иметь длину по меньшей мере 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24, 25, 26, 27, 28, 29, 30, 31, 32, 33, 34, 35, 36, 37, 38, 39, 40, 41, 42, 43, 44, 45 или более нуклеотидов. Праймеры и/или зонды могут быть предусмотрены в любой подходящей форме, включая, например, связанную с твердой подложкой, жидкую и лиофилизированную форму. Последовательности праймеров и зондов, описанные в данной заявке, можно модифицировать так, чтобы они содержали дополнительные нуклеотиды на 5'- или 3'-конце, или на обоих концах. Для квалифицированного специалиста должно быть очевидно, тем не менее, что дополнительные основания на 3'-конце праймеров для амплификации (не обязательно зондов) как правило комплементарны последовательности матрицы. Последовательности праймеров и зондов, описанные в данной заявке, также можно модифицировать, чтобы удалить нуклеотиды на 5'- или 3'-конце. Для квалифицированного специалиста должно быть очевидно, что для применения в реакции амплификации, будут использоваться праймеры или зонды минимальной длины и с минимальной температурой отжига, как описано в данной заявке.
[0035] Олигонуклеотидные праймеры и зонды могут связываться со своими мишенями при температуре отжига, которая представляет собой температуру, меньшую, чем температура плавления (Tm). В данной заявке, "Tm" и "температура плавления" представляют собой взаимозаменяемые термины, которые относятся к температуре, при которой 50% всех двунитевых полинуклеотидных молекул диссоциируют на отдельные нити. Формулы для вычисления Tm полинуклеотидов хорошо известны в данной области. Например, Tm можно рассчитать с помощью следующего уравнения: Tm=69,3+0,41 x.(G+С)% - 6-50/L, где L представляет собой длину зонда в нуклеотидах. Tm гибридного полинуклеотида также можно оценить, применяя формулу, позаимствованную из анализов гибридизации в 1 М соли и широко применяемую для вычисления Tm праймеров для ПЦР: [(количество А+Т)×2°С+(количество G+С)×4°С]. См., например, С.R. Newton и др. PCR, 2ое изд., Springer-Verlag (Нью-Йорк: 1997), стр. 24. В данной области существуют другие более изощренные расчеты, при которых для расчета Tm учитывают структурные свойства, а также свойства последовательности. Температура плавления олигонуклеотида может зависеть от комплементарности между олигонуклеотидным праймером или зондом и связываемой последовательностью, и от содержания соли. В некоторых вариантах реализации, олигонуклеотидный праймер или зонд, предложенный в данной заявке, обладает Tm менее чем приблизительно 90°С в 50 мМ KCl, 10 мМ Трис-HCl буфере, например, приблизительно 89°С, 88, 87, 86, 85, 84, 83, 82, 81, 80 79, 78, 77, 76, 75, 74, 73, 72, 71, 70, 69, 68, 67, 66, 65, 64, 63, 62, 61, 60, 59, 58, 57, 56, 55, 54, 53, 52, 50, 49, 48, 47, 46, 45, 44, 43, 42, 41, 40, 39°С или менее, включая диапазоны между любыми двумя из перечисленых значений.
[0036] Ниже подробнее обсуждается, что в некоторых вариантах реализации, праймеры, описанные в данной заявке, например, праймеры для амплификации, могут быть предложены в виде пары праймеров для амплификации, например, включающей прямой праймер и обратный праймер (первый праймер для амплификации и второй праймер для амплификации). Предпочтительно, прямой и обратный праймеры имеют Tm, которые отличаются не более чем на 10°С, например, которые отличаются менее чем на 10°С, менее чем на 9°С, менее чем на 8°С, менее чем на 7°С, менее чем на 6°С, менее чем на 5°С, менее чем на 4°С, менее чем на 3°С, менее чем на 2°С или менее чем на 1°С.
[0037] Последовательности праймеров и зондов можно модифицировать путем введения нуклеотидных замен (по сравнению с целевой последовательностью) в пределах олигонуклеотидной последовательности, в том случае, если указанный олигонуклеотид обладает достаточной комплементарностью для специфичной гибридизации с целевой последовательностью нуклеиновых кислот. Таким образом, можно заменить по меньшей мере 1, 2, 3, 4 или до приблизительно 5 нуклеотидов. В данной заявке термин "комплементарный" относится к комплементарности последовательностей между участками двух полинуклеотидных цепей или между двумя участками одной и той же полинуклеотидной цепи. Первый участок полинуклеотида комплементарен второму участку того же или отличного полинуклеотида, если, когда два участка расположены антипараллельно друг к другу, по меньшей мере один нуклеотид первого участка способен образовать пару его основания с основанием второго участка. Следовательно, не обязательно, чтобы два комплементарных полинуклеотида образовывали пары оснований в каждом положении нуклеотида. "Полностью комплементарный" относится к первому полинуклеотиду, который на 100% или "полностью" комплементарен второму полинуклеотиду, а значит, образует пару оснований в каждом положении нуклеотида. "Частично комплементарный" также относится к первому полинуклеотиду, который не на 100%) комплементарен (например, на 90%, или на 80% или на 70% комплементарен) и включает неспаренные нуклеотиды в одном или более положениях нуклеотидов. В некоторых вариантах реализации, олигонуклеотид включает универсальное основание.
[0038] В данной заявке термин "гибридизация" используют в отношении спаривания комплементарных (включая частично комплементарные) полинуклеотидных цепей. На гибридизацию и силу гибридизации (т.е. силу ассоциации между полинуклеотидными цепями) влияет множество факторов, хорошо известных в данной области, включая степень комплементарности между полинуклеотидами и строгость используемых условий, на которую влияют такие характеристики, как концентрация солей, температура плавления образовавшегося гибрида, присутствие других компонентов (например, присутствие или отсутствие полиэтиленгликоля), молярность гибридизующихся цепей и содержание G:C в полинуклеотидных цепях. В некоторых вариантах реализации, например, в вариантах реализации, предусматривающих более чем один олигонуклеотид, олигонуклеотиды разработаны таким образом, что Tm одного олигонуклеотида находится в пределах 2°С от Tm другого олигонуклеотида. Подробное руководство относительно гибридизации нуклеиновых кислот можно найти в Tijssen (1993), Laboratory Techniques in Biochemistry and Molecular Biology - Hybridization with Nucleic Acid Probes, часть I, глава 2 (Elsevier, Нью-Йорк); и Ausubel и др., ред. (1995), Current Protocols in Molecular Biology, глава 2 (Greene Publishing and Wiley-Interscience, Нью-Йорк). См. Sambrook и др. (1989), Molecular Cloning: A Laboratory Manual (2oe изд., Cold Spring Harbor Laboratory Press, Плейнвью, Нью-Йорк). Далее в данной заявке обсуждается, что термин "специфичная гибридизация" или "специфично гибридизуется" относится к гибридизации полинуклеотида, например, олигонуклеотидного праймера или зонда или тому подобной молекулы, с целевой последовательностью, такой как последовательность, количество которой нужно определить в образце, целевая последовательность нуклеиновых кислот положительного контроля или тому подобная последовательность, но не с посторонними последовательностями, при условиях, обычно используемых для амплификации нуклеиновых кислот.
[0039] В некоторых вариантах реализации, праймеры и/или зонды включают олигонуклеотиды, которые гибридизуются с целевой последовательностью нуклеиновых кислот по всей длине олигонуклеотидной последовательности. Такие последовательности можно назвать "полностью комплементарными" по отношению друг к другу. Если олигонуклеотид называют "по существу комплементарным" по отношению к последовательности нуклеиновых кислот, описанной в данной заявке, две указанные последовательности могут быть полностью комплементарными, или у них могут оказаться неспаренные нуклеотиды при гибридизации, но при этом сохранится способность гибридизоваться в строгих условиях или же в стандартных условиях амплификации нуклеиновых кислот, обсуждаемых ниже. В данной заявке, термин "по существу комплементарный" относится к комплементарности между двумя нуклеиновыми кислотами, например, к комплементарному участку олигонуклеотида и целевой последовательности. Комплементарность не обязательно должна быть идеальной; между указанными двумя нуклеиновыми кислотами может быть любое количество неспаренных оснований. Тем не менее, если количество неспаренных оснований настолько велико, что гибридизация не может произойти даже при наименее строгих условиях гибридизации, указанная последовательность не является по существу комплементарной последовательностью. Если две последовательности называют в данной заявке "по существу комплементарными", это означает, что указанные последовательности достаточно комплементарны друг другу, чтобы гибридизоваться при выбранных условиях реакции. Взаимосвязь комплементарности нуклеиновых кислот и строгости гибридизации, достаточной для достижения специфичности, хорошо известна в данной области и дополнительно описана ниже в отношении идентичности последовательностей, температуры плавления и условий гибридизации. Следовательно, по существу комплементарные последовательности можно применять в любых из способов обнаружения, описанных в данной заявке. Такие зонды, например, могут быть идеально комплементарными или могут включать от 1 до множества несовпадений при условии, что условия гибридизации достаточны для того, чтобы позволить, например, отличить целевую последовательность от нецелевой последовательности. Соответственно, по существу комплементарные последовательности могут относиться к последовательностям, процент идентичности которых составляет 100, 99, 98, 97, 96, 95, 94, 93, 92, 91, 90, 89, 88, 87, 86, 85, 84, 83, 82, 81, 80, 75, 70 или менее, или любое число между ними, по сравнению с эталонной последовательностью. Например, олигонуклеотиды, описанные в данной заявке, могут включать 1, 2, 3, 4, 5 или более несовпадений и/или вырожденных оснований по сравнению с целевой последовательностью, с которой гибридизуется олигонуклеотид, при условии, что олигонуклеотиды способны специфично гибридизоваться с целевой последовательностью, например, при стандартных условиях амплификации нуклеиновых кислот.
[0040] Соответственно, в качестве примера, термин "строгие условия гибридизации" может относиться к любому из двух или к обоим описанным далее условиям: а) 6×SSC при приблизительно 45°С, а затем одна или более промывок в 0,2×SSC, 0,1% ДСН при 65°С, и b) 400 мМ NaCl, 40 мМ PIPES pH 6,4, 1 мМ ЭДТА, 50°С или 70°С в течение 12-16 часов, а затем промывка. В некоторых вариантах реализации, термин "строгие условия" может относиться к стандартным условиям амплификации нуклеиновых кислот (например, ПЦР).
[0041] В некоторых вариантах реализации, осуществляют контакт образца или пробы с набором праймеров для амплификации при стандартных условиях амплификации нуклеиновых кислот, которые обсуждаются более подробно ниже. Для обзора методики ПЦР, включая стандартные условия амплификации нуклеиновых кислот, такие как условия ПЦР, применяемой в клинической микробиологии, см. DNA Methods in Clinical Microbiology, Singleton P., опубликованную Dordrecht; Boston: Kluwer Academic (2000), Molecular Cloning to Genetic Engineering White, B.A. ред. в Methods in Molecular Biology 67: Humana Press, Totowa (1997), и "PCR Methods and Applications", с 1991 no 1995 (Cold Spring Harbor Laboratory Press). Некоторые примеры "условий амплификации нуклеиновых кислот" и "условий ПЦР" включают условия, описанные в источниках, цитируемых в данной заявке, такие как, например, 50 мМ KCl, 10 мМ Трис-HCl (pH 9,0), 0,1% Triton X-100, 2,5 мМ MgCl2, с температурой отжига, равной 72°С; или 4 мМ MgCl2, 100 мМ Трис, pH 8,3, 10 мМ KCl, 5 мМ (NH4)2SO4, 0,15 мг БСА, 4% трегалоза, с температурой отжига, равной 59°С, или 50 мМ KCl, 10 мМ Трис-HCl (pH 9,0), 0,1% Triton Х-100, 2,5 мМ MgCl2, с температурой отжига, равной 55°С, или тому подобные условия.
[0042] Праймеры, описанные в данной заявке, можно получить, применяя методики, известные в данной области, включая, но не ограничиваясь перечисленными: клонирование, расщепление (рестрикцию) подходящих последовательностей и прямой химический синтез. Способы химического синтеза, которые можно применять, чтобы получить праймеры, описанные в данной заявке, включают, но не ограничены перечисленными: фосфотриэфирный способ, описанный у Narang и др. (1979), Methods in Enzymology 68:90, фосфодиэфирный способ, описанный у Brown и др. (1979), Methods in Enzymology 68:109, диэтилфосфорамидатный способ, описанный у Beaucage и др. (1981), Tetrahedron Letters 22:1859, и способ синтеза на твердой подложке, описанный в патенте США номер 4458066. Применение автоматического синтезатора олигонуклеотидов для получения синтетических олигонуклеотидных праймеров, описанных в данной заявке, также предполагается в данной заявке.
[0043] Соответственно, некоторые варианты реализации относятся к композициям, которые включают олигонуклеотиды (например, праймеры и зонды для амплификации), которые специфично гибридизуются (например, при стандартных условиях амплификации нуклеиновых кислот, например, при стандартных условиях ПЦР, и/или при строгих услових гибридизации) с полиморфными последовательностями на правом конце SCCmec в штаммах MRSA, которые содержат последовательности MREJ типа xxi. Например, в некоторых вариантах реализации, предложены композиции, которые включают олигонуклеотиды, которые специфично гибридизуются с полиморфными последовательностями на правом конце SCCmec, присутствующими в SEQ ID NO: 1, или с комплементарной последовательностью, например, в пределах положений нуклеотидов с 1 по 164 в последовательности SEQ ID NO: 1, или комплементарной последовательности. Пример олигонуклеотида, который специфично гибридизуется с полиморфными последовательностями на правом конце SCCmec MREJ типа xxi, включая полиморфные последовательности на правом конце в пределах SEQ ID NO: 1, предложен в последовательности SEQ ID NO: 2, или комплементарной ей последовательности. Таким образом, в данной заявке предложены олигонуклеотиды, которые по существу комплементарны последовательности SEQ ID NO: 2 или комплементарной последовательности, а также олигонуклеотиды, включающие 1, 2, 3, 4 или более несовпадений или универсальных нуклеотидов по сравнению с SEQ ID NO: 2 или с комплементарной последовательностью, например, включая олигонуклеотиды, которые по меньшей мере на 80% идентичны (например по меньшей мере на 81%, 82%, 83%, 84%, 85%, 86%, 87%, 88%, 89%, 90%, 91%, 92%, 93%, 94%, 95%, 96%, 97%, 98%, 99% или 100%) идентичны) последовательности SEQ ID NO: 2 или комплементарной ей последовательности.
[0044] В некоторых вариантах реализации, композиции и способы могут включать олигонуклеотиды, например, праймеры или специфичные к последовательности зонды для амплификации, которые специфично гибридизуются с одной или более полиморфными последовательностями на правом конце в пределах областей MREJ, отличных от MREJ типа xxi. Соответственно, в некоторых вариантах реализации предложены олигонуклеотиды, например, праймеры или специфичные к последовательности зонды для амплификации, которые специфично гибридизуются (при стандартных условиях амплификации нуклеиновых кислот и/или при строгих условиях гибридизации) с полиморфными последовательностями на правом конце в пределах одной или более областей MREJ, выбранных из областей MREJ типа i, областей MREJ типа И, областей MREJ типа iii, областей MREJ типа iv, областей MREJ типа v, областей MREJ типа vi, областей MREJ типа vii, областей MREJ типа viii, областей MREJ типа ix, областей MREJ типа х, областей MREJ типа xi, областей MREJ типа xii, областей MREJ типа xiii, областей MREJ типа xiv, областей MREJ типа xv, областей MREJ типа xvi, областей MREJ типа xvii, областей MREJ типа xviii, областей MREJ типа xix и областей MREJ типа хх.
[0045] В некоторых вариантах реализации, композиции и способы могут включать олигонуклеотиды, например, праймеры для амплификации, которые специфично гибридизуются, и позволяют получить ампликон, с последовательностями mecA, или их фрагментами. Соответственно, некоторые варианты реализации включают олигонуклеотиды, например, праймеры для амплификации, которые специфично гибридизуются и позволяют получить ампликон последовательностей в пределах SEQ ID NO: 156. Некоторые варианты реализации включают олигонуклеотиды, например, праймеры для амплификации, которые специфично гибридизуются, и позволяют получить ампликон, с последовательностями mecC, например, последовательностями в пределах SEQ ID NO: 157, или их фрагментами. Некоторые варианты реализации включают олигонуклеотиды, например, праймеры для амплификации, которые специфично гибридизуются и позволяют получить ампликон специфичных для Staphylococcus aureus последовательностей. Например, в некоторых вариантах реализации предложены олигонуклеотиды, которые специфично гибридизуются и позволяют получить ампликон последовательностей nuc (например, последовательностей, полученных из SEQ ID NO: 158), последовательностей femB (например, последовательностей, полученных из SEQ ID NO: 159), последовательностей Sa442 (например, из Martineau и др. 1998, J. Clin. Microbiol. 36(3):618-623) (SEQ ID NO: 160).
[0046] В некоторых вариантах реализации предложен олигонуклеотид, который специфично гибридизуется с полиморфными последовательностями на правом конце области MREJ типа xxi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа i при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа ii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа iii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа iv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа v при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа vi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа vii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа viii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа ix при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа х при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xiii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xiv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xvi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xvii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xviii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xix при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа хх при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и/или специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с последовательностями mecA. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и/или специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с последовательностями mecC. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и/или специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с последовательностями nuc. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и/или специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с последовательностями Sa442. В некоторых вариантах реализации, специфичные к последовательности олигонуклеотиды (например, праймеры для амплификации и/или специфичные к последовательности зонды), описанные в данной заявке, специфично гибридизуются с последовательностями femB.
[0047] Примеры последовательностей областей MREJ, относящихся к вариантам реализации, описанным в данной заявке, включают, например:

[0048] Дополнительно к олигонуклеотиду с последовательностью SEQ ID NO: 2, обсуждаемому выше, который специфично гибридизуется с полиморфными последовательностями на правом конце в пределах областей MREJ типа xxi, олигонуклеотиды, которые специфичны к одной или более полиморфным последовательностям в пределах последовательностей областей MREJ, или к хромосомным последовательностям ДНК S. aureus, полезные в вариантах реализации, описанных в данной заявке, включают, но не ограничены перечисленными далее:


[0049] Дополнительно к описанному выше, для квалифицированного специалиста должно быть очевидно, что композиции и способы, описанные в данной заявке, могут включать праймеры и/или зонды для специфичного обнаружения области на правом конце последовательностей SCCmec в различных штаммах MRSA, дополнительно к упомянутым выше в данной заявке. В качестве примера, в некоторых вариантах реализации, композиции и способы, описанные в данной заявке, включают последовательности, например, праймеры и/или зонды, описанные в международной заявке на патент, номер публикации WO 08/080620, включая их варианты и комплементарные последовательности. Соответственно, композиции и способы, описанные в данной заявке, могут включать праймеры и/или зонды, перечисленные ниже:


[0050] Соответственно, в типичных вариантах реализации предложены способы и композиции для обнаружения MRSA, содержащего MREJ xxi, и один или более типов MREJ, выбранных из группы, состоящей из MREJ типа i-хх. Например, в некоторых вариантах реализации предложено обнаружение и/или распознавание MRSA, содержащего нуклеиновые кислоты MREJ типа i, ii, iii и xxi, с применением специфичных к MREJ и специфичных к хромосомной ДНК S. aureus олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено обнаружение и/или распознавание MRSA, содержащего последовательности MREJ типа i, ii, iii, iv и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено обнаружение MRSA, содержащего нуклеиновые кислоты MREJ типа i, ii, iii, iv, v, vii и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено распознавание MRSA, содержащего нуклеиновые кислоты MREJ типа i, ii, iii, iv, v, vii и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено обнаружение MRSA, содержащего нуклеиновые кислоты MREJ типа i, ii, iii, iv, v, vii и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено распознавание MRSA, содержащего нуклеиновые кислоты MREJ типа i, ii, iii, iv, v, vii и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено обнаружение MRSA, содержащего нуклеиновые кислоты типа i, ii, iii, iv, v, vi, vii, ix, xiii, xiv и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено распознавание MRSA, содержащего нуклеиновые кислоты типа i, ii, iii, iv, v, vi, vii, ix, xiii, xiv и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке. В некоторых вариантах реализации предложено обнаружение и/или распознавание MRSA, содержащего нуклеиновые кислоты типа i, ii, iii, iv, vii, xvi и xxi, с применением комбинации специфичных к MREJ олигонуклеотидов, описанных в данной заявке.
Зонды
[0051] В некоторых вариантах реализации, предложены специфичные к последовательности зонды. Зонды включают, но не ограничены олигонуклеотидами, описанными в данной заявке. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с целевой последовательностью, такой как последовательность нуклеиновых кислот области MREJ типа xxi. Например, в некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностью SEQ ID NO: 1, или с комплементарной ей последовательностью, или с частью этой последовательности (например, с ампликоном некоторой области в пределах SEQ ID NO: 1). В некоторых вариантах реализации, специфичный к последовательности зонд специфично гибридизуется и полностью или по существу комплементарен последовательности нуклеотидов, фланкированной сайтами связывания прямого праймера и обратного праймера, описанных в данной заявке. В некоторых вариантах реализации, специфичные к последовательности зонды включают по меньшей мере 5, 6, 7, 8, 9, 10, 11, 12, 13, 14, 15, 16, 17, 18, 19, 20, 21, 22, 23, 24 или 25 нуклеотидов последовательности SEQ ID NO: 2 или 3, таким образом, что специфичный к последовательности зонд перекрывается с сайтом связывания праймера для амплификации, описанного в данной заявке.
[0052] В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с orfX. В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с mecA. В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с mecC. В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с nuc. В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с femB. В некоторых вариантах реализации, предложены специфичные к последовательности зонды, которые гибридизуются с Sa442.
[0053] Для квалифицированного специалиста должно быть очевидно, что подходящие пары праймеров для амплификации и специфичные к последовательности зонды могут быть предусмотрены вместе. То есть, варианты реализации, в которых предложены праймеры для амплификации, которые специфично гибридизуются с конкретной последовательностью и позволяют получить ампликон, также могут включать специфичный к последовательности зонд, который гибридизуется и специфичен к ампликону.
[0054] В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xxi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа i при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа ii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа iii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа iv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа v при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа vi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа vii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа viii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа ix при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа х при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xiii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xiv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xv при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xvi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xvii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xviii при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа xix при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с полиморфными последовательностями на правом конце области MREJ типа хх при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями mecA при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями mecC при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями nuc при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями femB при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями Sa442 при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации.
[0055] В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с хромосомными последовательностями S. aureus, расположенными в пределах 1 т.п.н. от точки вставки элемента SCCmec в хромосомную ДНК. Например, в некоторых вариантах реализации предложен специфичный к последовательности зонд, который специфично гибридизуется с последовательностями orfX S. aureus при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. В некоторых вариантах реализации предложен специфичный к последовательности зонд, который гибридизуется с orfSA0022 при стандартных условиях амплификации нуклеиновых кислот. В некоторых вариантах реализации, специфичные к последовательности зонды, описанные в данной заявке, специфично гибридизуются с последовательностями MREP, например, с последовательностями MREP типа i, ii, iii, iv, v, vi, vii, viii, ix, x, xi, xii, xiii, xiv, xv, xvi, xvii, xviii, xix, xx или xxi. В некоторых вариантах реализации, предусмотрен более чем один специфичный к последовательности зонд. Например, в некоторых вариантах реализации, специфичные к последовательности зонды, которые специфично гибридизуются с последовательностями MREP типа xxi, предусмотрены в комбинации с одним, двумя, тремя, четырьмя, пятью, шестью, семью, восемью, девятью, десятью, одиннадцатью, двенадцатью, тринадцатью, четырнадцатью, пятнадцатью, шестнадцатью, семнадцатью, восемнадцатью, девятнадцатью или двадцатью, или более, специфичными к последовательности зондами.
[0056] В некоторых вариантах реализации, олигонуклеотидные зонды могут включать детектируемую функциональную группу. Например, в некоторых вариантах реализации, олигонуклеотидные зонды, описанные в данной заявке, могут включать радиоактивную метку. Некоторые, но не все примеры радиоактивных меток включают 3Н, 14С, 32Р и 35S. В некоторых вариантах реализации, олигонуклеотидные зонды могут включать один или более нерадиоактивных детектируемых маркеров или молекул, включая, но не ограничиваясь перечисленными: лиганды, флуорофоры, хемилюминесцентные агенты, ферменты и антитела. Другие детектируемые маркеры, которые применяют вместе с зондами, могут обеспечить повышение чувствительности способа согласно настоящему изобретению, включают биотин и радиоактивные нуклеотиды. Для среднего специалиста будет понятно, что выбор конкретной метки обуславливает способ, которым она связана с зондом. Например, олигонуклеотидные зонды метят одним или более красителями таким образом, что при гибридизации с матричной нуклеиновой кислотой получают детектируемое изменение флуоресценции. Хотя для некоторых применений могут быть желательными неспецифичные красители, специфичные к последовательности зонды могут обеспечить более точные измерения амплификации. Одна конфигурация специфичного к последовательности зонда может включать связывание одного конца зонда с флуорофором, а другого конца зонда - с гасителем. Когда зонд негибридизован, он может сохранять структуру "шпильки", в которой флуорофор гасится гасителем, таким образом предотвращая флуоресцирование флуорофора. Когда зонд гибридизован с матричной нуклеиновой последовательностью, он линеаризуется, отдаляя флуорофор от гасителя, и, таким образом, флуорофор может флуоресцировать. Другая конфигурация специфичного к последовательности зонда может включать связывание первого зонда с первым флуорофором пары для резонансного переноса энергии флуоресценции (FRET), и второго зонда - со вторым флуорофором пары для FRET. Первый зонд и второй зонд можно разработать, чтобы они гибридизовались с последовательностями ампликона, которые находятся в достаточной близости, чтобы позволить перенос энергии путем FRET, когда первый зонд и второй зонд гибридизованы с одним и тем же ампликоном.
[0057] В некоторых вариантах реализации, специфичный к последовательности зонд включает олигонуклеотид, описанный в данной заявке, конъюгированный с флуорофором. В некоторых вариантах реализации, зонд конъюгирован с двумя или более флуорофорами. Примеры флуорофоров включают: ксантеновые красители, например, красители флуоресцеин и родамин, такие как флуоресцеинизотиоцианат (FITC), моногидрохлорид этилового эфира 2-[6(этиламино)-3-(этилимино)-2,7-диметил-3Н-ксантен-9-ил]бензойной кислоты (R6G) (излучает в ответ на длину волны в диапазоне от приблизительно 500 до 560 нм), 1,1,3,3,3',3'-гексаметилиндодикарбоцианина йодид (HIDC) (излучает в ответ на длину волны в диапазоне от приблизительно 600 до 660 нм), 6-карбоксифлуоресцеин (широко известный под сокращениями FAM и F), 6-карбокси-2',4',7',4,7-гексахлорфлуоресцеин (HEX), 6-карбокси-4',5'-дихлор-2',7'-диметоксифлуоресцеин (JOE или J), N,N,N',N'-тетраметил-6-карбоксиродамин (TAMRA или Т), 6-карбокси-Х-родамин (ROX или R), 5-карбоксиродамин-6G (R6G5 или G5), 6-карбоксиродамин-6G (R6G6 или G6), и родамин 110; цианиновые красители, например, красители Су3, Су5 и Су7; кумарины, например, умбеллиферон; бензимидные красители, например, хехст 33258; фенантридиновые красители, например, техасский красный; красители на основе этидия; акридиновые красители; карбазоловые красители; феноксазиновые красители; порфириновые красители; полиметиновые красители, например, цианиновые красители, такие как Су3 (излучает в ответ на длину волны в диапазоне от приблизительно 540 до 580 нм), Су5 (излучает в ответ на длину волны в диапазоне от приблизительно 640 до 680 нм), и т.д.; борфторидные комплексы дипиррометенов (красители BODIPY) и хинолиновые красители. Конкретные представляющие интерес флуорофоры включают: пирен, кумарин, диэтиламинокумарин, FAM, флуоресцеинхлортриазинил, флуоресцеин, R110, эозин, JOE, R6G, HIDC, тетраметилродамин, TAMRA, лиссамин, ROX, нафтофлуоресцеин, техасский красный, нафтофлуоресцеин, Су3 и Су5 и тому подобные флуорофоры.
[0058] В некоторых вариантах реализации, зонд конъюгирован с гасителем. Гаситель может поглощать электромагнитное излучение и рассеивать его в виде тепла, таким образом, свечения не наблюдается. Примеры гасителей включают DABCYL, NFQ, такие как BHQ-1 или BHQ-2 (Biosearch), IOWA BLACK FQ (IDT) и IOWA BLACK RQ (IDT). В некоторых вариантах реализации, гаситель выбирают, чтобы он был спарен с флуорофором таким образом, чтобы поглощать электромагнитное излучение, испускаемое данным флуорофором. Пары флуорофор/гаситель, пригодные для композиций и способов, описанных в данной заявке, хорошо известны в данной области, и их описание можно найти, например, в S. Marras, "Selection of Fluorophore and Quencher Pairs for Fluorescent Nucleic Acid Hybridization Probes", доступной на сайте сети интернет molecular-beacons. org/download/marras,mmb06%28335%293.pdf.
[0059] В некоторых вариантах реализации, флуорофор присоединен к первому концу зонда, а гаситель присоединен ко второму концу зонда. Присоединение может включать связывание ковалентной связью, и необязательно может включать по меньшей мере одну линкерную молекулу, расположенную между зондом и флуорофором или гасителем. В некоторых вариантах реализации, флуорофор присоединен к 5'-концу зонда, а гаситель присоединен к 3'-концу зонда. В некоторых вариантах реализации, флуорофор присоединен к 3'-концу зонда, а гаситель присоединен к 5'-концу зонда. Примеры зондов, которые можно применять для количественной амплификации нуклеиновых кислот, включают зонды Molecular Beacon, зонды SCORPION™ (Sigma), зонды TAQMAN™ (Life Technologies) и тому подобные зонды. Другие технологии детектирования нуклеиновых кислот, которые полезны в вариантах реализации, описанных в данной заявке, включают, но не ограничены перечисленными: технологию зондов с наночастицами (см., Elghanian, и др. (1997), Science 277:1078-1081) и технологию зондов Amplifluor (см., патенты США с номерами: 5866366; 6090592; 6117635 и 6117986).
[0060] В некоторых вариантах реализации, описанных в данной заявке, предложены зонды, которые специфично гибридизуются с последовательностью MREJ типа xxi, или с ампликоном последовательности MREJ типа xxi, например, SEQ ID NO: 1, или с частью указанной последовательности. Соответственно, в некоторых вариантах реализации, описанных в данной заявке, предложен зонд, который гибридизуется с ампликоном, полученным в результате амплификации матрицы, содержащей SEQ ID NO: 1, с применением праймеров для амплификации SEQ ID NO: 2 и 3. Исключительно в качестве примера, в некоторых вариантах реализации предусмотрено, что зонд может включать, состоять по существу из или состоять из последовательности SEQ ID NO: 4, или ее варианта. В некоторых вариантах реализации, зонд включает флуорофор и/или гаситель, описанный в данной заявке. В некоторых вариантах реализации предусмотрено, что зонд может включать, состоять по существу из или состоять из последовательности SEQ ID NO: 82, или ее варианта. В некоторых вариантах реализации, зонд включает флуорофор и/или гаситель, описанный в данной заявке. В предпочтительных вариантах реализации, зонды могут включать SEQ ID NO: 4 или SEQ ID NO: 82, или их варианты, с флуорофором 6-карбоксифлуоресцеином ("FAM"), присоединенным к 5'-концу зонда, и гасителем IOWA BLACK Black-hole Quencher ® 2 (IDT) ("BHQ"), присоединенным к 3'-концу зонда.
Наборы
[0061] Также в данной заявке предложены наборы. Наборы, праймеры и зонды, описанные в данной заявке, можно применять для обнаружения и/или распознавания MRSA с MREJ типа xxi (и, в некоторых вариантах реализации, дополнительно для обнаружения и/или распознавания MRSA с одним или более из MREJ типов i-хх), для применения in vitro и/или in situ. Например, предполагается, что наборы можно применять в комбинации с любыми описанными ранее праймерами/зондами для обнаружения MRSA с MREJ типов i-хх. Также предполагается, что диагностические наборы, праймеры и зонды, описанные в данной заявке, можно применять отдельно или в комбинации с любыми другими способами анализа, подходящими для обнаружения и/или распознавания микроорганизмов, включая, но не ограничиваясь перечисленными: любыми анализами, основанными на обнаружении нуклеиновых кислот, любыми способами иммуноанализа, любыми ферментативными анализами, любыми способами биохимического анализа, любым фаготипированием, любым способом серологического анализа, любой дифференциальной культуральной средой, любой обогащенной культуральной средой, любой селективной культуральной средой, любой специфичной средой для анализа, любой культуральной средой для распознавания, любой культуральной средой для подсчета, любым клеточным штаммом, любой культурой определенных линий клеток и любым анализом инфицирующей способности у животных.
[0062] Соответственно, в некоторых вариантах реализации, наборы, описанные в данной заявке, будут включать олигонуклеотид, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi (например, с SEQ ID NO: 1 или комплементарной последовательностью) при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации. Например, в некоторых вариантах реализации, наборы, описанные в данной заявке, будут включать олигонуклеотид (например, первый праймер для амплификации), который включает, состоит по существу из или состоит из SEQ ID NO: 2, или ее варианта. В некоторых вариантах реализации, набор также может включать один или несколько дополнительных олигонуклеотидов, например, второй праймер для амплификации, такой как олигонуклеотид, который включает, состоит по существу из или состоит из SEQ ID NO: 3, или ее варианта, который, вместе с первым праймером для амплификации, позволит получить ампликон полиморфной области соединения на правом конце последовательностей MREJ типа xxi. В некоторых вариантах реализации, набор также может включать зонд, описанный в данной заявке. Например, в некоторых вариантах реализации, набор может включать олигонуклеотидный зонд, который включает, состоит по существу из или состоит из SEQ ID NO: 4, или ее варианта.
[0063] В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с одной или более из полиморфных последовательностей на правом конце SCCmec (MREP) в MREJ типа i, ii, iii, iv, v, vi, vii, viii, ix, x, xi, xii, xiii, xiv, xv, xvi, xvii, xviii, xix или хх. В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с последовательностями mecA. В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с последовательностями mecC. В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с последовательностями nuc. В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с последовательностями нуклеотидов, специфичными для S. aureus, например, с последовательностями femB. В некоторых вариантах реализации, наборы, описанные в данной заявке, могут включать, дополнительно к олигонуклеотиду, который специфично связывается с полиморфными последовательностями на правом конце последовательностей MREJ типа xxi при стандартных условиях амплификации, один или более олигонуклеотидов, которые специфично связываются с последовательностями Sa442.
[0064] В некоторых вариантах реализации предложены наборы, включающие реагенты и композиции для осуществления способов, описанных в данной заявке. Такой набор может включать средство для транспортировки, разделенное на отсеки, в которые рядом друг с другом помещают один или более контейнеров, таких как пробирки или флаконы. Один из контейнеров может содержать по меньшей мере один немеченый или меченый детектируемой меткой праймер или зонд, описанный в данной заявке. Указанный праймер или праймеры могут присутствовать в лиофилизированной форме или в подходящем буфере, при необходимости. Один или более контейнеров могут включать один или более ферментов или реагентов для применения, например, для реакции амплификации нуклеиновых кислот. Данные ферменты могут присутствовать сами по себе или в смесях, в лиофилизированной форме или в подходящих буферах. Примеры ферментов, пригодных для реакций амплификации нуклеиновых кислот, описанных в данной заявке, включают, но не ограничены перечисленными, Taq ДНК-полимеразу FASTSTART™, ДНК-полимеразу APTATAQ™ (Roche), ДНК-полимеразу KLENTAQ 1™ (АВ peptides Inc.), ДНК-полимеразу HOTGOLDSTAR™ (Eurogentec), ДНК-полимеразу HotStart KAPATAQ™, ДНК-полимеразу Fast HotStart KAPA2G™ (Кара Biosystems), ДНК-полимеразу Hot Start PHUSION™ (Finnzymes), или тому подобные ферменты.
[0065] Кроме того, наборы, описанные в данной заявке, могут включать все дополнительные элементы, необходимые для осуществления способов, описанных в данной заявке, такие как буферы, реагенты для выделения, ферменты, пипетки, планшеты, нуклеиновые кислоты, нуклеозидтрифосфаты, фильтровальную бумагу, гелевые материалы, материалы для переноса, материалы для авторадиографии и тому подобные элементы.
[0066] В некоторых вариантах реализации, наборы включают дополнительные реагенты, которые необходимо или удобно и/или желательно включить в реакционную смесь, полученную в способах, описанных в данной заявке; такие реагенты включают: одну или более полимераз; водную буферную среду (либо готовую, либо присутствующую в виде составных компонентов, при этом один или более из компонентов могут быть предварительно смешаны или все компоненты могут находиться отдельно) и тому подобные реагенты. Различные реагенты как компоненты наборов могут находиться в отдельных контейнерах, или все могут быть заранее объединены в смесь реагентов для объединения с матричной нуклеиновой кислотой.
[0067] Дополнительно к описанным выше компонентам, в некоторых вариантах реализации, наборы также могут включать инструкции для осуществления способов, описанных в данной заявке. Данные инструкции могут присутствовать в наборах во множестве форм, одна или более из которых может присутствовать в наборе. Одна форма, в которой могут присутствовать данные инструкции, представляет собой информацию, напечатанную на подходящем носителе или основе, например, лист или листы бумаги, на которых напечатана информация, на упаковке набора, на листке-вкладыше в упаковке и т.д. Еще одними подходящими средствами являются машиночитаемые носители, например, дискета, компакт-диск и т.д., на которые записана информация. Еще одним подходящим средством, которое может присутствовать, является интернет-сайт, который можно использовать через интернет для доступа к информации на удаленном сайте. В наборах могут присутствовать любые удобные средства.
Способы
[0068] В данной заявке предложены способы обнаружения, распознавания и/или количественного анализа в образце MRSA, имеющего нуклеиновые кислоты MREJ типа xxi. В некоторых вариантах реализации, способы включают этап осуществления контакта анализируемого образца с олигонуклеотидом, который специфично гибридизуется с полиморфными последовательностями на правом конце области SCCmec MREJ типа xxi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации.
[0069] В некоторых вариантах реализации, образец, исследуемый на присутствие MRSA, имеющего область SCCmec MREJ типа xxi, обрабатывают перед осуществлением способов, описанных в данной заявке. Например, в некоторых вариантах реализации, образец можно выделить, концентрировать или подвергнуть различным другим этапам обработки перед осуществлением способов, описанных в данной заявке. Например, в некоторых вариантах реализации, образец можно обработать, чтобы выделить нуклеиновые кислоты из образца, перед приведением в контакт образца с олигонуклеотидами, описанными в данной заявке. В данной заявке, формулировка "выделять нуклеиновые кислоты" относится к очистке нуклеиновых кислот от одного или более клеточных компонентов. Для квалифицированного специалиста должно быть очевидно, что образцы прошедшие процедуру "выделения нуклеиновых кислот", могут содержать компоненты и примеси, отличные от нуклеиновых кислот. В некоторых вариантах реализации, способы, описанные в данной заявке, осуществляют на образце без культивирования образца in vitro. В некоторых вариантах реализации, способы, описанные в данной заявке, осуществляют на образце без выделения нуклеиновых кислот из образца перед осуществлением контакта указанного образца с олигонуклеотидами, описанными в данной заявке.
i. Способы, не основанные на амплификации
[0070] В некоторых вариантах реализации, олигонуклеотид содержит детектируемую молекулу, как описано в других местах в данной заявке, и специфичную гибридизацию олигонуклеотида с полиморфными последовательностями на правом конце области SCCmec MREJ типа xxi можно детектировать, например, с помощью прямых или непрямых средств. Соответственно, некоторые варианты реализации для обнаружения и/или распознавания устойчивого к метициллину Staphylococcus aureus (MRSA), включающего нуклеиновые кислоты MREJ типа xxi, включают этапы получения тестируемого образца; и осуществления контакта образца с олигонуклеотидным зондом, который специфично гибридизуется с полиморфными последовательностями на правом конце области SCCmec MREJ типа xxi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации, где олигонуклеотидный зонд имеет длину от 10 до 45 нуклеотидов, и содержит детектируемую молекулу, при этом осуществление контакта осуществляют при условиях, позволяющих специфичную гибридизацию праймера с областью соединения на правом конце mec последовательности MREJ типа xxi, если в образце присутствует S. aureus, содержащий последовательности MREJ типа xxi. Можно определить присутствие и/или количество зонда, который специфично связан с областью соединения на правом конце mec последовательности MREJ типа xxi (если она присутствует в исследуемом образце), при этом связанный зонд свидетельствует о присутствии в образце MRSA, имеющего последовательности SCCmec MREJ типа xxi. В некоторых вариантах реализации, количество связанного зонда используют для определения количества в образце MRSA, имеющего последовательности SCCmec MREJ типа xxi.
[0071] Аналогично, в некоторых вариантах реализации, способы, не основанные на амплификации, можно применять для обнаружения дополнительных последовательностей нуклеотидов. Например, в некоторых вариантах реализации, способы, описанные в данной заявке, могут включать применение способов, не основанных на амплификации, для обнаружения присутствия и/или количества других последовательностей MREJ, например, одного или более типов MREJ i, ii, iii, iv, v, vi, vii, viii, ix, x, xi, xii, xiii, xiv, xv, xvi, xvii, xviii, xix или хх, или любой их комбинации, дополнительно к последовательностям MREJ типа xxi. В некоторых вариантах реализации, способы, описанные в данной заявке, могут включать применение способов, не основанных на амплификации, для обнаружения присутствия последовательности mecA, дополнительно к последовательностям MREJ (или MREP) типа xxi. В некоторых вариантах реализации, способы, описанные в данной заявке, могут включать применение способов, не основанных на амплификации, для обнаружения присутствия последовательности mecC, дополнительно к последовательностям (MREP) MREJ типа xxi. В некоторых вариантах реализации, способы, описанные в данной заявке, могут включать применение способов, не основанных на амплификации, для обнаружения присутствия специфичных для S. aureus последовательностей, таких как последовательности nuc, последовательности femB, последовательности Sa442, последовательности 16S рРНК, или тому подобных последовательностей, дополнительно к последовательностям (MREP) MREJ типа xxi. Соответственно, в типичном варианте реализации, в данной заявке предложены способы и композиции для одновременного обнаружения последовательностей MREJ типа xxi, i, ii, iii, iv, vii, mecA, mecC и nuc.
[0072] Этап определения можно осуществить, применяя любые способы, известные специалистам в данной области, включая, но не ограничиваясь гибридизацией in situ, после этапа осуществления контакта. Обнаружение гибридных дуплексов (т.е. зонда, специфично связанного с полиморфными последовательностями на правом конце области MREJ типа xxi) можно осуществить с помощью множества способов. Обычно, гибридизованные дуплексы отделяют от негибридизованных нуклеиновых кислот, а затем детектируют метки, связанные с дуплексами. Такие метки относятся к радиоактивным, флуоресцентным, биологическим или ферментативным меткам или меткам, обычно применяемым в данной области. Метку можно конъюгировать либо с олигонуклеотидными зондами, либо с нуклеиновыми кислотами, полученными из биологического образца. Для специалиста в данной области очевидно, что можно использовать этапы промывки для вымывания избытка образца/целевых нуклеиновых кислот или олигонуклеотидного зонда (а также несвязанного конъюгата, при необходимости). Кроме того, форматы стандартного гетерогенного анализа подходят для обнаружения гибридов с использованием меток, присутствующих на олигонуклеотидных праймерах и зондах.
[0073] Таким образом, согласно некоторым вариантам реализации, способы, описанные в данной заявке, могут включать, например, ELISA (например, в формате индикаторных полосок, в многолуночном формате или тому подобных форматах), с применением способов, известных в данной области.
ii. Способы, основанные на амплификаиии
[0074] В некоторых вариантах реализации, способы обнаружения и/или распознавания устойчивого к метициллину Staphylococcus aureus (MRSA), содержащего нуклеиновые кислоты MREJ типа xxi, включают этапы получения тестируемого образца; и осуществления контакта образца с олигонуклеотидным зондом, который специфично гибридизуется с полиморфными последовательностями на правом конце области SCCmec MREJ типа xxi при стандартных условиях амплификации нуклеиновых кислот и/или строгих условиях гибридизации, при этом олигонуклеотидный зонд имеет длину от 10 до 45 нуклеотидов.
[0075] В некоторых вариантах реализации, образец приводят в контакт при стандартных условиях амплификации, или условиях, позволяющих специфичную гибридизацию праймера с полиморфной последовательностью на правом конце mec (последовательностью MREP) последовательности MREJ типа xxi, и удлинение праймера, если в образце присутствует S. aureus, содержащий последовательности MREJ типа xxi. Соответственно, способы включают этап специфичной амплификации нуклеиновых кислот MREJ типа xxi в образцах, например, с получением ампликонов или продуктов амплификации, которые включают область соединения на правом конце mec последовательности MREJ типа xxi. В некоторых вариантах реализации, контакт указанного образца осуществляют при стандартных условиях амплификации, или условиях, обеспечивающих возможность специфичной гибридизации пары праймеров, например, с первым праймером, который гибридизуется с полиморфной последовательностью на правом конце mec (последовательностью MREP), и вторым праймером, который гибридизуется с хромосомной последовательностью S. aureus, расположенной рядом с правым концом SCCmec, т.е. orfX, с получением ампликона на протяжении области соединения SCCmec и хромосомы. В некоторых вариантах реализации предложены способы получения информации о последовательности области соединения на правом конце SCCmec путем осуществления контакта образца при стандартных условиях амплификации, или условиях, обеспечивающих возможность специфичной гибридизации пары праймеров, например, с первым праймером, который гибридизуется с полиморфной последовательностью на правом конце mec (последовательностью MREP), и вторым праймером, который гибридизуется с хромосомной последовательностью S. aureus, расположенной рядом с правым концом SCCmec, т.е. orfX, с получением ампликона на протяжении области соединения SCCmec и хромосомы.
[0076] В некоторых вариантах реализации, образец приводят в контакт, например, одновременно (как в мультиплексной ПЦР) или последовательно с приведением в контакт со специфичным к MREJ типа xxi олигонуклеотидом(ами), при одинаковых стандартных условиях амплификации, с дополнительными праймерами, которые обеспечивают возможность специфичной амплификации одной или более дополнительных последовательностей типов MREJ, например, одной или более из последовательностей MREJ типа i, ii, iii, iv, v, vi, vii, viii, ix, x, xi, xii, xiii, xiv, xv, xvi, xvii, xviii, xix или хх. В некоторых вариантах реализации, образец приводят в контакт, например, одновременно (мультиплексная ПЦР) или последовательно с приведением в контакт со специфичным к MREJ типа xxi олигонуклеотидом(ами), при одинаковых стандартных условиях амплификации, с дополнительными праймерами, которые обеспечивают возможность специфичной амплификации последовательностей mecA и/или mecC. В некоторых вариантах реализации, образец приводят в контакт, например, одновременно (мультиплексная ПЦР) или последовательно с приведением в контакт со специфичным к MREJ типа xxi олигонуклеотидом(ами), при одинаковых стандартных условиях амплификации, с дополнительными праймерами, которые обеспечивают возможность специфичной амплификации одной или более последовательностей, которые уникальны для S. aureus (специфичные для S. aureus последовательности), таких как nuc, femB, Sa442 и тому подобные последовательности. Соответственно, в некоторых вариантах реализации, образец приводят в контакт с праймерами, специфичными для последовательностей MREP типа xxi, i, ii, iii, iv, v, vii, ix, xiii, xiv и xxi, а также последовательностей mecA, mecC и nuc.
[0077] В некоторых вариантах реализации, способы включают распознавание конкретного типа последовательности (например, последовательности MREJ типа xxi). Например, в вариантах реализации, включающих специфичную амплификацию только последовательностей MREJ типа xxi, присутствие или отсутствие ампликона свидетельствует о присутствии или отсутствии в образце последовательностей MREJ типа xxi. В некоторых вариантах реализации, указанные способы включают специфичную амплификацию дополнительных последовательностей, например, последовательностей MREJ, последовательностей тес, последовательностей, специфичных для S. aureus и тому подобных последовательностей, в мультиплексе вместе со специфичной амплификацией последовательностей MREJ типа xxi. В вариантах реализации, которые включают мультиплексную амплификацию, в некоторых вариантах реализации, способы могут включать распознавание или обнаружение специфичных последовательностей, например, с применением специфичных к последовательности зондов, которые гибридизуются только с одним ампликоном. В некоторых вариантах реализации, способы не позволяют различить некоторые или все различные возможные ампликоны, присутствующие в образце после амплификации. Например, в некоторых вариантах реализации, специфичный к последовательности зонд, который гибридизуется с orfX, можно применять для обнаружения присутствия ампликонов одного или более типов MREJ. В некоторых вариантах реализации, способы включают обнаружение продукта амплификации, без специфичного обнаружения конкретной последовательности.
[0078] Несколько способов специфичной амплификации целевых нуклеиновых кислот известно в данной области, и они пригодны в вариантах реализации, описанных в данной заявке. Лишь некоторые из примеров способов амплификации включают полимеразную цепную реакцию (ПЦР; см. Saiki и др., 1985, Science 230:1350-1354, включена в данную заявку посредством ссылки), лигазную цепную реакцию (ЛЦР; см. Wu и др., 1989, Genomics 4:560-569; Barringer и др., 1990, Gene 89:117-122; Barany, 1991, Proc. Natl. Acad. Sci. USA 88:189-193, каждая из которых включена в данную заявку посредством ссылки), транскрипционно-опосредованную амплификацию (ТМА; см. Kwoh и др., 1989, Proc. Natl. Acad. Sci. USA 86:1173-1177, включена в данную заявку посредством ссылки), самоподдерживающуюся репликацию последовательностей (3SR; см. Guatelli и др., 1990, Proc. Natl. Acad. Sci. USA 87:1874-1878, включена в данную заявку посредством ссылки), амплификацию по типу катящегося кольца (RCA), амплификацию, основанную на последовательности нуклеиновых кислот (NASBA), Q-бета репликазную систему (Lizardi и др., 1988, BioTechnology 6:1197-1202, включена в данную заявку посредством ссылки) и амплификацию с замещением цепи (SDA; см. Walker и др., 1992, Proc. Natl. Acad. Sci. USA 89:392-396; Walker и др., 1992, Nuc. Acids. Res. 20:1691-1696; и ЕР 0497272, каждая из которых включена в данную заявку посредством ссылки), включая термофильную SDA (tSDA).
[0079] В различных вариантах реализации, способы, описанные в данной заявке, полезны для обнаружения присутствия нуклеиновых кислот или последовательностей SCCmec MREJ типа xxi в клинических образцах. Например, в некоторых вариантах реализации, способы, описанные в данной заявке, полезны для обнаружения и идентификации образцов S. aureus, содержащего области MREJ типа xxi, концентрация бактерий в которых находится в пределах физиологических диапазонов (т.е. концентрация бактерий в образце, собранном у субъекта, инфицированного бактериями). Таким образом, образец можно непосредственно подвергнуть скринингу без необходимости выделения, концентрирования или наращивания (например, культивирования) бактериальной популяции, чтобы обнаружить присутствие MRSA, содержащего нуклеиновые кислоты MREJ типа xxi. В различных вариантах реализации, способы, описанные в данной заявке, позволяют обнаружить присутствие MRSA, содержащего нуклеиновые кислоты MREJ типа xxi, в образце, концентрация бактерий в котором составляет приблизительно 1 КОЕ/мл, 10 КОЕ/мл, 100 КОЕ/мл, 1×103 КОЕ/мл, 1×103 КОЕ/мл, приблизительно 1×104 КОЕ/мл, приблизительно 1×105 КОЕ/мл или приблизительно 1×106 КОЕ/мл, или любое число между ними.
[0080] В некоторых вариантах реализации, способы, описанные в данной заявке, включают осуществление ПЦР или количественной ПЦР для получения ампликона. Множество различных протоколов ПЦР и количественной ПЦР известно в данной области и проиллюстрировано ниже в данной заявке, и их можно непосредственно применять или адаптировать для применения с использованием описанных в данной заявке композиций для обнаружения и/или распознавания в образце MRSA, имеющего нуклеиновые кислоты MREJ типа xxi.
[0081] Как правило, при ПЦР целевую полинуклеотидную последовательность амплифицируют путем осуществления реакции по меньшей мере с одним олигонуклеотидным праймером или парой олигонуклеотидных праймеров. Указанный праймер(ы) специфично гибридизуется с комплементарной областью целевой нуклеиновой кислоты и ДНК-полимераза удлиняет праймер(ы), что приводит к амплификации целевой последовательности. При условиях, подходящих для получения с помощью полимеразы продуктов амплификации нуклеиновых кислот, фрагмент нуклеиновой кислоты одного размера доминирует среди продуктов реакции (целевая полинуклеотидная последовательность, которая представляет собой продукт амплификации). Цикл амплификации повторяют, чтобы увеличить концентрацию единственной целевой полинуклеотидной последовательности. Реакцию можно осуществить в любом термоциклере, широко применяемом для ПЦР. Тем не менее, предпочтительны термоциклеры с возможностью измерения флуоресценции в реальном времени, например, SMARTCYCLER® (Cepheid, Саннивейл, Калифорния), ABI PRISM 7700® (Applied Biosystems, Фостер Сити, Калифорния), ROTOR-GENE™; (Corbett Research, Сидней, Австралия), LIGHTCYCLER® (Roche Diagnostics Corp, Индианаполис, Индиана), ICYCLER® (Biorad Laboratories, Геркулес, Калифорния) и МХ4000® (Stratagene, Ла-Хойя, Калифорния)
[0082] В некоторых вариантах реализации предложены способы, включающие количественную ПЦР (также называемую ПЦР в реальном времени). Количественная ПЦР позволяет осуществлять количественные измерения, а также обладает преимуществом меньшего времени анализа и меньшей контаминации. В данной заявке, "количественная ПЦР" (или "количественная ПЦР в реальном времени") относится к непосредственному отслеживанию развития ПЦР-амплификации, так как она происходит без необходимости многократного отбора образцов продуктов реакции. При количественной ПЦР, продукты реакции можно отслеживать с помощью механизма передачи сигналов (например, флуоресценции), так как они образуются и отслеживаются после того, как сигнал оказывается выше фонового уровня, но до того, как реакция достигнет плато. Количество циклов, необходимое для достижения детектируемого или "порогового" уровня флуоресценции (называемого в данной заявке пороговым циклом или "ПЦ"), изменяется прямо пропорционально концентрации амплифицируемых мишеней с начала процесса ПЦР, позволяя измерить интенсивность сигнала, чтобы получить меру количества целевой нуклеиновой кислоты в образце в реальном времени.
[0083] Способы отладки ПЦР и количественной ПЦР хорошо известны специалистам в данной области. В реакционную смесь входит минимальное количество матричной нуклеиновой кислоты (например, присутствующей в тестируемых образцах, за исключением случая отрицательного контроля, описанного ниже) и олигонуклеотидные праймеры и/или зонды в комбинации с подходящими буферами, солями и тому подобными компонентами, и подходящая концентрация полимеразы нуклеиновых кислот. В данной заявке, "полимераза нуклеиновых кислот" относится к ферменту, который катализирует полимеризацию нуклеозидтрифосфатов. Как правило, фермент будет запускать синтез с 3'-конца праймера, гибридизованного с целевой последовательностью, и синтез будет протекать в 5'-направлении вдоль матрицы до тех пор, пока синтез не остановится. Подходящая концентрация включает такую концентрацию, которая катализирует данную реакцию в описанных в настоящем изобретении способах. Известные ДНК-полимеразы, пригодные в способах, описанных в данной заявке, включают, например, ДНК-полимеразу I Е. coli, ДНК-полимеразу Т7, ДНК-полимеразу Thermus thermophilus (Tth), ДНК-полимеразу Bacillus stearothermophilus, ДНК-полимеразу Thermococcus litoralis, ДНК-полимеразу Thermus aquaticus (Taq) и ДНК-полимеразу Pyrococcus furiosus (Pfu), Taq ДНК-полимеразу FASTSTART™, ДНК-полимеразу APTATAQ™ (Roche), ДНК-полимеразу KLENTAQ1™ (AB peptides Inc.), ДНК-полимеразу HOTGOLDSTAR™ (Eurogentec), HotStart ДНК-полимеразу KAPATAQ™, Fast HotStart ДНК-полимеразу KAPA2G™ (Кара Biosystems), Hot Start ДНК-полимеразу PHUSION™ (Finnzymes) или тому подобные полимеразы.
[0084] Дополнительно к описанным выше компонентам, реакционная смесь согласно настоящим способам включает праймеры, зонды и дезоксирибонуклеозидтрифосфаты (дНТФ).
[0085] Обычно, реакционная смесь дополнительно содержит четыре различных типа дНТФ, соответствующих четырем встречающимся в природе нуклеозидным основаниям, т.е. дАТФ, дТТФ, дЦТФ и дГТФ. В способах согласно настоящему изобретению, каждый дНТФ, как правило, будет присутствовать в количестве, находящемся в диапазоне приблизительно от 10 до 5000 мкМ, обычно приблизительно от 20 до 1000 мкМ, приблизительно от 100 до 800 мкМ или приблизительно от 300 до 600 мкМ.
[0086] Реакционная смесь может дополнительно содержать водную буферную среду, которая включает источник одновалентных ионов, источник двухвалентных катионов и буферное вещество. Можно применять любой удобный источник одновалентных ионов, такой как хлорид калия, ацетат калия, ацетат аммония, глутамат калия, хлорид аммония, сульфат аммония и тому подобные источники. Двухвалентным катионом может быть магний, марганец, цинк и тому подобные катионы, при этом катион обычно представляет собой магний. Можно применять любой удобный источник катиона магния, включая хлорид магния, ацетат магния и тому подобные источники. Количество магния, присутствующее в буфере, может находиться в диапазоне от 0,5 до 10 мМ, и может находиться в диапазоне от приблизительно 1 до приблизительно 6 мМ, или от приблизительно 3 до приблизительно 5 мМ. Примеры буферных веществ или солей, которые могут присутствовать в буфере, включают Трис, Трицин, HEPES, MOPS и тому подобные вещества, при этом количество буферного вещества, как правило, будет находиться в диапазоне от приблизительно 5 до 150 мМ, обычно от приблизительно 10 до 100 мМ, и чаще от приблизительно 20 до 50 мМ, при этом в некоторых предпочтительных вариантах реализации буферное вещество будет присутствовать в количестве, достаточном для обеспечения pH в диапазоне от приблизительно 6,0 до 9,5, например, приблизительно pH 6,0, 6,5, 7,0, 7,5, 8,0, 8,5, 9,0 или 9,5. Другие агенты, которые могут присутствовать в буферной среде, включают хелатирующие агенты, такие как ЭДТА, ЭГТА и тому подобные агенты. В некоторых вариантах реализации, реакционная смесь может включать БСА или тому подобные агенты. Кроме того, в некоторых вариантах реализации, реакционные смеси могут включать криопротектор, такое как трегалоза, особенно если реагенты предусмотрены в виде общей реакционной смеси (мастер-микс), которую можно хранить в течение некоторого времени.
[0087] При приготовлении реакционной смеси, различные составляющие ее компоненты можно объединить в любом удобном порядке. Например, буфер можно объединить с праймером, полимеразой, а затем с матричной нуклеиновой кислотой, или все различные составляющие смесь компоненты можно объединить одновременно с получением реакционной смеси.
[0088] В качестве альтернативы, доступные для приобретения предварительно смешанные реагенты можно применять в способах, описанных в данной заявке, следуя инструкциям производителя, или модифицировать, чтобы улучшить условия реакции (например, модификация концентрации буфера, концентрации катионов или концентрации дНТФ, при необходимости), включая, например, универсальную общую реакционную смесь для ПЦР TAQMAN® (Applied Biosystems), OMNIMIX® или SMARTMIX® (Cepheid), IQ™ Supermix (Bio-Rad Laboratories), LIGHTCYCLER® FastStart (Roche Applied Science, Индианаполис, Индиана), или общую реакционную смесь для количественной ПЦР BRILLIANT® (Stratagene, Ла-Хойя, Калифорния).
[0089] Реакционную смесь можно подвергнуть условиям реакции удлинения праймеров ("условиям, достаточным для получения с помощью полимеразы продуктов амплификации нуклеиновых кислот"), т.е. условиям, которые позволяют опосредованное полимеразой удлинение праймеров путем добавления нуклеотидов к концу молекулы праймера с применением матричной нити в качестве матрицы. Во многих вариантах реализации, условия реакции удлинения праймеров представляют собой условия амплификации, которые включают множество циклов реакции, при этом каждый цикл реакции включает: (1) этап денатурации, (2) этап отжига и (3) этап элонгации. Ниже обсуждается, что, в некоторых вариантах реализации, протокол амплификации не включает конкретное время, заранее определенное для отжига, а вместо этого включает только конкретное время, заранее определенное для денатурации и элонгации. Количество циклов реакции будет изменяться в зависимости от осуществляемого способа, но, как правило, будет составлять по меньшей мере 15, чаще по меньшей мере 20, и может достигать столь большого количества, как 60 или более циклов, при этом количество различных циклов, как правило, будет находиться в диапазоне приблизительно от 20 до 40. Для способов, в которых осуществляют более чем приблизительно 25, обычно более чем приблизительно 30 циклов, может быть удобно или желательно ввести в реакционную смесь дополнительное количество полимеразы, для того, чтобы условия, подходящие для ферментативного удлинения праймеров, сохранялись.
[0090] Этап денатурации включает нагрев реакционной смеси до повышенной температуры и поддержание повышенной температуры смеси в течение периода времени, достаточного для того, чтобы любая двунитевая или гибридизованная нуклеиновая кислота, присутствующая в реакционной смеси, диссоциировала. Для денатурации, температура реакционной смеси, как правило, будет поднята и будет поддерживаться на уровне температуры в диапазоне от приблизительно 85 до 100°С, обычно, от приблизительно 90 до 98°С, и чаще от приблизительно 93 до 96°С, в течение периода времени в диапазоне от приблизительно 3 до 120 сек, обычно приблизительно 3 сек.
[0091] После денатурации, реакционную смесь можно подвергнуть условиям, достаточным для отжига праймера на матричную нуклеиновую кислоту, присутствующую в смеси (если она есть), и для полимеризации нуклеотидов с концами праймера таким образом, что праймер удлиняется в 5'-3' направлении, применяя в качестве матрицы нуклеиновую кислоту, с которой он гибридизуется, т.е. условиям, достаточным для ферментативного получения продукта элонгации праймеров. В некоторых вариантах реализации, процессы отжига и элонгации происходят на одном этапе. Температура, до которой снижают температуру реакционной смеси, чтобы достичь данных условий, как правило, будет выбрана таким образом, чтобы обеспечить оптимальную эффективность и специфичность, и, как правило, будет находиться в диапазоне от приблизительно 50 до 85°С, обычно от приблизительно 55 до 70°С, и чаще от приблизительно 60 до 68°С. В некоторых вариантах реализации, условия отжига могут поддерживаться в течение периода времени в диапазоне от приблизительно 15 сек до 30 мин, обычно от приблизительно 20 сек до 5 мин, или от приблизительно 30 сек до 1 минуты, или приблизительно 30 секунд.
[0092] Данный этап необязательно может включать каждый из этапов отжига и элонгации, с изменением и оптимизацией температуры и отрезка времени для каждого этапа. При двухэтапном отжиге и элонгации, этапу отжига позволяют проходить, как описано выше. После отжига праймера на матричную нуклеиновую кислоту, реакционную смесь дополнительно подвергнут условиям, достаточным для полимеризации нуклеотидов с концами праймеров, как описано выше. Чтобы добиться условий полимеризации, температуру реакционной смеси, как правило, будут повышать или поддерживать на уровне температуры в диапазоне от приблизительно 65 до 75°С, обычно от приблизительно 67 до 73°С, и поддерживать в течение периода времени в диапазоне от приблизительно 15 сек до 20 мин, обычно от приблизительно 30 сек до 5 мин. В некоторых вариантах реализации, способы, описанные в данной заявке, не включают отдельные этапы отжига и элонгации. Предпочтительнее, способы включают этапы денатурации и элонгации, без какого-либо этапа, предназначенного конкретно для отжига.
[0093] Описанные выше циклы денатурации, отжига и элонгации можно осуществить, применяя автоматическое устройство, широко известное как термоциклер. Термоциклеры, которые можно применять, описаны в других местах в данной заявке, а также в патентах США с номерами 5612473; 5602756; 5538871 и 5475610; содержание которых включено в данную заявку посредством ссылки.
[0094] Способы, описанные в данной заявке, также можно применять для не основанных на ПЦР применений для обнаружения целевой последовательности нуклеиновых кислот, в которых такая мишень может быть иммобилизована на твердой подложке. Способы иммобилизации последовательности нуклеиновых кислот на твердой подложке известны в данной области и описаны у Ausubel и др., ред. (1995), Current Protocols in Molecular Biology (Greene Publishing and Wiley-Interscience, Нью-Йорк), и в протоколах, предоставленных производителями, например, для мембран: Pall Corporation, Schleicher & Schuell; для магнитных гранул: Dynal; для культуральных планшетов: Costar, Nalgenunc; для носителей с набором гранул: Luminex и Becton Dickinson; и для других подложек, полезных согласно вариантам реализации, предложенным в данной заявке, CPG, Inc.
[0095] Изменения конкретных количеств различных реагентов и условий проведения ПЦР или других подходящих процедур амплификации (например, буферные условия, время проведения циклов и т.д.), которые приводят к аналогичным результатам амплификации или обнаружения/количественного анализа, известны специалистам в данной области и рассматриваются как эквивалентные. В одном варианте реализации, заявленное обнаружение с помощью количественной ПЦР обладает чувствительностью обнаружения, меньшей чем 50 копий (предпочтительно меньше чем 25 копий, более предпочтительно меньше чем 15 копий, еще более предпочтительно меньше чем 10 копий, например, 5, 4, 3, 2 или 1 копия) целевой нуклеиновой кислоты (т.е. нуклеиновых кислот MREJ типа xxi) в образце.
Контроли
[0096] Анализы, описанные в данной заявке, в некоторых случаях могут включать контроли. Реакции ПЦР или количественной ПЦР, описанные в данной заявке, могут включать различные контроли. Такие контроли могут включать отрицательный контроль "без матрицы", в котором праймеры, буфер, фермент(ы) и другие необходимые реагенты (например, MgCl2, нуклеотиды и тому подобные реагенты) подвергают циклам реакции без добавления тестируемого образца. Это позволяет гарантировать то, что реагенты не контаминированы полинуклеотидами, которые реагируя с праймерами, дают ложные продукты амплификации. Дополнительно к контролям "без матрицы", отрицательные контроли также могут включать реакции амплификации с добавлением в реакцию неспецифичной целевой нуклеиновой кислоты, или могут представлять собой образцы, полученные с применением любого или всех этапов получения образца (начиная с выделения нуклеиновой кислоты и заканчивая получением амплификата) без добавления тестируемого образца (например, в каждом этапе либо отсутствует тестовый образец, либо присутствует образец, который свободен от устойчивых к карбапенемам микроорганизмов).
[0097] В некоторых вариантах реализации, способы, описанные в данной заявке, могут включать положительный контроль, например, чтобы убедиться, что способы и реагенты работают, как ожидалось. Положительный контроль может включать известную мишень, которая не относится к целевым нуклеиновым кислотам MREJ типа xxi, описанным в данной заявке. Перед амплификацией, нуклеиновую кислоту положительного контроля (например, в виде плазмиды, которая либо линеаризована, либо не линеаризована) можно добавить в реакцию амплификации. Одна реакция может содержать либо матрицу положительного контроля, либо отрицательный контроль, либо матрицу образца, или одна реакция может содержать как матрицу образца, так и положительный контроль. Предпочтительно, положительный контроль будет включать последовательности, которые по существу комплементарны прямому и обратному праймерам для амплификации MREJ типа xxi, полученным с помощью последовательностей MREJ типа xxi, описанных в данной заявке, таким образом, что пара праймеров для амплификации, используемая для амплификации последовательностей MREJ типа xxi, также будет амплифицировать контрольные нуклеиновые кислоты при тех же условиях анализа. В некоторых вариантах реализации, ампликон, полученный по матрице нуклеиновых кислот положительного контроля, длиннее, чем целевой ампликон. Предпочтительно, за исключением последовательностей в нуклеиновых кислотах положительного контроля, которые комплементарны или по существу комплементарны прямому и обратному праймерам, нуклеиновая кислота положительного контроля не обладает существенным подобием с целевым ампликоном/последовательностями MREJ типа xxi, описанными в данной заявке. Другими словами, за пределами прямого и обратного праймеров, ампликон положительного контроля предпочтительно менее чем на 80%, менее чем на 70%, менее чем на 60%, менее чем на 50%, менее чем на 40%, менее чем на 30%, менее чем на 20%, и еще более предпочтительно, менее чем на 10% идентичен полинуклеотиду положительного контроля, например, когда идентичность последовательностей сравнивают, применяя средство NCBI BLAST ALIGN.
[0098] Положительные и/или отрицательные контроли можно применять для установки параметров, в пределах которых тестируемый образец будет классифицирован как содержащий или не содержащий MRSA, включающий последовательности MREJ типа xxi. Например, при реакции количественной ПЦР, пороговый цикл, при котором ампликон обнаруживают в образце положительного контроля, можно применять для установления порога для классификации образца как "положительного", а пороговый цикл, при котором ампликон обнаруживают в образце отрицательного контроля, можно применять для установления порога для классификации образца как "отрицательного". Для каждого контроля можно использовать ПЦ из одной реакции, или можно использовать медианное или среднее значение по повторным образцам. В еще одном варианте реализации, можно использовать известные контрольные значения. Минимальный уровень обнаружения для каждого из отрицательного и положительного контролей обычно устанавливают на нижней границе 95% доверительного интервала среднего значения ПЦ для нескольких реакций. Данное значение можно изменять в зависимости от требований диагностического анализа.
[0099] Предпочтительно, контроли ПЦР должны тестироваться одновременно с тестируемым образцом, с применением тех же реагентов, в той же реакции амплификации.
[0100] В некоторых вариантах реализации предложено определение идентичности и/или количества целевых продуктов амплификации во время реакции амплификации, например, в реальном времени. Например, в некоторых вариантах реализации предложено проводить измерение, например, степени связывания зонда, который специфично связывается с нуклеиновыми кислотами целевого ампликона и/или ампликонов положительного контроля (например, по появлению флуоресценции). В некоторых вариантах реализации, вместо того, чтобы использовать специфичные к последовательности олигонуклеотидные зонды, в способах могут применяться неспецифичные к последовательности зонды, которые неспецифично связываются с двунитевой нуклеиновой кислотой, например, интеркалирующие агенты или тому подобные агенты. Интеркалирующие агенты обладают относительно низким уровнем флуоресценции, когда они не связаны, и относительно высоким уровнем флуоресценции при связывании с двунитевыми нуклеиновыми кислотами. По этой причине, интеркалирующие агенты можно применять для отслеживания накопления двунитевых нуклеиновых кислот в процессе реакции амплификации нуклеиновых кислот. Примеры таких неспецифичных красителей включают интеркалирующие агенты, такие как SYBR Green I™ (Molecular Probes), йодид пропидия, бромистый этидий, LC green, SYTO9, флуоресцентный краситель EVAGREEN®, CHROMOFY®, ВЕВО и тому подобные агенты, которые флуоресцируют и вырабатывают детектируемый сигнал в присутствии двунитевых нуклеиновых кислот. Измерения можно осуществлять в определенный момент во время каждого цикла реакции амплификации, например, после каждого этапа элонгации (перед каждым этапом денатурации). Независимо от того, используется ли специфичный к последовательности олигонуклеотидный зонд, или не специфичный к последовательности олигонуклеотидный зонд, измерения количества зонда, который специфично связан с нуклеиновыми кислотами целевого ампликона, и/или с ампликонами положительного контроля, можно осуществлять непрерывно на всем протяжении каждого цикла.
[0101] В качестве альтернативы, в некоторых вариантах реализации, идентичность/количество ампликонов (например, целевого и/или положительного контроля) можно подтвердить после завершения реакции амплификации, применяя стандартные молекулярные методики, включая (например) саузерн-блоттинг, дот-блоттинг и тому подобные способы.
ПРИМЕРЫ
[0102] Следующие примеры предложены, чтобы продемонстрировать конкретные ситуации и условия, в которых можно применять данную методику, и не предполагаются ограничивающими объем настоящего изобретения и формулу изобретения, включенную в настоящее описание.
ПРИМЕР 1: ОБНАРУЖЕНИЕ И РАСПОЗНАВАНИЕ MRSA С MREJ ТИПА XXI В КЛИНИЧЕСКИХ ОБРАЗЦАХ
[0103] Осуществляли количественную реакцию ПЦР для обнаружения и распознавания MRSA, содержащего нуклеиновые кислоты MREJ типа xxi. Клинические образцы собирали у пациентов, применяя Amies Liquid Swabs (Copan Diagnostics, Inc).
[0104] ДНК возможно выделяли из клинических образцов, применяя набор BD GeneOhm™ Lysis (Becton Dickinson), следуя инструкциям производителя. Образец выделенной ДНК приводили в контакт с праймерами, которые специфично гибридизуются при стандартных условиях амплификации с последовательностями orfX, специфичными для видов S. aureus, и с полиморфными последовательностями на правом конце MREJ типа xxi, т.е. по 0,2-0,7 мкМ каждой из поледовательностей SEQ ID NO: 2 и 3, 0,3 мкМ дНТФ (Roche), 4 мМ MgCl2 (SIGMA), 2,8 единиц Taq-полимеразы FASTSTART® (Roche), 100 мМ трис, pH 8,3 (EMD), 10 мМ KCl (LaboratoireMat), 5 мМ (NH4)2SO4 (SIGMA), 0,15 мг/мл БСА (SIGMA), 4% трегалозы (SIGMA). В реакционную смесь также добавляли детектируемые зонды Molecular beacon (молекулярные маяки), которые специфично гибридизуются с продуктами амплификации областей соединения на правом конце MREJ типа xxi, которые содержат детектируемые молекулы, обнаруживаемые с помощью устройства BD MAX™ (Becton Dickinson), SMARTCYCLER® (Cepheid) или другого устройства, разработанного для ПЦР в реальном времени, в каналах FAM, техасского красного и Tet.
[0105] ПЦР осуществляли на BD MAX™ (Becton Dickinson) или SMARTCYCLER® (Cepheid), применяя одинаковые параметры циклов, описанные далее:
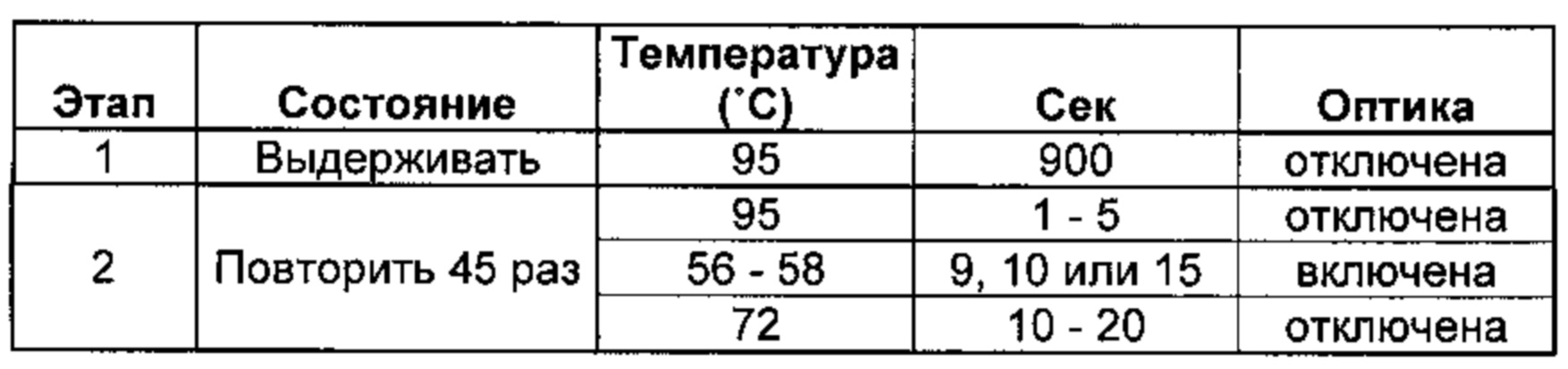
[0106] Пороговый цикл (ПЦ) в каналах FAM, техасского красного и ТЕТ определяли, применяя программное обеспечение BD МАХ™ или SMARTCYCLER®.
ПРИМЕР 2: МУЛЬТИПЛЕКСНОЕ ОБНАРУЖЕНИЕ MRSA С MREJ ТИПА I-XXI В КЛИНИЧЕСКИХ ОБРАЗЦАХ
[0107] Осуществляли мультиплексную реакцию амплификации для обнаружения присутствия MRSA, содержащего MREJ любого из типов i-vii, ix, xiii, xiv и xxi. Клинические образцы собирали у пациентов, применяя Amies Liquid Swabs (Copan Diagnostics, Inc).
[0108] ДНК возможно выделяли из клинических образцов, применяя набор BD GeneOhm™ Lysis (Becton Dickinson), следуя инструкциям производителя. Образец выделенной ДНК приводили в контакт с праймерами, которые специфично гибридизуются при стандартных условиях амплификации с последовательностями orfX, специфичными для видов S. aureus, и с полиморфными последовательностями на правом конце MREJ типа i-vii, ix, xiii, xiv и xxi, т.е. по 0,2-0,7 мкМ каждой из поледовательностей SEQ ID NO: 2, 3, 39, 77 и 81, 0,3 мкМ дНТФ (Roche), 4 мМ MgCl2 (SIGMA), 2,8 единиц Taq-полимеразы FASTSTART® (Roche), 100 мМ трис, pH 8,3 (EMD), 10 мМ KCl (LaboratoireMat), 5 мМ (NH4)2SO4 (SIGMA), 0,15 мг/мл БСА (SIGMA), 4% трегалозы (SIGMA). В реакционную смесь также добавляли детектируемые зонды - молекулярные маяки, - которые специфично гибридизуются с продуктами амплификации областей соединения на правом конце MREJ типа xxi, которые содержат детектируемые молекулы, обнаруживаемые с помощью устройства BD MAX™ (Becton Dickinson), SMARTCYCLER® (Cepheid) или другого устройства, разработанного для ПЦР в реальном времени, в каналах FAM, техасского красного и Tet, т.е. SEQ ID NO: 4.
[0109] ПЦР осуществляли на BD MAX™ (Becton Dickinson) или SMARTCYCLER® (Cepheid), применяя одинаковые параметры циклов, описанные далее:
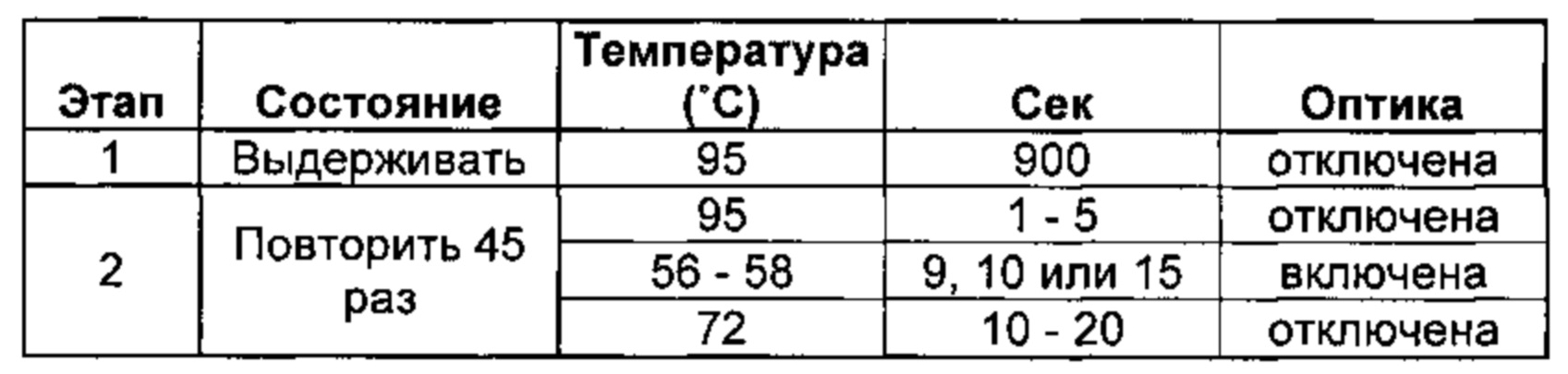
[0110] Пороговый цикл (ПЦ) в каналах FAM, техасского красного и ТЕТ определяли, применяя программное обеспечение BD МАХ™ или SMARTCYCLER®. ПЦ применяли для определения того, присутствует ли MRSA с MREJ любого из типов i-vii, xvi, ix, xiii, xiv и xxi.
ПРИМЕР 3: ПОСЛЕДОВАТЕЛЬНОСТИ MREJ ТИПА XXI АССОЦИИРОВАНЫ С ГОМОЛОГОМ МЕСА, МЕСС
[0111] Осуществляли амплификацию путем ПЦР области MREJ типа xxi и гена mecC из 51 изолята MRSА, полученных от быка или человека.
[0112] Перечень изолятов приведен в таблице ниже:
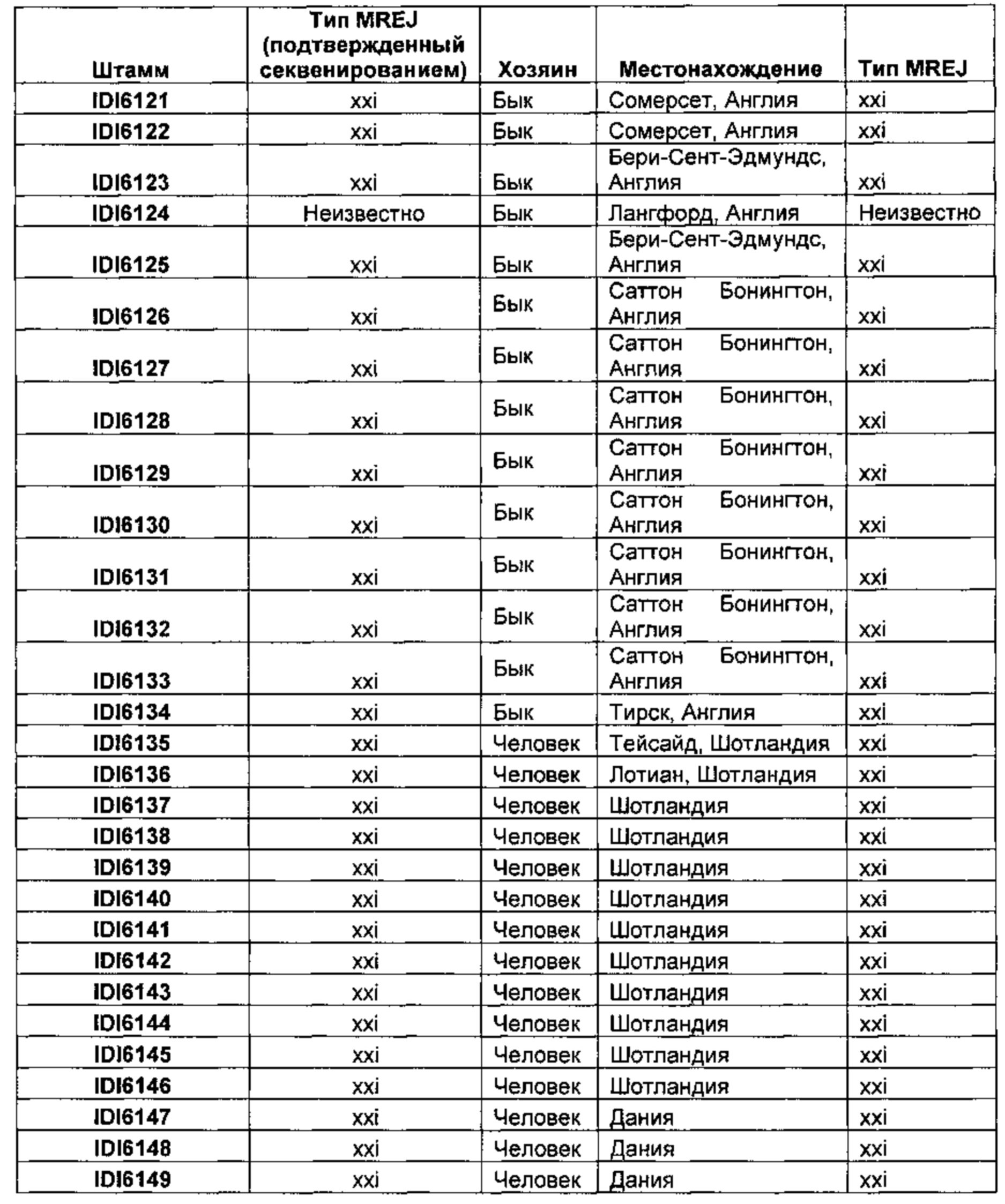
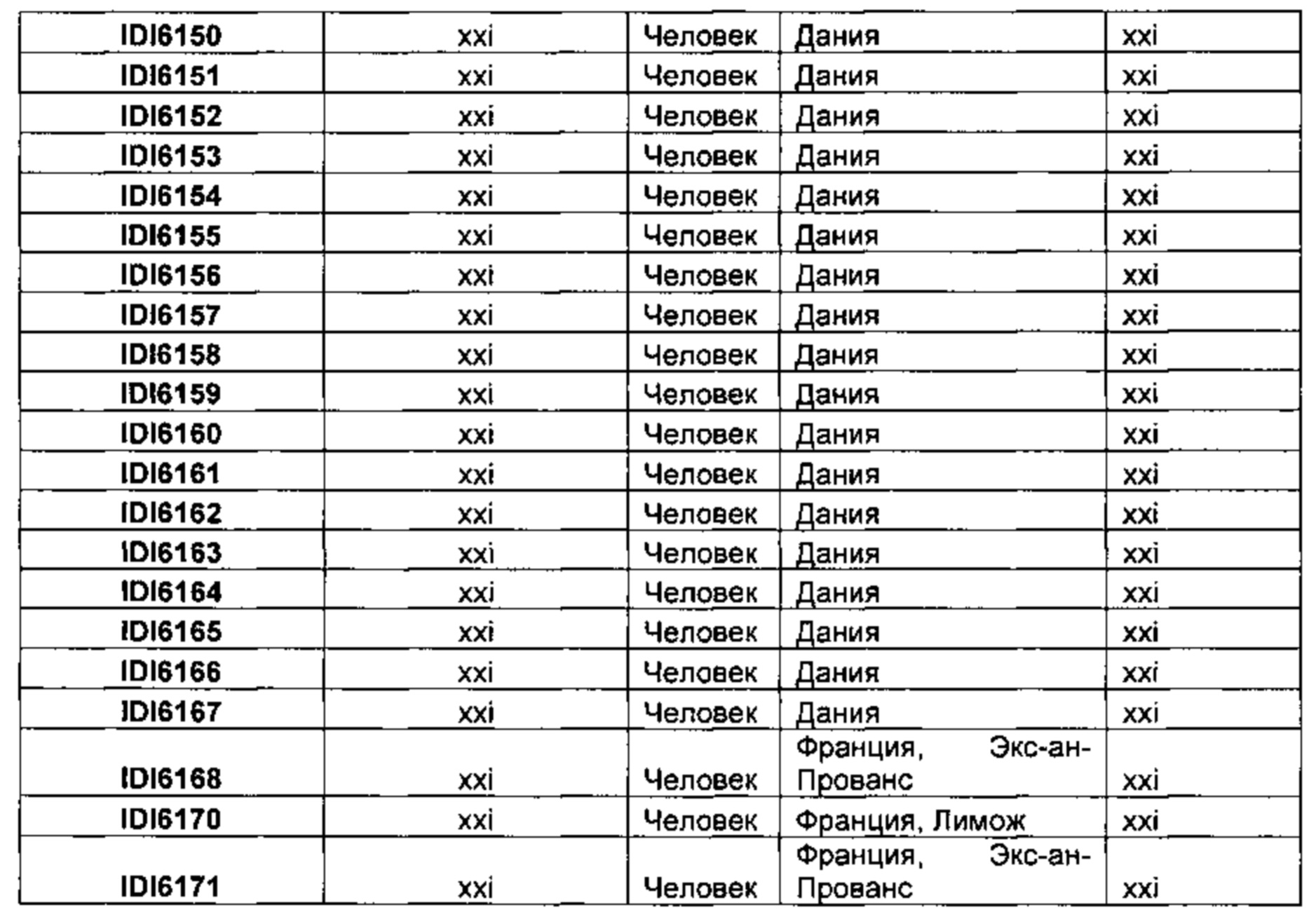
[0113] ДНК выделяли из описанных выше клинических образцов, про которые известно, что они несут mecC, применяя набор BD GeneOhm™ Lysis (Becton Dickinson), следуя инструкциям производителя. Состав реакционных смесей для ПЦР описан далее: по 0,2-0,7 мкМ каждой из последовательностей SEQ ID NO: 182, 183 и 187, 0,3 мкМ дНТФ (Roche), 4 мМ MgCl2 (SIGMA), 2,8 единиц Taq-полимеразы FASTSTART® (Roche), 100 мМ трис, pH 8,3 (EMD), 10 мМ KCl (LaboratoireMat), 5 мМ (NH4)2SO4 (SIGMA), 0,15 мг/мл БСА (SIGMA), 4% трегалозы (SIGMA).
[0114] Реакции осуществляли на системе BD МАХ®, используя канал для FAM.
[0115] Полученные результаты свидетельствуют о том, что 50 из 51 штамма MRSA, которые несли ген mecC, содержали последовательность MREJ типа xxi, таким образом, обеспечивая доказательства высокой специфичности обнаружения штаммов MRSA с mecC с применением последовательностей MREJ типа xxi.
[0116] После описания настоящего изобретения в общих чертах, для дополнительного понимания мы предложили ссылки на некоторые конкретные примеры, которые предусмотрены в данной заявке исключительно с целью иллюстрирования и не предполагаются ограничивающими настоящее изобретение.
--->
ПЕРЕЧЕНЬ ПОСЛЕДОВАТЕЛЬНОСТЕЙ
<110> GENEOHM SCIENCES CANADA, INC.
Menard, Christian
Roger-Dalbert, Celine
<120> ПОСЛЕДОВАТЕЛЬНОСТИ ДЛЯ ОБНАРУЖЕНИЯ И РАСПОЗНАВАНИЯ
УСТОЙЧИВОГО К МЕТИЦИЛЛИНУ ЗОЛОТИСТОГО СТАФИЛОКОККА
(STAPHYLOCOCCUS AUREUS) (MRSA) С MREJ ТИПА XXI
<130> GENOM.114WO
<150> 61/621368
<151> 2012-04-06
<160> 187
<170> FastSEQ для Windows версии 4.0
<210> 1
<211> 772
<212> ДНК
<213> Staphylococcus aureus
<220>
<223> n= c, a, t или g
<220>
<221> Прочее
<222> 4, 6
<223> n = A,T,C или G
<400> 1
tcangnggga tcaaacggcc tgcacaagga cgtcttacaa cgcagtaact acgcactatc 60
attcagcaaa atgacatttc cacatcaaat gatgcgggtt gtgttaattg agcaagtgta 120
tagagcgttt aagattatgc gcggagaagc gtatcacaaa tgatgcggtt tttttaacct 180
ctttacgtat gaggtttatg agaattgccg ttatgttttg cgagagttat caatcttttt 240
gatagtaaga aagtacatag aaactaaaag agtattttta tctacaatag catttataat 300
ttattctatt attgtatact tattttaatt attagtatca ttgctgagat gttacttgat 360
attctatgtc tattttttag gaaattctat actattaaaa ttatggtatt ttatacgcaa 420
taaaggacta atctatttta tacagattag tcctttattg tagtctttaa aaactagtta 480
ctcattaata ttttttagta caatttcagc aacctcactt actattttgt cattaggttt 540
accatctttt ctatctttat ttgtaaatat caccagaatt ataggtttat cttgtccatc 600
tggataaaca aagcgaacat cgtttcttga accgtatgtt agtgcttgac cgctcttatc 660
cataacttta aagtttgaag gtgcaccatc cttaattaat gtatcgccac ttttattttt 720
gaacattaga ttaagtaaga aatctttgtt tgctttgcta agatctccat ca 772
<210> 2
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 2
cggcaattct cataaacctc a 21
<210> 3
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 3
ggatcaaacg gcctgcaca 19
<210> 4
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 4
tagttactgc gttgtaagac gtcc 24
<210> 5
<211> 3050
<212> ДНК
<213> Staphylococcus aureus
<220>
<223> синтетический олигонуклеотид
<400> 5
tcgtgccatt gatgcagagg gacatacatt agatatttgg ttgcgtaagc aacgagataa 60
tcattcagca tatgcgttta tcaaacgtct cattaaacaa tttggtaaac ctcaaaaggt 120
aattacagat caggcacctt caacgaaggt agcaatggct aaagtaatta aagcttttaa 180
acttaaacct gactgtcatt gtacatcgaa atatctgaat aacctcattg agcaagatca 240
ccgtcatatt aaagtaagaa agacaaggta tcaaagtatc aatacagcaa agaatacttt 300
aaaaggtatt gaatgtattt acgctctata taaaaagaac cgcaggtctc ttcagatcta 360
cggattttcg ccatgccacg aaattagcat catgctagca agttaagcga acactgacat 420
gataaattag tggttagcta tattttttta ctttgcaaca gaaccgaaaa taatctcttc 480
aatttatttt tatatgaatc ctgtgactca atgattgtaa tatctaaaga tttcagttca 540
tcatagacaa tgttcttttc aacatttttt atagcaaatt gattaaataa attctctaat 600
ttctcccgtt tgatttcact accatagatt atattatcat tgatatagtc aatgaataat 660
gacaaattat cactcataac agtcccaacc cctttatttt gatagactaa ttatcttcat 720
cattgtaaaa caaattacac cctttaaatt taactcaact taaatatcga caaattaaaa 780
aacaataaaa ttacttgaat attattcata atatattaac aactttatta tactgctctt 840
tatatataaa atcattaata attaaacaag ccttaaaata tttaactttt ttgtgattat 900
tacacattat cttatctgct ctttatcacc ataaaaatag aaaaaacaag attcctaaag 960
aatataggaa tcttgtttca gactgtggac aaactgattt tttatcagtt agcttattta 1020
gaaagtttta tttaaattac agtttctatt tttattagat cacaatttta ttttagctct 1080
tgttcaagta atcatttttc gccaaaaact ttatactgaa tagcttctac attaaatact 1140
ttgtcaatga gatcatctac atctttaaat tcagaataat ttgcatatgg atctataaaa 1200
taaaattgtg gttctttacc ggaaacatta aatattctta atattaaata tttctgctta 1260
tattctttca tagcaaacat ttcatttagc gacataaaaa atggttcctc aatactagaa 1320
gatgtagatg ttttaatttc aataaatttt tctacagctt tatctgtatt tgttggatca 1380
aaagctacta aatcatagcc atgaccgtgt tgagagcctg gattatcatt taaaatattc 1440
ctaaactgtt ctttcttatc ttcgtctatt ttattatcaa ttagctcatt aaagtaattt 1500
agcgctaatt tttctccaac tttaccggtt aatttattct ctttatttga tttttcaatt 1560
tctgaatcat ttttagtagt ctttgataca ccttttttat attttggaat tattccttta 1620
ggtgcttcca cttccttgag tgtcttatct ttttgtgctg ttctaatttc ttcaatttcg 1680
ctgtcttcct gtatttcgtc tatgctattg accaagctat cataggatgt ttttgtaact 1740
tttgaagcta attcattaaa tagttctaaa aatttcttta aatcctctag catatcttct 1800
tctgtgaatc cttcattcaa atcataatat ttgaatctta ttgatccatg agaatatcct 1860
gatggataat cattttttaa atcataagat gaatctttat tttctgcgta ataaaatctt 1920
ccagtattaa attcatttga tgtaatatat ttattgagtt cggaagataa agttaatgct 1980
ctttgttttg cagcattttt atcccgcgga aacatatcac ttatctttga ccatccttga 2040
ttcaaagata agtatatgcc ttctccttcc ggatgaaaaa gatataccaa ataatatcca 2100
tcctttgttt cttttgttat attctcatca tatattgaaa tccaaggaac tttactatag 2160
ttcccagtag caaccttccc tacaactgaa tatttatctt cttttatatg cacttttaac 2220
tgcttgggta acttatcatg gactaaagtt ttatatagat cacctttatc ccaatcagat 2280
tttttaacta cattattggt acgtttctct ttaattaatt taaggacctg cataaagttg 2340
tctatcattt gaaattccct cctattataa aatatattat gtctcatttt cttcaatatg 2400
tacttattta tattttaccg taatttacta tatttagttg cagaaagaat tttctcaaag 2460
ctagaacttt gcttcactat aagtattcag tataaagaat atttcgctat tatttacttg 2520
aaatgaaaga ctgcggaggc taactatgtc aaaaatcatg aacctcatta cttatgataa 2580
gcttctcctc gcataatctt aaatgctctg tacacttgtt caattaacac aacccgcatc 2640
atttgatgtg ggaatgtcat tttgctgaat gatagtgcgt agttactgcg ttgtaagacg 2700
tccttgtgca ggccgtttga tccgccaatg acgaaaacaa agtcgctttg cccttgggtc 2760
atgcgttggt tcaattcttg ggccaatcct tcggaagata gcatctttcc ttgtatttct 2820
aatgtaatga ctgtggattg tggtttgatt ttggctagta ttcgttggcc ttctttttct 2880
tttacttgct caatttcttt gtcactcata ttttctggtg ctttttcgtc tggaacttct 2940
atgatgtcta tcttggtgta tgggcctaaa cgtttttcat attctgctat ggcttgcttc 3000
caatatttct cttttagttt ccctacagct aaaatggtga ttttcatgtc 3050
<210> 6
<211> 3050
<212> ДНК
<213> Staphylococcus aureus
<400> 6
acctcattga gcaagatcac cgtcatatta aagtaagaaa gacaaggtat caaagtatca 60
atacagcaaa gaatacttta aaaggtattg aatgtattta cgctctatat aaaaagaacc 120
gcaggtctct tcagatctac ggattttcgc catgccacga aattagcatc atgctagcaa 180
gttaagcgaa cactgacatg ataaattagt ggttagctat atttttttac tttgcaacag 240
aaccgaaaat aatctcttca atttattttt atatgaatcc tgtgactcaa tgattgtaat 300
atctaaagat ttcagttcat catagacaat gttcttttca acatttttta tagcaaattg 360
attaaataaa ttctctaatt tctcccgttt gatttcacta ccatagatta tattatcatt 420
gatatagtca atgaataatg acaaattatc actcataaca gtcccaaccc ctttcttttg 480
atagactaat tatcttcatc attgtaaaac aaattacacc ctttaaattt aactcaactt 540
aaatatcgac aaattaaaaa acaataaaat tacttgaata ttattcataa tatattaaca 600
actttattat actgctcttt atatataaaa tcattaataa ttaaacaagc cttaaaatat 660
ttaacttttt tgtgattatt acacattatc ttatctgctc tttatcacca taaaaataga 720
aaaaacaaga ttcctaaaga atataggaat cttgtttcag actgtggaca aactgatttt 780
ttatcagtta gcttatttag aaagttttat ttaaattaca gtttctattt ttattagatc 840
acaattttat tttagctctt gttcaagtaa tcatttttcg ccaaaaactt tatactgaat 900
agcttctaca ttaaatactt tgtcaatgag atcatctaca tctttaaatt cagaataatt 960
tgcatatgga tctataaaat aaaattgtgg ttctttaccg gaaacattaa atattcttaa 1020
tattaaatat ttctgcttat attctttcat agcaaacatt tcatttagcg acataaaaaa 1080
tggttcctca atactagaag atgtagatgt tttaatttca ataaattttt ctacagcttt 1140
atctgtattt gttggatcaa aagctactaa atcatagcca tgaccgtgtt gagagcctgg 1200
attatcattt aaaatattcc taaactgttc tttcttatct tcgtctattt tattatcaat 1260
tagctcatta aagtaattta gcgctaattt ttctccaact ttaccggtta atttattctc 1320
tttatttgat ttttcaattt ctgaatcatt tttagtagtc tttgatacac cttttttata 1380
ttttggaatt attcctttag gtgcttccac ttccttgagt gtcttatctt tttgtgctgt 1440
tctaatttct tcaatttcgc tgtcttcctg tatttcgtct atgctattga ccaagctatc 1500
ataggatgtt tttgtaactt ttgaagctaa ttcattaaat agttctaaaa atttctttaa 1560
atcctctagc atatcttctt ctgtgaatcc ttcattcaaa tcataatatt tgaatcttat 1620
tgatccatga gaatatcctg atggataatc attttttaaa tcataagatg aatctttatt 1680
ttctgcgtaa taaaatcttc cagtattaaa ttcatttgat gtaatatatt tattgagttc 1740
ggaagataaa gttaatgctc tttgttttgc agcattttta tcccgcggaa acatatcact 1800
tatctttgac catccttgat tcaaagataa gtatatgcct tctccttccg gatgaaaaag 1860
atataccaaa taatatccat cctttgtttc ttttgttata ttctcatcat atattgaaat 1920
ccaaggaact ttactatagt tcccagtagc aaccttccct acaactgaat atttatcttc 1980
ttttatatgc acttttaact gcttgggtaa cttatcatgg actaaagttt tatatagatc 2040
acctttatcc caatcagatt ttttaactac attattggta cgtttctctt taattaattt 2100
aaggacctgc ataaagttgt ctatcatttg aaattccctc ctattataaa atatattatg 2160
tctcattttc ttcaatatgt acttatttat attttaccgt aatttactat atttagttgc 2220
agaaagaatt ttctcaaagc tagaactttg cttcactata agtattcagt ataaagaata 2280
tttcgctatt atttacttga aatgaaagac tgcggaggct aactatgtca aaaatcatga 2340
acctcattac ttatgataag cttcttaaaa acataacagc aattcacata aacctcatat 2400
gttctgatac attcaaaatc cctttatgaa gcggctgaaa aaaccgcatc atttatgata 2460
tgcttctcca cgcataatct taaatgctct atacacttgc tcaattaaca caacccgcat 2520
catttgatgt gggaatgtca ttttgctgaa tgatagtgcg tagttactgc gttgtaagac 2580
gtccttgtgc aggccgtttg atccgccaat gacgaataca aagtcgcttt gcccttgggt 2640
catgcgttgg ttcaattctt gggccaatcc ttcggaagat agcatctttc cttgtatttc 2700
taatgtaatg actgtggatt gtggtttaat tttggctagt attcgttggc cttctttttc 2760
ttttacttgc tcaatttctt tgtcgctcat attttctggt gctttttcgt ctggaacttc 2820
tatgatgtct atcttggtgt atgggcctaa acgtttttca tattctgcta tggcttgctt 2880
ccaatatttc tcttttagtt tccctacagc taaaatggtg attttcatgt cgtttggtcc 2940
tccaaattgt tatcaacttt ccagttatcc acaagttatt aacttgttca cactgttccc 3000
tcttattata ccaatatttt ttgcagtttt tgatattttc ctgacattta 3050
<210> 7
<211> 1256
<212> ДНК
<213> Staphylococcus aureus
<400> 7
ttcagaaaaa tgattaatgt gtttcaataa aatctctcct tctttgtgaa catattcatt 60
tttatactaa ttaatataat ttccaaaaaa gtttctgttt aaaagtgaaa aatattattt 120
accgtttgac ttaaatcttc aatatatagg tgtttatatg tatcattttg cgccaatttg 180
aataaacggg aatcaagtct gtttctgagt ttatttcaac tttcttatag taaacattgt 240
cttaatatga tgaacttcaa taaaactttc cctatgcccc ataaaatttt ctcaaaatca 300
aaaataacat accttacaac ttttaccgtc gatatcaatt gctcttttct taatttagga 360
ttgctttcaa attttgtact ataacgtgaa actacttttc cttctttata attaaaattt 420
actaattcac aatcattttt acttccattt acaaaaacat ccactgtttc taacacaaaa 480
tctaataaac ttccttttat taatcgtagg cattgtatat ttcctttcat tctttcttga 540
ttccattagt ttaaatttaa aatttcatcc atcaatttct taatttaatt gtagttccat 600
aatcaatata atttgtacag ttattatata ttctagatca tcaatagttg aaaaatggtt 660
tattaaacac tctataaaca tcgtatgata ttgcaaggta taatccaata tttcatatat 720
gtaattcctc cacatctcat taaattttta aattatacac aacctaattt ttagttttat 780
ttatgatacg cttctccacg cataatctta aatgctctgt acacttgttc aattaacaca 840
acccgcatca tttgatgtgg gaatgtcatt ttgctgaatg atagtgcgta gttactgcgt 900
tgtaagacgt ccttgtgcag gccgtttgat ccgccaatga cgaatacaaa gtcgctttgc 960
ccttgggtca tgcgttggtt caattcttgg gccaatcctt cggaagatag catctttcct 1020
tgtatttcta atgtaatgac tgttgattgt ggtttgattt tggctagtat tcgttggcct 1080
tctttttctt ttacttgctc aatttctttg tcgctcatat tttctggtgc tttttcgtct 1140
ggaacttcta tgatgtctat cttggtgtat gggcctaaac gtttttcata ttctgctatg 1200
gcttgcttcc aatatttctc ttttagtttc cctacagcta aaatggtgat tttcat 1256
<210> 8
<211> 2122
<212> ДНК
<213> Staphylococcus aureus
<400> 8
ggaaactaaa agagaaatat tggaagcaag ccatagcaga atatgaaaaa cgtttaggcc 60
catacaccaa gatagacatc atagaagttc cagacgaaaa agcaccagaa aatatgagcg 120
acaaagaaat tgagcaagta aaagaaaaag aaggccaacg aatactagcc aaaatcaaac 180
cacaatcaac agtcattaca ttagaaatac aaggaaagat gctatcttcc gaaggattgg 240
cccaagaatt gaaccaacgc atgacccaag ggcaaagcga ctttgtattc gtcattggcg 300
gatcaaacgg cctgcacaag gacgtcttac aacgcagtaa ctacgcacta tcattcagca 360
aaatgacatt cccacatcaa atgatgcggg ttgtgttaat tgaacaagtg tacagagcat 420
ttaagattat gcgtggagaa gcgtaccaca aatgatgcgg ttttttatcc agttttttgt 480
ttaatgaaca aggtaaatta cgagataata tttgaagaaa acaataaagt agagatggat 540
ttccatatcc tctttagtag cggtttttat ctgtaaggtt tattaataat taaataaata 600
ggcgggatag ttatatatag cttattaatg aaagaatatg attattaatt tagtattata 660
ttttaatatt aaaaagaaga tatgaaataa ttattcatac cttccacctt acaataatta 720
gttttcaatc gaatattaag attattagta gtcttaaaag ttaagacttc cttatattaa 780
tgacctaatt tattatttgc ctcatgaatt atctttttat ttctttgata tgtcccaaac 840
cacatcgtga tatacactac aataaatatt atgatgaaac taataatatt ctcaaagttc 900
agatggaacc aacctgctag aatagcgagt gggaagaata ggattatcat caatataaag 960
tgaactacag tctgttttgt tatactccaa tcggtatctg taaatatcaa attaccataa 1020
gtaaacaaaa ttccaatcaa tgcccatagt gctacacata ttagcataat aaccgcttca 1080
ttaaagtttt cataataaat tttacccata aaagaatctg gatatagtgg tacatattta 1140
tcccttgaaa aaaataagtg aagtaatgac agaaatcata agaccagtga acgcaccttt 1200
ttgaacagcg tggaataatt ttttcatagt gagatggacc attccatttg tttctaactt 1260
caagtgatca atgtaattta gattgataat ttctgatttt gaaatacgca cgaatattga 1320
accgacaagc tcttcaattt ggtaaagtcg ctgataaagt tttaaagctt tattattcat 1380
tgttatcgca tacctgttta tcttctacta tgaactgtgc aatttgttct agatcaattg 1440
ggtaaacatg atggttctgt tgcaaagtaa aaaaatatag ctaaccacta atttatcatg 1500
tcagtgttcg cttaacttgc tagcatgatg ctaatttcgt ggcatggcga aaatccgtag 1560
atctgatgag acctgcggtt ctttttatat agagcgtaaa tacattcaat accttttaaa 1620
gtattctttg ctgtattgat actttgatac cttgtctttc ttactttaat atgacggtga 1680
tcttgctcaa tgaggttatt cagatatttc gatgtacaat gacagtcagg tttaagttta 1740
aaagctttaa ttactttagc cattgctacc ttcgttgaag gtgcctgatc tgtaattacc 1800
ttttgaggtt taccaaattg tttaatgaga cgtttgataa acgcatatgc tgaatgatta 1860
tctcgttgct tacgcaacca aatatctaat gtatgtccct ctgcatcaat ggcacgatat 1920
aaatagctcc attttccttt tattttgatg tacgtctcat caatacgcca tttgtaataa 1980
gcttttttat gctttttctt ccaaatttga tacaaaattg gggcatattc ttgaacccaa 2040
cggtagaccg ttgaatgatg aacgtttaca ccacgttccc ttaatatttc agatatatca 2100
cgataactca atgtatatct ta 2122
<210> 9
<211> 1696
<212> ДНК
<213> Staphylococcus aureus
<400> 9
aaagagaaat attggaagca agccatagca gaatatgaaa aacgtttagg cccatacacc 60
aagatagaca tcatagaagt tccagacgaa aaagcaccag aaaatatgag tgacaaagaa 120
attgagcaag taaaagaaaa agaaggccaa cgaatactag ccaaaatcaa accacaatcc 180
acagtcatta cattagaaat acaaggaaag atgctatctt ccgaaggatt ggcccaagaa 240
ttgaaccaac gcatgaccca agggcaaagc gactttgttt tcgtcattgg cggatcaaac 300
ggcctgcaca aggacgtctt acaacgcagt aactacgcac tatcattcag caaaatgaca 360
ttcccacatc aaatgatgcg ggttgtgtta attgaacaag tgtacagagc atttaagatt 420
atgcgaggag aagcatatca taaatgatgc ggttatttca gccgtaattt tataatataa 480
agcagagttt attaaatttt aatgattact ttttattaag aattaattct agttgatata 540
ttataatgtg aaacacaaaa taataatttg taattgttag tttataggca tctgtatttg 600
gaattttttg tagactattt aaaaaatagt gtatataagt attgagttca tgtattaact 660
gtcttttttc atcgttcatc aagtataagg atgtagagat ttgttggata atttcttcgg 720
atgtttttaa aattatcatt aaattagatg gtatctgatc ttgagttttg tttttagtgt 780
atgtatattt taaaaaattt ttgattgttg ttatttgact ctcttttaat ttgacaccct 840
catcaataaa tgtgttaaat atatcttcat ttgtacttaa atcatcaaaa tttgccaaca 900
aatatttgaa cgtctctaaa tcattatgtt tgagttccgt tttgctattc cataattcca 960
aaccatttgg tagaaagccc aagctgtgat tttgatctcc ccatatagct gaatttaaat 1020
cagtgagttg attaattttt tcaacacaga aatgtaattt tggaatgagg aatcgaagtt 1080
gttcttctac ttgctgtact tttcttttgt tttcaataaa atttctacac catactgtta 1140
tcaaaccgcc aattattgtg cacaatcctc caatgattgt agataaaatt gacaatatat 1200
tacacacctt tcttagaggt ttattaacat ctatttttga atttaaaatt attactttgg 1260
tagcgttata acctatttaa cagattagag aaaaattgaa tgatcgattg aagaatttcc 1320
aaaataccgt cccatatgcg ttgaaggaga tttctatttt cttctgtatt caaatctttg 1380
gctttatcct ttgctttatt caataaatca tctgagtttt tttcaatatt ttttaataca 1440
tctttggcat tttgtttaaa tactttagga tcggaagtta gggcattaga gtttgccaca 1500
ttaatcatat tattattaat catttgaatt tgattatctg ataatatctc tgataaccta 1560
cgctcatcga ggactttatt aacagtgtct tcaacttgtt gttgtgtgat ttgtttatct 1620
tgattttgtt taatatctgc aagttgttct ttaatatctg ctatagaagc atttaaagct 1680
tcatctgaat acccat 1696
<210> 10
<211> 991
<212> ДНК
<213> Staphylococcus aureus
<400> 10
accattttag ctgtagggaa actaaaagag aaatactgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgaactacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atcaacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcga ggagaagcgt atcataagtg atggtaaaaa 480
atatgagtaa gtagatgaag agtgaaaatc agattaatta ataataatgt atcaaattta 540
aataaagggg tttttaagta tgaatttaag aggtcatgaa aatagactta aatttcatgc 600
gaaatatgat gtgacaccta tatcacattt aaaattatta gaaggtcaaa agaaagacgg 660
tgaaggcggc atactgacag atagctatta ctgtttttca tacagcttaa aaggtaattc 720
taaaaaagtt ttaggtacgt ttaattgtgg ttatcatatt gctgaagatt tactaaaatt 780
atcaaatcaa gataaattac ctttatttaa cccgtttaaa gtaattaatg aaggtaatca 840
attgcagggc gtaacgaata aaggtaattt aaatattaat aggcaaagaa aacagtataa 900
tgaagtggct ttacagcttt caaatgctat taatttaatc ataatttgtt atgaggataa 960
tattaaagaa ccactttcaa cgataaaata c 991
<210> 11
<211> 1282
<212> ДНК
<213> Staphylococcus aureus
<400> 11
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactat 360
gcactatcat ttagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtata gagcatttaa gattatgcgt ggagaagcgt accacaaata aaactaaaaa 480
atatgagaaa attattaaat tagctcaaat ctttgaagaa taaaaagtga atattaagtt 540
tgataattta ggtacaagta aagattaaga atttccatta tttaatacat ggtgtgtaaa 600
tcgacttctt tttgtattag atgtttgcag taagcgatgt aaagaagatg ctaataaata 660
tgtgaggaat gattacgata ctagataagc ggctaatgaa attttttaaa gtacatatat 720
agacatattt ttcatttagt aaaattttga atttcacttt gctaagacta gtgtctagaa 780
atttataatg atttattaac acctatttga aacttaagta taataaatga ttcggatttt 840
atttttaata aagacaaact tgaacgtagc aaagtagttt ttatgataaa taataagttt 900
taataatgtg acgcttttat ataagcacat tattatgaac aatgtgaatt gagcatctac 960
aattacatta ataaatatat aaatgatgat ttaaattcac atatatttat aatacacata 1020
ctatatgaaa gttttgatta tccgaataaa tgctaaaatt aataaaataa ttaaaggaat 1080
catacttatt atacgtatac gtttagctac tgaactactg gattcatttg gagattctag 1140
tagttctttt tcaatctcta aatctaaatc agttttgtaa taaccattaa ttcctaatct 1200
ttcatctagc tctgtacttt tttcatcatt tttatctttg ttgatatgtt ccattttctc 1260
gcctcttttt aatcaagtag aa 1282
<210> 12
<211> 1530
<212> ДНК
<213> Staphylococcus aureus
<400> 12
ttagctgtag ggaaactaaa agagaaatat tggaagcaag ccatagcaga atatgaaaaa 60
cgtttaggcc catacaccaa gatagacatc atagaagttc cagacgaaaa agcaccagaa 120
aatatgagcg acaaagaaat tgagcaagta aaagaaaaag aaggccaacg aatactagcc 180
aaaatcaaac cacaatccac agtcattaca ttagaaatac aaggaaagat gctatcttcc 240
gaaggattgg cccaagaatt gaaccaacgc atgacccaag ggcaaagcga ctttgtattc 300
gtcattggcg gatcaaacgg cctgcacaag gacgtcttac aacgcagtaa ctatgcacta 360
tcatttagca aaatgacatt cccacatcaa atgatgcggg ttgtgttaat tgaacaagtg 420
tatagagcat ttaagattat gcgtggagaa gcatatcata aatgatgcgg ttttttcagc 480
cgcttcataa aggggggtga tcatatcgga acgtatgagg tttatgagaa ttgctgctat 540
gtttttatga agcgtatcat aaatgatgca gtttttgata attttttctt tatcagagat 600
tttactaaaa atcccctcaa agtttgtttt tttcaacttc aactttgaag ggaataaata 660
aggaacttat ttatatttat cctttatctc attaatatct atttttttat taataatatt 720
ataaatatta aattctttag aaaagtcact atcactctta ttcttcatac taaacgttat 780
taatctaata atatcagcta ctatttcttt aaattctatt gcatcttctt ttttataagt 840
agcgcctgta tgaacaattt tatttctcat accatagtaa tctttcatat atttttttac 900
acaattttta atttcattag aattatccaa atctagatta tcaattgtct ttaataaatg 960
atcattaaca acattagcat acccacatcc aagcttcttt tttatctctt catcacttaa 1020
attttcatct aatttataat atctttctaa aaaatttgtg ataaaaactt ctaatgcagt 1080
ctgaatttgt acaattgcta aattatagtc agatttataa aaagaacgtt caccttttct 1140
catagccaaa acataaatat tgctaggatg attattgaaa atattataat tttttttaat 1200
atttaataaa tcactttttt tgatagatga atactgatct tcttctatct ttccaggcat 1260
gtcaatcatg aaaatactca tctcttttat atttccatct atagtatata ttatataata 1320
tggaatactt aatatatccc ctaatgatag ctggtatata ttatgatact gatatttaac 1380
gctaataatt ttaataagat tatttagaca attaaattgc ttattaaaaa ttttcgttag 1440
actattactt ttctttgatt ccctagaagt agaatttgat ttcaattttt taaactgatt 1500
gtgcttgatt attgaagtta tttcaacata 1530
<210> 13
<211> 1256
<212> ДНК
<213> Staphylococcus aureus
<400> 13
gctgtaggga aactaaaaga gaaatattgg aagcaagcca tagcagaata tgaaaaacgt 60
ttaggcccat acaccaagat agacatcata gaagttccag acgaaaaagc accagaaaat 120
atgagcgaca aagaaattga gcaagtaaaa gaaaaagaag gccaacgaat actagccaaa 180
attaaaccac aatccacagt cattacatta gaaatacaag gaaagatgct atcttccgaa 240
ggattggccc aagaattgaa ccaacgcatg acccaagggc aaagcgactt tgtattcgtc 300
attggcggat caaacggcct gcacaaggac gtcttacaac gcagtaacta cgcactatca 360
ttcagcaaaa tgacattccc acatcaaatg atgcgggttg tgttaattga gcaagtgtat 420
agagcattta agattatgcg tggagaagca tatcataaat gatgcggttt tttcagccgc 480
ttcataaagg gattttgaat gtatcagaac atatgaggtt tatgtgaatt gctgttatgt 540
ttttaagaag catatcataa gtgatgcggt ttttattaat tagttgctaa aaaatgaagt 600
atgcaatatt aattattatt aaattttgat atatttaaag aaagattaag tttagggtga 660
atgaatggct tatcaaagtg aatatgcatt agaaaatgaa gtacttcaac aacttgagga 720
attgaactat gaaagagtaa atatacataa tattaaatta gaaattaatg aatatctcaa 780
agaactagga gtgttgaaaa atgaataagc agacaaatac tccagaacta agatttccag 840
agtttgatga ggaatggaaa aaaaggaaat taggtgaagt agtaaattat aaaaatggtg 900
gttcatttga aagtttagtg aaaaaccatg gtgtatataa actcataact cttaaatctg 960
ttaatacaga aggaaagttg tgtaattctg gaaaatatat cgatgataaa tgtgttgaaa 1020
cattgtgtaa tgatacttta gtaatgatac tgagcgagca agcaccagga ctagttggaa 1080
tgactgcaat tatacctaat aataatgagt atgtactaaa tcaacgagta gcagcactag 1140
tgcctaaaca atttatagat agtcaatttc tatctaagtt aattaataga aaccagaaat 1200
atttcagtgt gagatctgct ggaacaaaag tgaaaaatat ttctaaagga catgta 1256
<210> 14
<211> 748
<212> ДНК
<213> Staphylococcus aureus
<400> 14
atcgtttaac gtgtcacatg atgcgataga tccgcaattt tatattttcc ataataacta 60
taagaagttt acgattttaa cagatacggg ttacgtgtct gatcgtatga aaggtatgat 120
acgtggcagc gatgcattta tttttgagag taatcatgac gtcgatatgt tgagaatgtg 180
tcgttatcca tggaagacga aacaacgcat tttaggcgat atgggtcatg tatctaatga 240
ggatgcgggt catgcgatga cagacgtgat tacaggtaac acgaaacgta tttacttatc 300
gcatttatca caagataata atatgaaaga tttggcgcgt atgagtgttg gccaagtatt 360
gaacgaacac gatattgata cggaaaaaga agtattgcta tgtgatacgg ataaagctat 420
tccaacacca atatatacaa tataaatgag agtcatccga taaagttccg cactgctgtg 480
aaacgacttt atcgggtgct tttttatgtt gttggtggga aatggctgtt gttgagttga 540
atcggattga ttgaaatgtg taaaataatt cgatattaaa tgtaatttat aaataattta 600
cataaaatca aacattttaa tataaggatt atgataatat attggtgtat gacagttaat 660
ggagggaacg aaatgaaagc tttattactt aaaacaagtg tatggctcgt tttgcttttt 720
agtgtgatgg gattatggca tgtctcga 748
<210> 15
<211> 1100
<212> ДНК
<213> Staphylococcus aureus
<400> 15
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa ttaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgag 420
caagtgtata gagcatttaa gattatgcgt ggagaagcgt atcacaaata aaactaaaaa 480
ataagttgta tataacttat tttgaaattg gttaagtata taatatctcc aataaaatgt 540
agttaactta cgataatgct gaactatagc tttgtaaact aaaatgtaaa taattacaat 600
caaattgcaa caatatagtt caagaatgct acaatttgag gacagattga tagcattaat 660
ccctttaaaa tgaagctagg agataactta cattatgatt agtaaacaaa taaaggattt 720
acgaaagcaa cataattata ctcaagaaga gctagctgaa aaattaaata cttcaagaca 780
aacaatttct aaatgggaac aaggtatttc agaaccagac ttaattatgc ttatgcaatt 840
gtcacaatta ttttctgtta gtacagacta tctcattaca ggaagtgaca atattattaa 900
aaaagataat aaaagctatt atgaaatgaa tttttgggca tttatgtctg aaaaatggtg 960
ggtaattatt attatagtaa tcataatttg tggaacaata ggacaaattt tttcaaacta 1020
atgtaagtat ctctcaaata ttttgggagg ttttattatg aaaatcaaaa aattattaaa 1080
gacattatta attattttat 1100
<210> 16
<211> 1109
<212> ДНК
<213> Staphylococcus aureus
<400> 16
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atcaacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcgt ggagaagcgt atcacaaata aaactaaaaa 480
ataagttgta tataacttat tttgaaattg gttaagtata tagtatctcc aataaaatgt 540
agttaactta cgataatgct gaactatagc tttgtaaact aaaatgtaaa taattacaat 600
caaattgcaa caatatagtt caagaatgct acaatttgag gacagattga tagcattaat 660
ccctttaaaa tgaagctagg agataactta cattatgatt agtaaacaaa taaaggattt 720
acgaaagcaa cataattata ctcaagaaga gctagctgaa aaattaaata cttcaagaca 780
aacaatttct aaatgggaac aaggtatttc agaaccagac ttaattatgc ttatgcaatt 840
gtcacaatta ttttctgtta gtacagacta tctcattaca ggaagtgaca atattattaa 900
aaaagataat aaaagctatt atgaaatgaa tttttgggca tttatgtctg aaaaatggtg 960
ggtaattatt attatagtaa tcataatttg tggaacaata ggacaaattt tttcaaacta 1020
atgtaagtat ctctcaaata ttttgggagg ttttattatg aaaatcaaaa aattattaaa 1080
gacattatta attattttat tatgttttg 1109
<210> 17
<211> 1159
<212> ДНК
<213> Staphylococcus aureus
<400> 17
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atcaacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcgt ggagaagcgt atcacaaata aaactaaaaa 480
ataagttgta tataacttat tttgaaattg gttaagtata tagtatctcc aataaaatgt 540
agttaactta cgataatgct gaactatagc tttgtaaact aaaatgtaaa taattacaat 600
caaattgcaa caatatagtt caagaatgct acaatttgag gacagattga tagcattaat 660
ccctttaaaa tgaagctagg agataactta cattatgatt agtaaacaaa taaaggattt 720
acgaaagcaa cataattata ctcaagaaga gctagctgaa aaattaaata cttcaagaca 780
aacaatttct aaatgggaac aaggtatttc agaaccagac ttaattatgc ttatgcaatt 840
gtcacaatta ttttctgtta gtacagacta tctcattaca ggaagtgaca atattattaa 900
aaaagataat aaaagctatt atgaaatgaa tttttgggca tttatgtctg aaaaatggtg 960
ggtaattatt attatagtaa tcataatttg tggaacaata ggacaaattt tttcaaacta 1020
atgtaagtat ctctcaaata ttttgggagg ttttattatg aaaatcaaaa aattattaaa 1080
gacattatta attattttat tatgttttgt attgtctgtt attgtgcaaa atatttcaat 1140
gctatggcat attgtgagc 1159
<210> 18
<211> 1125
<212> ДНК
<213> Staphylococcus aureus
<400> 18
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactat 360
gcactatcat ttagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtata gagcatttaa gattatgcgt ggagaggcgt atcataaata aaactaaaaa 480
acggattgtg tataatatat tttaaatata aaaaggattg attttatgtt aaataaatta 540
gaaaatgtta gttataaatc attcgataat tacactagtg aagatgattt gactaaagta 600
aatatatttt ttggaagaaa tgggagtgga aaaagctcat taagtgaatg gttaagaaga 660
ctagataatg aaaaaagtgt tatctttaat actggttact taaaaaataa tattgaagaa 720
gttgaagaaa tagatggtgt gaatttggtt attggagaag aatctataaa tcatagtgac 780
caaattaagc atttaaatag cgctataaat agtttagaaa attttattac tcggaaaaat 840
agtgaactta agcattcaaa agaaagaatt tacaataaaa tgaatatcag actaaatgaa 900
gctagagaaa gatttgaaat aggtagtaat gtggttaagc agaagaggaa tgctgacaaa 960
gatccagtta atgcttttta tagttggaag aaaaatgcta acgatataat tcaagagatg 1020
actattgaat ctttagatga attagaagaa agaataacaa gaaaagaagt cttattaaat 1080
aatataaaaa caccaatttt agcttttgat tataatgatt ttagt 1125
<210> 19
<211> 4149
<212> ДНК
<213> Staphylococcus aureus
<400> 19
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa ttaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgag 420
caagtgtata gagcatttaa gattatgcgt ggagaagcat atcataagtg atgcggtttt 480
tattaattag ttgctaaaaa atgaagtatg caatattaat tattattaaa ttttgatata 540
tttaaagaaa gattaagttt agggtgaatg aatggcttat caaagtgaat atgcattaga 600
aaatgaagta cttcaacaac ttgaggaatt gaactatgaa agagtaaata tacataatat 660
taaattagaa attaatgaat atctcaaaga actaggagtg ttgaaaaatg aataagcaga 720
caaatactcc agaactaaga tttccagagt ttgatgagga atggaaaaaa aggaaattag 780
gtgaagtagt aaattataaa aatggtggtt catttgaaag tttagtgaaa aaccatggtg 840
tatataaact cataactctt aaatctgtta atacagaagg aaagttgtgt aattctggaa 900
aatatatcga tgataaatgt gttgaaacat tgtgtaatga tactttagta atgatactga 960
gcgagcaagc accaggacta gttggaatga ctgcaattat acctaataat aatgagtatg 1020
tactaaatca acgagtagca gcactagtgc ctaaacaatt tatagatagt caatttctat 1080
ctaagttaat taatagaaac cagaaatatt tcagtgtgag atctgctgga acaaaagtga 1140
aaaatatttc taaaggacat gtagaaaact ttaatttttt atctcctaat tacactgaac 1200
aacaaaaaat aggtaatttc ttcagcaaac tcgaccgcca gattgagtta gaagaagaga 1260
aacttgaact cttagagcaa caaaagcgtg gatatattca gaagattttt tctcaagatt 1320
taagatttaa agatgaaaat ggaaacagtt atcctgattg gtctattaaa aagattgaag 1380
atatttctaa agttaataaa gggtttactc caaatacaaa aaatgataaa tactgggatg 1440
aattaaatga aaattggtta tctatagcag gtatgacaca gaaatatttg tataaaggaa 1500
ataaaggaat tactgaaaaa ggtgcatcaa agcatgtaaa agtagataaa gatactctaa 1560
taatgagttt taaattgact ttaggtaagt tagctatagt aaaagagcct atctatacaa 1620
atgaagctat atgccatttc gtatggaaag aaagtaatgt taatactgag tatatgtact 1680
actatttaaa ttctataaat ataagtactt ttggtgcaca ggcagttaaa ggagtaacat 1740
taaataacga tgcaattaat agtattatag taaagttacc agtgatacaa gaacaaaata 1800
aaatagcata ctttttcaat aaattagata aattaattga aaaacaatct tctaaagtag 1860
aattattaaa acaacgcaaa caaggatttt tacagaaaat gtttgtttaa ttcttataaa 1920
gttctattat gtaaaatatt aaatagagat aacattatga aagcgagccc aagacataaa 1980
gtttttgaat aaataaaaaa gataatttct atcaaattaa tatagaaatt gtctttttta 2040
taaatttttt gattattttt agctgattga gctgttactt ttcttataat aagtgctatt 2100
agcacaaatc ctagttctct tttggctttg tttattcctc ttacggacat tcgagtgaaa 2160
cccattttaa ttttattaga agtaatttag gtttgaaccc acctaaataa atatatgagt 2220
tattttttta tgctacaaaa tatattcaga tttcaataat gacataaaat aggcatcttt 2280
atatttacct ttagtgtaga attgctcttt gagtaatcct tctgttttaa atccttgtga 2340
ctcgtatata tgcacagctt ttttgttatc tgtatcaaca tatagataaa ttttgtgcat 2400
gtttaatata tcgaatgcat aatttatcgc tttttcgaat gcgaattttg cataaccttt 2460
accactgaac tcaggtttaa taattatttg tatttcacaa ttacgatgga tgtaattaat 2520
ttctactaat tcaacaatac ctacgacttg attttcatct tcaacaataa aacgtctctc 2580
tgattcatct aataaatgct tatcaaataa atattgaagt tccgttaagg attcatatgg 2640
ttcttcaaac caataagaca taatagaata ttcattattt aattcatgaa caaaaagtaa 2700
atcactatac tctaatgctc ttagtttcat aattccactc ccaaaatttt ctcatatatt 2760
tgcattataa atataaataa cgaataagtc atcattcact gtgaatactc tattttaaca 2820
attcaccaca tactaattct cattttcttg ttattctcga tttattactc ttactatgaa 2880
acctataaaa ttctcacatt tgtttgtatt aagaataaat acgtcgatag taacaataaa 2940
aaaataaata ataaagcatc cctcaccgta aaagtgaagg atgctctagt tttattgaaa 3000
tatacatttc attttgttaa ataattatta ataatatttt gaaaatcatt attacgtgaa 3060
atcttcatag attttatcaa gtatttcttt gccttcaatt gctgtgaagt gatgtaccaa 3120
tctattttta caatcatatg taattttgtg acgctaggta attagtaatt gttcgtcagt 3180
ctgattgtat agtatcaagt ttcatagata atactctttg attttaatgt ccactttgac 3240
gtgctttaag attgagtata tacataatgt cattgtggaa tgttaaaaat cctacaaatg 3300
tttattcatc tgcaggattt ttaaatctcc aagaataaaa atcatcatag gacaactgga 3360
ttattgtttg gataaataac gtaaacaata attaggtact attatttatt tttgtttatt 3420
ctttttccta acaaaataaa gaaaagaata aacgcaattg ttaaaaatat gtgtcctaaa 3480
ccagcaatac cagcaatagc aggacttaca cttagatctt taatggtaga aataccgttc 3540
acgaattgca ttgccacagt aacaagcaca cctaaatggt atatataaaa gaaactgtta 3600
aacagctttg tatgagttgt taatttgaat tggccctcga taatcatgaa aattaagaac 3660
ataattgtac ctagtactaa taaatgtgta tgtgtaacat ttaattgaga aaaaccgcta 3720
aaatcttccg cttttgtcat ttctctataa aatagaccac ttaataaccc taatagtgta 3780
tagagcgctg aactatacat taatcttttc attttaattc cccctatttt taattacgag 3840
ataagtatag cggtagttta tgaactgagt atgaacttac aacaaaaaaa ttaatgaagt 3900
actttacaat aaactcaatt tattagatgg tggagggacg aaaaaggatt ttagaaaaat 3960
aaattaatat atttttattt tgataagtaa taattaataa tatcttggaa atcattgtta 4020
agtattgttg taatacaatc gtcattcata aaatcttcat agtttttatc aagaatttct 4080
tcatcttcga tagatgtgaa atgattagct aaccctttta taatttaagt gtaatttgtg 4140
aatctaaac 4149
<210> 20
<211> 1081
<212> ДНК
<213> Staphylococcus aureus
<400> 20
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atcaacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcgt ggagaagcat atcataagtg atgcggtttt 480
tattaattag ttgctaaaaa atgaagtatg caatattaat tattattaaa ttttgatata 540
tttaaagaaa gattaagttt agggtgaatg aatggcttat caaagtgaat atgcattaga 600
aaatgaagta cttcaacaac ttgaggaatt gaactatgaa agagtaaata tacataatat 660
taaattagaa attaatgaat atctcaaaga actaggagtg ttgaaaaatg aataagcaga 720
caaatactcc agaactaaga tttccagagt ttgatgagga atggaaaaaa aggaaattag 780
gtgaagtagt aaattataaa aatggtggtt catttgaaag tttagtgaaa aaccatggtg 840
tatataaact cataactctt aaatctgtta atacagaagg aaagttgtgt aattctggaa 900
aatatatcga tgataaatgt gttgaaacat tgtgtaatga tactttagta atgatactga 960
gcgagcaagc accaggacta gttggaatga ctgcaattat acctaataat aatgagtatg 1020
tactaaatca acgagtagca gcactagtgc ctaaacaatt tatagatagt caatttctat 1080
c 1081
<210> 21
<211> 1054
<212> ДНК
<213> Staphylococcus aureus
<400> 21
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atcaacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcgt ggagaagcat atcataagtg atgcggtttt 480
tattaattag ttgctaaaaa atgaagtatg caatattaat tattattaaa ttttgatata 540
tttaaagaaa gattaagttt agggtgaatg aatggcttat caaagtgaat atgcattaga 600
aaatgaagta cttcaacaac ttgaggaatt gaactatgaa agagtaaata tacataatat 660
taaattagaa attaatgaat atctcaaaga actaggagtg ttgaaaaatg aataagcaga 720
caaatactcc agaactaaga tttccagagt ttgatgagga atggaaaaaa aggaaattag 780
gtgaagtagt aaattataaa aatggtggtt catttgaaag tttagtgaaa aaccatggtg 840
tatataaact cataactctt aaatctgtta atacagaagg aaagttgtgt aattctggaa 900
aatatatcga tgataaatgt gttgaaacat tgtgtaatga tactttagta atgatactga 960
gcgagcaagc accaggacta gttggaatga ctgcaattat acctaataat aatgagtatg 1020
tactaaatca acgagtagca gcactagtgc ctaa 1054
<210> 22
<211> 4353
<212> ДНК
<213> Staphylococcus aureus
<400> 22
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagcgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa ttaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gtattcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgag 420
caagtgtata gagcatttaa gattatgcgt ggagaagcat atcataaatg atgcggtttt 480
ttcagccgct tcataaaggg attttgaatg tatcagaaca tatgaggttt atgtgaattg 540
ctgttatgtt tttaagaagc atatcataaa tgatgcggtt ttttcagccg cttcataaag 600
ggattttgaa tgtatcagaa catatgaggt ttatgtgaat tgctgttatg tttttaagaa 660
gcatatcata agtgatgcgg tttttattaa ttagttgcta aaaaatgaag tatgcaatat 720
taattattat taaattttga tatatttaaa gaaagattaa gtttagggtg aatgaatggc 780
ttatcaaagt gaatatgcat tagaaaatga agtacttcaa caacttgagg aattgaacta 840
tgaaagagta aatatacata atattaaatt agaaattaat gaatatctca aagaactagg 900
agtgttgaaa aatgaataag cagacaaata ctccagaact aagatttcca gagtttgatg 960
aggaatggaa aaaaaggaaa ttaggtgaag tagtaaatta taaaaatggt ggttcatttg 1020
aaagtttagt gaaaaaccat ggtgtatata aactcataac tcttaaatct gttaatacag 1080
aaggaaagtt gtgtaattct ggaaaatata tcgatgataa atgtgttgaa acattgtgta 1140
atgatacttt agtaatgata ctgagcgagc aagcaccagg actagttgga atgactgcaa 1200
ttatacctaa taataatgag tatgtactaa atcaacgagt agcagcacta gtgcctaaac 1260
aatttataga tagtcaattt ctatctaagt taattaatag aaaccagaaa tatttcagtg 1320
tgagatctgc tggaacaaaa gtgaaaaata tttctaaagg acatgtagaa aactttaatt 1380
ttttatctcc taattacact gaacaacaaa aaataggtaa tttcttcagc aaactcgacc 1440
gccagattga gttagaagaa gagaaacttg aactcttaga gcaacaaaag cgtggatata 1500
ttcagaagat tttttctcaa gatttaagat ttaaagatga aaatggaaac agttatcctg 1560
attggtctat taaaaagatt gaagatattt ctaaagttaa taaagggttt actccaaata 1620
caaaaaatga taaatactgg gatgaattaa atgaaaattg gttatctata gcaggtatga 1680
cacagaaata tttgtataaa ggaaataaag gaattactga aaaaggtgca tcaaagcatg 1740
taaaagtaga taaagatact ctaataatga gttttaaatt gactttaggt aagttagcta 1800
tagtaaaaga gcctatctat acaaatgaag ctatatgcca tttcgtatgg aaagaaagta 1860
atgttaatac tgagtatatg tactactatt taaattctat aaatataagt acttttggtg 1920
cacaggcagt taaaggagta acattaaata acgatgcaat taatagtatt atagtaaagt 1980
taccagtgat acaagaacaa aataaaatag catacttttt caataaatta gataaattaa 2040
ttgaaaaaca atcttctaaa gtagaattat taaaacaacg caaacaagga tttttacaga 2100
aaatgtttgt ttaattctta taaagttcta ttatgtaaaa tattaaatag agataacatt 2160
atgaaagcga gcccaagaca taaagttttt gaataaataa aaaagataat ttctatcaaa 2220
ttaatataga aattgtcttt tttataaatt ttttgattat ttttagctga ttgagctgtt 2280
acttttctta taataagtgc tattagcaca aatcctagtt ctcttttggc tttgtttatt 2340
cctcttacgg acattcgagt gaaacccatt ttaattttat tagaagtaat ttaggtttga 2400
acccacctaa ataaatatat gagttatttt tttatgctac aaaatatatt cagatttcaa 2460
taatgacata aaataggcat ctttatattt acctttagtg tagaattgct ctttgagtaa 2520
tccttctgtt ttaaatcctt gtgactcgta tatatgcaca gcttttttgt tatctgtatc 2580
aacatataga taaattttgt gcatgtttaa tatatcgaat gcataattta tcgctttttc 2640
gaatgcgaat tttgcataac ctttaccact gaactcaggt ttaataatta tttgtatttc 2700
acaattacga tggatgtaat taatttctac taattcaaca atacctacga cttgattttc 2760
atcttcaaca ataaaacgtc tctctgattc atctaataaa tgcttatcaa ataaatattg 2820
aagttccgtt aaggattcat atggttcttc aaaccaataa gacataatag aatattcatt 2880
atttaattca tgaacaaaaa gtaaatcact atactctaat gctcttagtt tcataattcc 2940
actcccaaaa ttttctcata tatttgcatt ataaatataa ataacgaata agtcatcatt 3000
cactgtgaat actctatttt aacaattcac cacatactaa ttctcatttt cttgttattc 3060
tcgatttatt actcttacta tgaaacctat aaaattctca catttgtttg tattaagaat 3120
aaatacgtcg atagtaacaa taaaaaaata aataataaag catccctcac cgtaaaagtg 3180
aaggatgctc tagttttatt gaaatataca tttcattttg ttaaataatt attaataata 3240
ttttgaaaat cattattacg tgaaatcttc atagatttta tcaagtattt ctttgccttc 3300
aattgctgtg aagtgatgta ccaatctatt tttacaatca tatgtaattt tgtgacgcta 3360
ggtaattagt aattgttcgt cagtctgatt gtatagtatc aagtttcata gataatactc 3420
tttgatttta atgtccactt tgacgtgctt taagattgag tatatacata atgtcattgt 3480
ggaatgttaa aaatcctaca aatgtttatt catctgcagg atttttaaat ctccaagaat 3540
aaaaatcatc ataggacaac tggattattg tttggataaa taacgtaaac aataattagg 3600
tactattatt tatttttgtt tattcttttt cctaacaaaa taaagaaaag aataaacgca 3660
attgttaaaa atatgtgtcc taaaccagca ataccagcaa tagcaggact tacacttaga 3720
tctttaatgg tagaaatacc gttcacgaat tgcattgcca cagtaacaag cacacctaaa 3780
tggtatatat aaaagaaact gttaaacagc tttgtatgag ttgttaattt gaattggccc 3840
tcgataatca tgaaaattaa gaacataatt gtacctagta ctaataaatg tgtatgtgta 3900
acatttaatt gagaaaaacc gctaaaatct tccgcttttg tcatttctct ataaaataga 3960
ccacttaata accctaatag tgtatagagc gctgaactat acattaatct tttcatttta 4020
attcccccta tttttaatta cgagataagt atagcggtag tttatgaact gagtatgaac 4080
ttacaacaaa aaaattaatg aagtacttta caataaactc aatttattag atggtggagg 4140
gacgaaaaag gattttagaa aaataaatta atatattttt attttgataa gtaataatta 4200
ataatatctt ggaaatcatt gttaagtatt gttgtaatac aatcgtcatt cataaaatct 4260
tcatagattt tatcaagaat ttcttcatct tcgatagatg tgaaatgatt agctaaccct 4320
tttataattt aagtgtaatt tgtgaatcta aac 4353
<210> 23
<211> 2031
<212> ДНК
<213> Staphylococcus aureus
<400> 23
accattttag ctgtagggaa actaaaagag aaatattgga agcaagccat agcagaatat 60
gaaaaacgtt taggcccata caccaagata gacatcatag aagttccaga cgaaaaagca 120
ccagaaaata tgagtgacaa agaaattgag caagtaaaag aaaaagaagg ccaacgaata 180
ctagccaaaa tcaaaccaca atccacagtc attacattag aaatacaagg aaagatgcta 240
tcttccgaag gattggccca agaattgaac caacgcatga cccaagggca aagcgacttt 300
gttttcgtca ttggcggatc aaacggcctg cacaaggacg tcttacaacg cagtaactac 360
gcactatcat tcagcaaaat gacattccca catcaaatga tgcgggttgt gttaattgaa 420
caagtgtaca gagcatttaa gattatgcga ggagaagcgt atcacaaata aaactaaaaa 480
atagattgtg tataatataa aaggagcgga tttatattaa aactttgaat tcaaaaatta 540
ttgaaaggga agctacctta gaaattgaat ctatggcaac taatacattg aaaataaacc 600
cggatattaa ttcaaacgat acaaaaatgt ctttcgatgg agaattggaa gtgtatgatt 660
ctgaaaattt gagtaaaaaa aatttcgttg gaaaaataca agttcaagtt aaaggaaagg 720
aagtagctaa aagaggaggt aaggttattc atcgaagtaa tgtcaaaatg aatgatttaa 780
aggcatacca acgagaaggt ggtgtgtatt actttgtcgt gtatttaatc gttgagaata 840
aaaaagttgt tgagaagcag gtttatggta aacaattaca tcaattagat ttacaatttt 900
tactgcaaaa aaagcagaaa agcgtcacta taaaaatgta tgaaattgaa aatgaaaaaa 960
ttttatataa taattgcgta aaatatataa atgaaaagag attacaaaat caagtaggtc 1020
aagttaaagt aaagaacata gaaaaagctt tatcatatat agctacacct gaaaatattg 1080
tgatggatca tagaggttta ccattaaatg atttttatgg ctatataaaa attaattcat 1140
ctgaattaga tgtaactata ccagatggag tattaagtat ggaaaaagta aaaagagtga 1200
acaaaaagca gataataaaa gaaggcaagt tattatttga aggtatggta ggtattgaaa 1260
cttcaaagga gtccatctct ataactatag atgatatttt caaaattcaa acatttgaaa 1320
gtgataacaa aagtatatat acaatgttac catttaaaaa attaaatata gccgaacaat 1380
cttttaatgt tataaacgaa ttgtcgaaag gcggcgaatt ctttttggat caaatccaat 1440
tagtaatcca accttttgaa attaatatta ttgaaataaa agaaacaata aataagttga 1500
atattaaatt atcagagtat agtaacttgc tttcgtttga tgtgagtcta aaatctacag 1560
aatttgataa gcagatgaat gagataaaag gtttattaga attattggaa tataaaaatt 1620
ttaaagattt taaaatgcat aataatggat actataaaat gaagttttgt ggaaaattta 1680
tattattatt taaagacaat acatcattgt ataatgtcta ctctaatgac tttgtagata 1740
gatttgaggc tgttacaaaa gaaagagttg tacagatgcc aattgtttac acattaacaa 1800
gagatatgat tgtagatgta ctgaattttg atataaatgt tattaaagaa tgtattgaat 1860
cagataaaat tgctattcaa tctgacatta aatgggagaa attaaataac tttgcattag 1920
aattaatcgc agcctatgat gaaactcaaa gaaccgattt attagaatta gctgaatatg 1980
tattaaataa tttgctgaat tttgataatg ataaaatatt tatgaactta a 2031
<210> 24
<211> 818
<212> ДНК
<213> Staphylococcus aureus
<400> 24
aatattggaa gcaagccata gcagaatatg aaaaacgttt aggcccatac accaagatag 60
acatcataga agttccagac gaaaaagcac cagaaaatat gagcgacaaa gaaattgagc 120
aagtgtatag agcatttaag attatgcgtg gagaagcata tcataaatga tgcggttttt 180
tcagccgctt cataaaggga ttttgaatgt atcagaacat atgaggttta tgtgaattgc 240
tgttatgttt ttaagaagct tatcataagt aatgaggttc atgatttttg acatagttag 300
cctccgcagt ctttcatttc aagtaaataa tagcgaaata ttctttatac tgaatactta 360
tagtgaagca aagttctagc tttgagaaaa ttctttctgc aactaaatat agtaaattac 420
ggtaaaatat aaataagtac atattgaaga aaatgagaca taatatattt tataatagga 480
gggaatttca aatgatagac aactttatgc aggtccttaa attaattaaa gagaaacgta 540
ccaataatgt agttaaaaaa tctgattggg ataaaggtga tctatataaa actttagtcc 600
atgataagtt acccaagcag ttaaaagtgc atataaaaga agataaatat tcagttgtag 660
ggaaggttgc tactgggaac tatagtaaag ttccttggat ttcaatatat gatgagaata 720
taacaaaaga aacaaaggat ggatattatt tggtatatct ttttcatccg gaaggagaag 780
gcatatactt atcttgaatc aaggatggtc aaagataa 818
<210> 25
<211> 1073
<212> ДНК
<213> Staphylococcus aureus
<400> 25
tattggaagc aagccatagc agaatatgaa aaacgtttag gcccatacac caagatagac 60
atcatagaag ttccagacga aaaagcacca gaaaatatga gcgacaaaga aattgagcaa 120
gtaaaagaaa aagaaggcca acgaatacta gccaaaatca aaccacaatc cacagtcatt 180
acattagaaa tacaaggaaa gatgctatct tccgaaggat tggcccaaga attgaaccaa 240
cgcatgaccc aagggcaaag cgactttgta ttcgtcattg gcggatcaaa cggcctgcac 300
aaggacgtct tacaacgcag taactatgca ctatcattta gcaaaatgac attcccacat 360
caaatgatgc gggttgtgtt aattgaacaa gtgtatagag catttaagat tatgcgtgga 420
gaggcgtatc ataagtgatg cttgttagaa tgatttttaa caatatgaaa tagctgtgga 480
agcttaaaca atttgtttat ctaagtactt atttaataat tgattgaact gtgattggca 540
ccaggctgtc tggtaaattg agaagttggg ttttggagcg tataaatgat agaattaata 600
taaaattcaa tttgaggagt aggagattat gtcgaatata aaaacaacac tagagacgtc 660
cgtaggacta gaaaaagaca acgataagct atttgattat ataactgaat tagagattca 720
aaacacgcct gaaaaccggg aagcaaaagt tgttattgaa gaaaggttac ataaagaata 780
taaatatgaa ttagatcaaa tgacaccaga gtatggaata caaaaaggca gtgttagaat 840
aggtcatgca gatgttgtaa tatttcatga ttctaaagat aaatctcaag agaatattaa 900
aataatagta gagtgtaaaa gaaagaatcg cagggatggt attgaacaat taaaaacata 960
tcttgcaggg tgtgagtctg cagaatacgg cgtttggttt aatggagaag atatagtata 1020
tataaaacga ttgaaaaaag caccacattg gaaaacagta tttaatatac cga 1073
<210> 26
<211> 756
<212> ДНК
<213> Staphylococcus aureus
<400> 26
tgacattccc acatcaaatg atgcgggttg tgttaattga gcaagtgtat agagcattta 60
agattatgcg tggagaagcg tatcacaaat aaaactaaaa aataggttgc gcataatata 120
attagaaagg aattagacat aaattaggag tccttcacag aatagcgaag gactcccatt 180
aaatatatta tggtgtaaag aaatcacaaa tcaatatata tacttaatac catatattaa 240
cttgtactat tataaagtac gacatcagta ttaggtatca ctttgaacac atgaatttca 300
ttatcacttt tattattcac aaaaaatttt ccaattctca attactgaat tatgtgtata 360
catgttgtta aaaattaata aaggatattt atgtttgttt aaagcatatc acaagtgatg 420
cggtttttta taaagattta cttgttagtg attttgataa aaatgcttaa tactatttca 480
ataatatgta tttaaaaatt agattaatag tatttaactt caaatggcct cgtataaact 540
catagcaaat taacgtaaat caatgaaata aaatgaaaac aatttcaaga atacattata 600
aacataaagt atacaaaaaa taaatgagcg tatttgttta aacgtataca ctcattttta 660
ttaaattaat ttattatatt ttacgattgt tatttatgaa attaacaaat tccatttttg 720
atagtgaaat taaaagcttt atcacttatt attgat 756
<210> 27
<211> 771
<212> ДНК
<213> Staphylococcus aureus
<400> 27
tgacattccc acatcaaatg atgcgggttg tgttaattga gcaagtgtat agagcattta 60
agattatgcg tggagaagcg tatcacaaat aaaactaaaa aataggttgc gcataatata 120
attagaaagg aattagacat aaattaggag tccttcacag aatagcgaag gactcccatt 180
aaatatatta tggtgtaaag aaatcacaaa tcaatatata tacttaatac catatattaa 240
cttgtactat tataaagtac gacatcagta ttaggtatca ctttgaacac atgaatttca 300
ttatcacttt tattattcac aaaaaatttt ccaattctca attactgaat tatgtgtata 360
catgttgtta aaaattaata aaggatattt atgtttgttt aaagcatatc acaagtgatg 420
cggtttttta taaagattta cttgttagtg attttgataa aaatgcttaa tactatttca 480
ataatatgta tttaaaaatt agattaatag tatttaactt caaatggcct cgtataaact 540
catagcaaat taacgtaaat caatgaaata aaatgaaaac aatttcaaga atacattata 600
aacataaagt atacaaaaaa taaatgagcg tatttgttta aacgtataca ctcattttta 660
ttaaattaat ttattatatt ttacgattgt tatttatgaa attaacaaat tccatttttg 720
atagtgaaat taaaagcttt atcacttatt attgataatt ttgactgcat c 771
<210> 28
<211> 680
<212> ДНК
<213> Staphylococcus aureus
<400> 28
ttcagcaaaa tgacattccc acatcaaatg atgcgggttg tgttaattga acaagtgtac 60
agagcattta agattatgcg tggagaagcg tatcataagt agcggaggag ttttttacct 120
tgtgacttat cataaagtac gatgtttatg taagtgatta tcattattta agcaggtttt 180
tcaaattaaa taataacaag aataaaatgc acttagcgac attgaaattt attaatctag 240
taaactaata gatttataga aaattttatt tgcaagggga taattttgaa aagtagtatt 300
ttctatcttt ccataataca ttgtaattac aacggagggg atattgtgat gaagtgtata 360
gataaaacgt gggttagcta ttataaagaa ttagctgata agttaacaga ttatcaaaat 420
aaacgttatg aattaattga aatagtgaag gaagtatata aaaaaacggg aataaaattc 480
cctactttag caagtgataa tgtattgatg gacatagatc cttttacaat atttgcatta 540
tttaataaaa attccatgag agaaactaat aaggtaaaaa tattaacaga attagcttcg 600
gaattgaata ttaagtccaa aattccgtca gtttttgaca gtattccaac agtcaataat 660
ctgaatgcta catattataa 680
<210> 29
<211> 1119
<212> ДНК
<213> Staphylococcus aureus
<400> 29
gacattccca catcaaatga tgcgggttgt gttaattgag caagtgtata gagcatttaa 60
gattatgcgt ggagaggcgt atcataagta aaactaaaaa attctgtatg aggagataat 120
aatttggagg gtgttaaatg gtggacatta aatccacgtt cattcaatat ataagatata 180
tcacgataat tgcgcatata acttaagtag tagctaacag ttgaaattag gccctatcaa 240
attggtttat atctaaaatg attaatatag aatgcttctt tttgtcctta ttaaattata 300
aaagtaactt tgcaatagaa acagttattt cataatcaac agtcattgac gtagctaagt 360
aatgataaat aatcataaat aaaattacag atattgacaa aaaatagtaa atataccaat 420
gaagtttcaa aagaacaatt ccaagaaatt gagaatgtaa ataataaggt caaagaattt 480
tattaagatt tgaaagagta tcaatcaaga aagatgtagt tttttaataa actatttgga 540
aaataattat cataatttaa aaactgacaa tttgcgagac tcataaaatg taataatgga 600
aatagatgta aaatataatt aaggggtgta atatgaagat taatatttat aaatctattt 660
ataattttca ggaaacaaat acaaattttt tagagaatct agaatcttta aatgatgaca 720
attatgaact gcttaatgat aaagaacttg ttagtgattc aaatgaatta aaattaatta 780
gtaaagttta tatacgtaaa aaagacaaaa aactattaga ttggcaatta ttaataaaga 840
atgtatacct agatactgaa gaagatgaca atttattttc agaatccggt catcattttg 900
atgcaatatt atttctcaaa gaagatacta cattacaaaa taatgtatat attatacctt 960
ttggacaagc atatcatgat ataaataatt tgattgatta tgacttcgga attgattttg 1020
cagaaagagc aatcaaaaat gaagacatag ttaataaaaa tgttaatttt tttcaacaaa 1080
acaggcttaa agagattgtt aattatagaa ggaatagtg 1119
<210> 30
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 30
caaattgtag cattcttgaa ctat 24
<210> 31
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 31
ctattgttcc acaaattatg attact 26
<210> 32
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 32
ctcccatttc ttccaaaaaa tata 24
<210> 33
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 33
cttcttttct tgttattctt tcttct 26
<210> 34
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 34
taatttcctt tttttccatt cctc 24
<210> 35
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 35
gagcggattt atattaaaac tttg 24
<210> 36
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 36
gttgccatag attcaatttc taag 24
<210> 37
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 37
cgaggagaag cgtatcacaa 20
<210> 38
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 38
agttgccata gattcaattt ctaaggt 27
<210> 39
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 39
gtcaaaaatc atgaacctca ttacttatg 29
<210> 40
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 40
caccctgcaa gatatgttt 19
<210> 41
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 41
cgtggagagg cgtatcataa gt 22
<210> 42
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 42
gctccaaaac ccaacttctc aa 22
<210> 43
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 43
aatggaattt gttaatttca taaat 25
<210> 44
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 44
ttccgaagct aattctgtta ata 23
<210> 45
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 45
ttccgaagtc ataatcaatc aaatt 25
<210> 46
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 46
gccaaaatca aaccacaatc cac 23
<210> 47
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 47
gccaaaatca aaccacaatc aac 23
<210> 48
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 48
accacgaatg tttgctgcta atg 23
<210> 49
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 49
cgcttgccac atcaaatgat gcgggttgtg caagcg 36
<210> 50
<211> 36
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 50
cccaccccac atcaaatgat gcgggttgtg ggtggg 36
<210> 51
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 51
cccgcgcgta gttactgcgt tgtaagacgt ccgcggg 37
<210> 52
<211> 48
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 52
cgaccggatt cccacatcaa atgatgcggg ttgtgttaat tccggtcg 48
<210> 53
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 53
cccgcgcrta gttactrcgt tgtaagacgt ccgcggg 37
<210> 54
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 54
ccccgtagtt actgcgttgt aagacgggg 29
<210> 55
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 55
cccgcgcata gttactgcgt tgtaagacgt ccgcggg 37
<210> 56
<211> 37
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 56
cccgcgcgta gttactacgt tgtaagacgt ccgcggg 37
<210> 57
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 57
ggatcaaacg gcctgcaca 19
<210> 58
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 58
atcaaatgat gcgggttgtg t 21
<210> 59
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 59
tcattggcgg atcaaacgg 19
<210> 60
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 60
acaacgcagt aactacgcac ta 22
<210> 61
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 61
taactacgca ctatcattca gc 22
<210> 62
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 62
acatcaaatg atgcgggttg tg 22
<210> 63
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 63
tcaaatgatg cgggttgtgt ta 22
<210> 64
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 64
caaatgatgc gggttgtgtt aatt 24
<210> 65
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 65
gtcaaaaatc atgaacctca ttacttatg 29
<210> 66
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 66
ctatgtcaaa aatcatgaac ctcattac 28
<210> 67
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 67
ggaggctaac tatgtcaaaa atc 23
<210> 68
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 68
gctgaaaaaa ccgcatcatt trtgrta 27
<210> 69
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 69
gctgaaaaaa ccgcatcatt tatgata 27
<210> 70
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 70
atttcatata tgtaattcct ccacatctc 29
<210> 71
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 71
tttagtttta tttatgatac gcttctcca 29
<210> 72
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 72
ctctataaac atcgtatgat attgc 25
<210> 73
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 73
caaatattat ctcgtaattt accttgttc 29
<210> 74
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 74
ctctgcttta tattataaaa ttacggctg 29
<210> 75
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 75
ttcgttccct ccattaactg tc 22
<210> 76
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 76
tcaccgtctt tcttttgacc tt 22
<210> 77
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 77
cactttttat tcttcaaaga tttgagc 27
<210> 78
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 78
atggaaattc ttaatcttta cttgtacc 28
<210> 79
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 79
cagcaattcw cataaacctc ata 23
<210> 80
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 80
acaaactttg aggggatttt tagtaaa 27
<210> 81
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 81
tgataagcca ttcattcacc ctaa 24
<210> 82
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 82
tagttactgt gttgtaagac gtcc 24
<210> 83
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 83
ctactatgaa ctgtgcaatt tgttct 26
<210> 84
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 84
gcaattcaca taaacctcat atgttc 26
<210> 85
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 85
acctcatatg ttctgataca ttca 24
<210> 86
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 86
gcaattcaca taaacctcat at 22
<210> 87
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 87
cataacagca attcacataa acctc 25
<210> 88
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 88
taacagcaat tcacataaac ct 22
<210> 89
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 89
cgctattatt tacttgaaat gaaagac 27
<210> 90
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 90
cttgaaatga aagactgcgg a 21
<210> 91
<211> 54
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 91
ttgcttcact ataagtattc agtataaaga atttacttga aatgaaagac tgcg 54
<210> 92
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 92
atttacttga aatgaaagac tgcg 24
<210> 93
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 93
aaagaatatt tcgctattat ttacttgaa 29
<210> 94
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 94
tcagtataaa gaatatttcg ctattattt 29
<210> 95
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 95
tgaaatgaaa gactgcggag 20
<210> 96
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 96
aacctcatat gttctgatac attcaaa 27
<210> 97
<211> 28
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 97
tatgtcaaaa atcatgaacc tcattact 28
<210> 98
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 98
cataacagca attcacataa acctc 25
<210> 99
<211> 17
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 99
gactgcggag gctaact 17
<210> 100
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 100
atccctttat gaagcggc 18
<210> 101
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 101
tgaaatgaaa gactgcggag 20
<210> 102
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 102
gcaaggtata atccaatatt tcatatatgt 30
<210> 103
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 103
agttccataa tcaatataat ttgtacagt 29
<210> 104
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 104
acatcgtatg atattgcaag gta 23
<210> 105
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 105
ctttcattct ttcttgattc cattag 26
<210> 106
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 106
cactctataa acatcgtatg atattgc 27
<210> 107
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 107
ttcttaattt aattgtagtt ccataatcaa 30
<210> 108
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 108
aattatacac aacctaattt ttagttttat 30
<210> 109
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 109
aatttttagt tttatttatg atacgcttc 29
<210> 110
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 110
acacaaccta atttttagtt ttatttatga 30
<210> 111
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 111
tttattaaac actctataaa catcgtatga 30
<210> 112
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 112
tcacatctca ttaaattttt aaattataca c 31
<210> 113
<211> 31
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 113
ccacatctca ttaaattttt aaattataca c 31
<210> 114
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 114
atattataca caatccgttt tttagtttta 30
<210> 115
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 115
acacaatccg ttttttagtt ttatttatg 29
<210> 116
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 116
ttctaattta tttaacataa aatcaatcct 30
<210> 117
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 117
caatcctttt tatatttaaa atatattata cac 33
<210> 118
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 118
aagtcgcttt gcctttgggt ca 22
<210> 119
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 119
tacaaagtcg ctttgccttt gggtca 26
<210> 120
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 120
ggccgtttga tccgccaat 19
<210> 121
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 121
aagtcgcttt gcccttgggt a 21
<210> 122
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 122
aagtcgcttt gcccttgggt 20
<210> 123
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 123
aagtcgcttt gcccttgggt ca 22
<210> 124
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 124
aagtcgcttt gcccttggg 19
<210> 125
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 125
caagaattga accaacgcat 20
<210> 126
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 126
caatgacgaa tacatagtcg ctttgccctt 30
<210> 127
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 127
cgtttgatcc gccaatgacg a 21
<210> 128
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 128
gccaatcctt cggaagatag ca 22
<210> 129
<211> 18
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 129
attaacacaa cccgcatc 18
<210> 130
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 130
gtcgctttgc ccttgggtc 19
<210> 131
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 131
tcgctttgcc cttgggtcat 20
<210> 132
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 132
ggccgtttga tccgccaat 19
<210> 133
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 133
gtccttgtgc aggccgtttg at 22
<210> 134
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 134
cttgggtcat gcgttggttc aatt 24
<210> 135
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 135
cgaatacaaa gtcgctttgc ccttggg 27
<210> 136
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 136
atgcgttggt tcaattcttg 20
<210> 137
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 137
gcgttggttc aattcttggg 20
<210> 138
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 138
acccaagggc aaagcgactt 20
<210> 139
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 139
ggtaatgcgt tggttcaatt cttg 24
<210> 140
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 140
acaaagtcgc tatgcccttg ggtca 25
<210> 141
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 141
ctttccttgt atttctaatg taatgactg 29
<210> 142
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 142
ttgatgtggg aatgtcattt tgctgaa 27
<210> 143
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 143
gcgttggttc aattcttggg ccaat 25
<210> 144
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 144
gttggttcaa ttcttgggcc aatccttcg 29
<210> 145
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 145
cgaatacaaa gtcgctttgc ccttgg 26
<210> 146
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 146
gccaatgacg aatacaaagt cgctttgcc 29
<210> 147
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 147
tgggccaatc cttcggaaga tagca 25
<210> 148
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 148
atgcgttggt tcgattcttg 20
<210> 149
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 149
catgcgttgg ttcgattctt g 21
<210> 150
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 150
aagtcgcttt gcccttggg 19
<210> 151
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 151
catgcgttgg ttcgattctt g 21
<210> 152
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 152
aagtcgcttt gcccttgggt cat 23
<210> 153
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 153
tgctcaatta acacaacccg catca 25
<210> 154
<211> 33
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 154
gccgcgctgc tcaattaaca caacccgcgc ggc 33
<210> 155
<211> 32
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 155
gccgcgcatg cgttggttca attctgcgcg gc 32
<210> 156
<211> 2123
<212> ДНК
<213> Staphylococcus aureus
<400> 156
ctccatatca caaaaattat aacattattt tgacataaat actacatttg taatatacta 60
caaatgtagt cttatataag gaggatattg atgaaaaaga taaaaattgt tccacttatt 120
ttaatagttg tagttgtcgg gtttggtata tatttttatg cttcaaaaga taaagaaatt 180
aataatacta ttgatgcaat tgaagataaa aatttcaaac aagtttataa agatagcagt 240
tatatttcta aaagcgataa tggtgaagta gaaatgactg aacgtccgat aaaaatatat 300
aatagtttag gcgttaaaga tataaacatt caggatcgta aaataaaaaa agtatctaaa 360
aataaaaaac gagtagatgc tcaatataaa attaaaacaa actacggtaa cattgatcgc 420
aacgttcaat ttaattttgt taaagaagat ggtatgtgga agttagattg ggatcatagc 480
gtcattattc caggaatgca gaaagaccaa agcatacata ttgaaaattt aaaatcagaa 540
cgtggtaaaa ttttagaccg aaacaatgtg gaattggcca atacaggaac agcatatgag 600
ataggcatcg ttccaaagaa tgtatctaaa aaagattata aagcaatcgc taaagaacta 660
agtatttctg aagactatat caaacaacaa atggatcaaa attgggtaca agatgatacc 720
ttcgttccac ttaaaaccgt taaaaaaatg gatgaatatt taagtgattt cgcaaaaaaa 780
tttcatctta caactaatga aacagaaagt cgtaactatc ctctagaaaa agcgacttca 840
catctattag gttatgttgg tcccattaac tctgaagaat taaaacaaaa agaatataaa 900
ggctataaag atgatgcagt tattggtaaa aagggactcg aaaaacttta cgataaaaag 960
ctccaacatg aagatggcta tcgtgtcaca atcgttgacg ataatagcaa tacaatcgca 1020
catacattaa tagagaaaaa gaaaaaagat ggcaaagata ttcaactaac tattgatgct 1080
aaagttcaaa agagtattta taacaacatg aaaaatgatt atggctcagg tactgctatc 1140
caccctcaaa caggtgaatt attagcactt gtaagcacac cttcatatga cgtctatcca 1200
tttatgtatg gcatgagtaa cgaagaatat aataaattaa ccgaagataa aaaagaacct 1260
ctgctcaaca agttccagat tacaacttca ccaggttcaa ctcaaaaaat attaacagca 1320
atgattgggt taaataacaa aacattagac gataaaacaa gttataaaat cgatggtaaa 1380
ggttggcaaa aagataaatc ttggggtggt tacaacgtta caagatatga agtggtaaat 1440
ggtaatatcg acttaaaaca agcaatagaa tcatcagata acattttctt tgctagagta 1500
gcactcgaat taggcagtaa gaaatttgaa aaaggcatga aaaaactagg tgttggtgaa 1560
gatataccaa gtgattatcc attttataat gctcaaattt caaacaaaaa tttagataat 1620
gaaatattat tagctgattc aggttacgga caaggtgaaa tactgattaa cccagtacag 1680
atcctttcaa tctatagcgc attagaaaat aatggcaata ttaacgcacc tcacttatta 1740
aaagacacga aaaacaaagt ttggaagaaa aatattattt ccaaagaaaa tatcaatcta 1800
ttaactgatg gtatgcaaca agtcgtaaat aaaacacata aagaagatat ttatagatct 1860
tatgcaaact taattggcaa atccggtact gcagaactca aaatgaaaca aggagaaact 1920
ggcagacaaa ttgggtggtt tatatcatat gataaagata atccaaacat gatgatggct 1980
attaatgtta aagatgtaca agataaagga atggctagct acaatgccaa aatctcaggt 2040
aaagtgtatg atgagctata tgagaacggt aataaaaaat acgatataga tgaataacaa 2100
aacagtgaag caatccgtaa cga 2123
<210> 157
<211> 1998
<212> ДНК
<213> Staphylococcus aureus
<400> 157
ttactgatct atatcaaatt gagtttttcc attatcatac aaatcatcat aaacttttcc 60
agatatagta gcattatagc tggccatccc tttattttga acgtctttaa cattaatcgc 120
cattaacata ttaggattat ttttattata tgaaacaaac caacctattt gtcttccagt 180
ttccccttga ttcattttta attctgctgt gccagattta ccaataattc gggcataatt 240
tttgtatata tcatccctat gtgttttatt aactacacgt tccataccat tagttaatat 300
atctatgtct tttttaggta taatatcttt tttccatatt tgagattttg ttttacgtaa 360
aacatgagga ttttgtatat ttccgttatt ttctaaagca ctgtatattg ataaaatttg 420
tatagggttt actagtatct cgccttggcc atatcctgaa tctgctaata atatttcatt 480
ttttaaatta ctatttgaga tttgtgcttt ataaaaggga taatcactcg ggatattttc 540
accgattccc aaatcttgca taccttgctc aaattttttg gctcctaatg ctaatgcaat 600
gcgggcaaaa aatatgttgt ctgatgattc tattgcttgc tttaaatcga tattgccgtc 660
tactacttta aatcttgtga tattataatt accccatgat gcatcttttt gccaaccctt 720
accataaata tcaaaattag tatttttgtc tagtttattt tcttttaagg ctataataga 780
tgttaatatt ttttgggttg aacctggtga tgtagtgatt tgaaatttgt tgagcaaagg 840
ctctttttta ttgttagtta atttacggta gtcattattg cttaatccat tcatgaatgg 900
ataaacatcg tacgatgggg tacttaccaa agctaaaatt tctccagttt ttggttgtaa 960
tgctgtacca gatccatcgt catttttcat atgtttataa atactttctt gtactctagc 1020
atctatagtt aaatgaagat cttttccgtt ttcagccttt ttctccaata atgtgtctaa 1080
aggtttattg tcataagtat ttgcaatgga taccttaaaa ccatcagtgt tttgcaattg 1140
tttatcatag aggcgttcta agcctttttt tccaataaca gtatttttgc tatagtttct 1200
aaattgctta ctttttaact cgtcagaatt aattggaccc acataaccta aaaggtgtac 1260
tgttgcttcg ttcaatggat aaacacggct ttttatagtg tttatttgta aattgtatga 1320
tttaattaat ttgtctatat attcatcttg tttatttatc tttttaattg gtacaaatga 1380
atctggctga acccattttt gattaacttt attggttata gcttttgtat caatttgtaa 1440
gtcacgagca atatcatcat atttttcttt gggtgttttg ttagggacaa taccgatttc 1500
atatgtattt ccagttttag ctaattctat accatttcta tcttttattt tgcctcgctc 1560
tgattttaat gtttctatat taattttctg tccatttttc aaaccaggta ctattacgtc 1620
tggtctccaa tctaatttcc aatgcttatc ttcataaata aagtttaatt gtgtattacg 1680
tcgtatagtt ccatattttg tatatatgtt atatttaaca tcaacttgct ttttatcttt 1740
tccagttttt ttaatttcat gattagtaat ttttaagtta ttgacactta aatctttgta 1800
aattttttta ttcctatcta caatttcttc ttctccatat gccagtttag atttttctga 1860
actattttta tatacttcgt tatagtttcc tttttcaata gaactaattg ttttctcaat 1920
atcgtcatct ttgaataacc aagttattat aatcataatt agtaaaagaa ctagcacact 1980
aatataaatt tttttcat 1998
<210> 158
<211> 686
<212> ДНК
<213> Staphylococcus aureus
<400> 158
atgacagaat acttattaag tgctggcata tgtatggcaa ttgtttcaat attacttata 60
gggatggcta tcagtaatgt ttcgaaaggg caatacgcaa agaggttttt ctttttcgct 120
actagttgct tagtgttaac tttagttgta gtttcaagtc taagtagctc agcaaatgca 180
tcacaaacag ataacggcgt aaatagaagt ggttctgaag atccaacagt atatagtgca 240
acttcaacta aaaaattaca taaagaacct gcgacattaa ttaaagcgat tgatggtgat 300
acggttaaat taatgtacaa aggtcaacca atgacattca gactattatt agttgataca 360
cctgaaacaa agcatcctaa aaaaggtgta gagaaatatg gccctgaagc aagtgcattt 420
acgaaaaaaa tggtagaaaa tgcaaataaa attgaagtcg agtttgacaa aggtcaaaga 480
actgataaat atggacgtgg cttagcgtat atttatgctg atggaaaaat ggtaaacgaa 540
gctttagttc gtcaaggctt ggctaaagtt gcttatgttt ataaacctaa caatacacat 600
gaacaacttt taagaaaaag tgaagcacaa gcgaaaaaag agaaattaaa tatttggagc 660
gaagacaacg ctgattcagg tcaata 686
<210> 159
<211> 686
<212> ДНК
<213> Staphylococcus aureus
<400> 159
atgacagaat acttattaag tgctggcata tgtatggcaa ttgtttcaat attacttata 60
gggatggcta tcagtaatgt ttcgaaaggg caatacgcaa agaggttttt ctttttcgct 120
actagttgct tagtgttaac tttagttgta gtttcaagtc taagtagctc agcaaatgca 180
tcacaaacag ataacggcgt aaatagaagt ggttctgaag atccaacagt atatagtgca 240
acttcaacta aaaaattaca taaagaacct gcgacattaa ttaaagcgat tgatggtgat 300
acggttaaat taatgtacaa aggtcaacca atgacattca gactattatt agttgataca 360
cctgaaacaa agcatcctaa aaaaggtgta gagaaatatg gccctgaagc aagtgcattt 420
acgaaaaaaa tggtagaaaa tgcaaataaa attgaagtcg agtttgacaa aggtcaaaga 480
actgataaat atggacgtgg cttagcgtat atttatgctg atggaaaaat ggtaaacgaa 540
gctttagttc gtcaaggctt ggctaaagtt gcttatgttt ataaacctaa caatacacat 600
gaacaacttt taagaaaaag tgaagcacaa gcgaaaaaag agaaattaaa tatttggagc 660
gaagacaacg ctgattcagg tcaata 686
<210> 160
<211> 442
<212> ДНК
<213> Staphylococcus aureus
<400> 160
gatcaatctt tgtcggtaca cgatattctt cacgactaaa taaacgctca ttcgcgattt 60
tataaatgaa tgttgataac aatgttgtat tatctactga aatctcatta cgttgcatcg 120
gaaacattgt gttctgtatg taaaagccgt cttgataatc tttagtagta ccgaagctgg 180
tcatacgaga gttatatttt ccagccaaaa cgatattttt ataatcatta cgtgaaaaag 240
gtttcccttc attatcacac aaatatttta gcttttcagt ttctatatca actgtagctt 300
ctttatccat acgttgaata attgtacgat tctgacgcac catcttttgc acacctttaa 360
tgttatttgt tttaaaagca tgaataagtt tttcaacaca acgatgtgaa tcttctaaga 420
agtcaccgta aaatgaagga tc 442
<210> 161
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 161
acagaatact tattaagtgc tggc 24
<210> 162
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 162
gtttcaatat tacttatagg gatggct 27
<210> 163
<211> 24
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 163
ctgatggaaa aatggtaaac gaag 24
<210> 164
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 164
atggaaaaat ggtaaacgaa gc 22
<210> 165
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 165
tgctgagcta cttagacttg aa 22
<210> 166
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 166
gcatttgctg agctacttag ac 22
<210> 167
<211> 25
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 167
gttgttcatg tgtattgtta ggttt 25
<210> 168
<211> 22
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 168
ttattgacct gaatcagcgt tg 22
<210> 169
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 169
cgaaacatta ctgatagcca tccctataag 30
<210> 170
<211> 40
<212> ДНК
<213> cgcaccgaaa cattactgat agccatccct ataaggtgcgArtific
<220>
<223> синтетический олигонуклеотид
<400> 170
cgcaccgaaa cattactgat agccatccct ataaggtgcg 40
<210> 171
<211> 29
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 171
acataagcaa ctttagccaa gccttgacg 29
<210> 172
<211> 41
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 172
cgcgcaacat aagcaacttt agccaagcct tgacgtgcgc g 41
<210> 173
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 173
aatctttgtc ggtacacgat attcttcacg 30
<210> 174
<211> 30
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 174
cgtaatgaga tttcagtaga taatacaaca 30
<210> 175
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 175
ttacagagtt aactgttacc 20
<210> 176
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 176
atacaaatcc agcacgctct 20
<210> 177
<211> 19
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 177
tkggaccmac ataacctaa 19
<210> 178
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 178
ggacccacat aacctaaaag 20
<210> 179
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 179
ctaacaaaac acccaaagaa 20
<210> 180
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 180
cctgaatctg ctaataatat ttc 23
<210> 181
<211> 20
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 181
tcaccaggtt caacccaaaa 20
<210> 182
<211> 21
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 182
ggccatatcc tgaatctgct a 21
<210> 183
<211> 23
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 183
gcaagcaata gaatcatcag aca 23
<210> 184
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 184
ttggtacaaa tgaatctggc tgaaccc 27
<210> 185
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 185
tgcttcgttc aatggataaa cacggc 26
<210> 186
<211> 26
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 186
tggctcctaa tgctaatgca atgcgg 26
<210> 187
<211> 27
<212> ДНК
<213> Искусственная последовательность
<220>
<223> синтетический олигонуклеотид
<400> 187
ccgcattgca ttagcattag gagccaa 27
<---