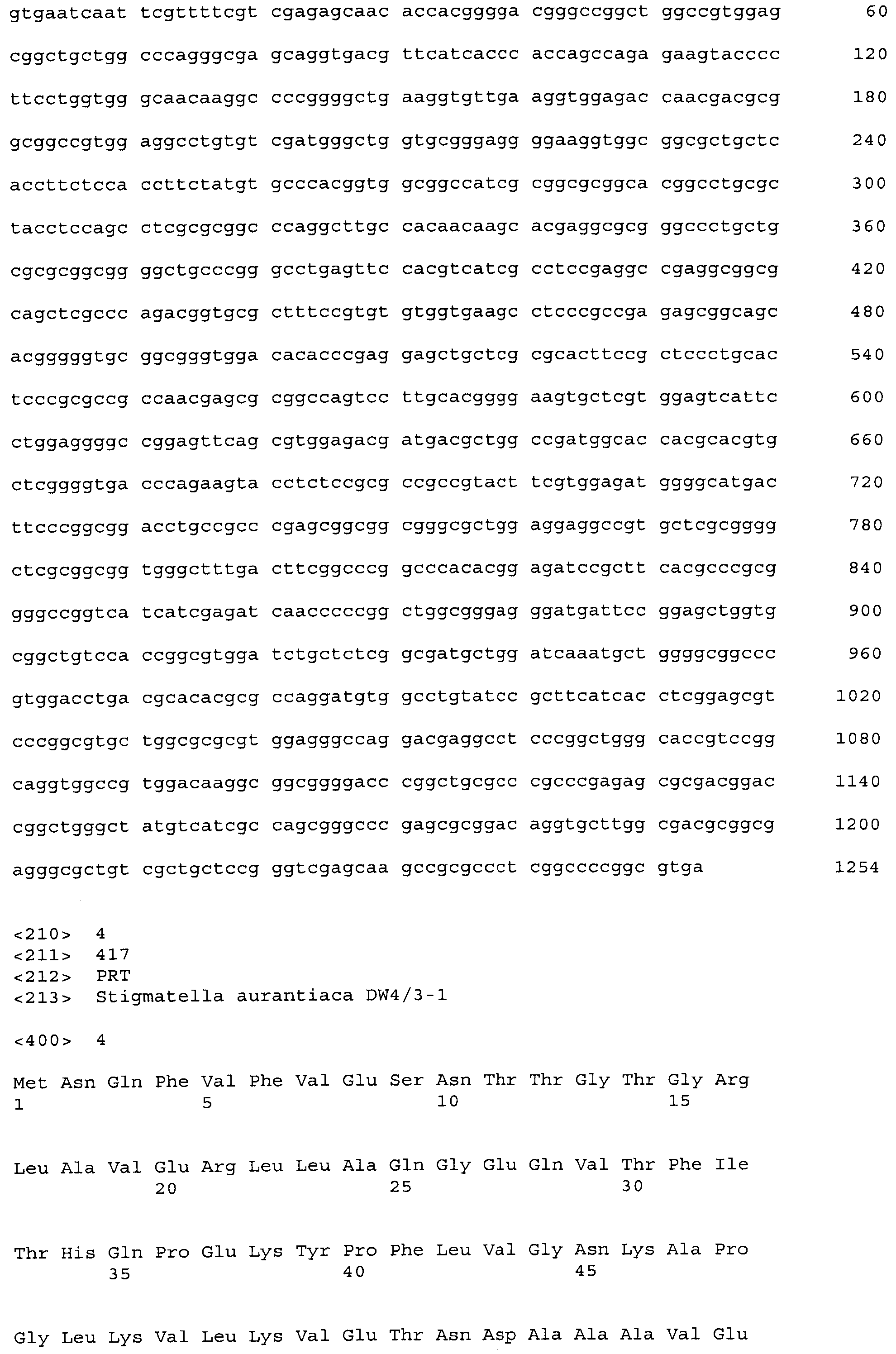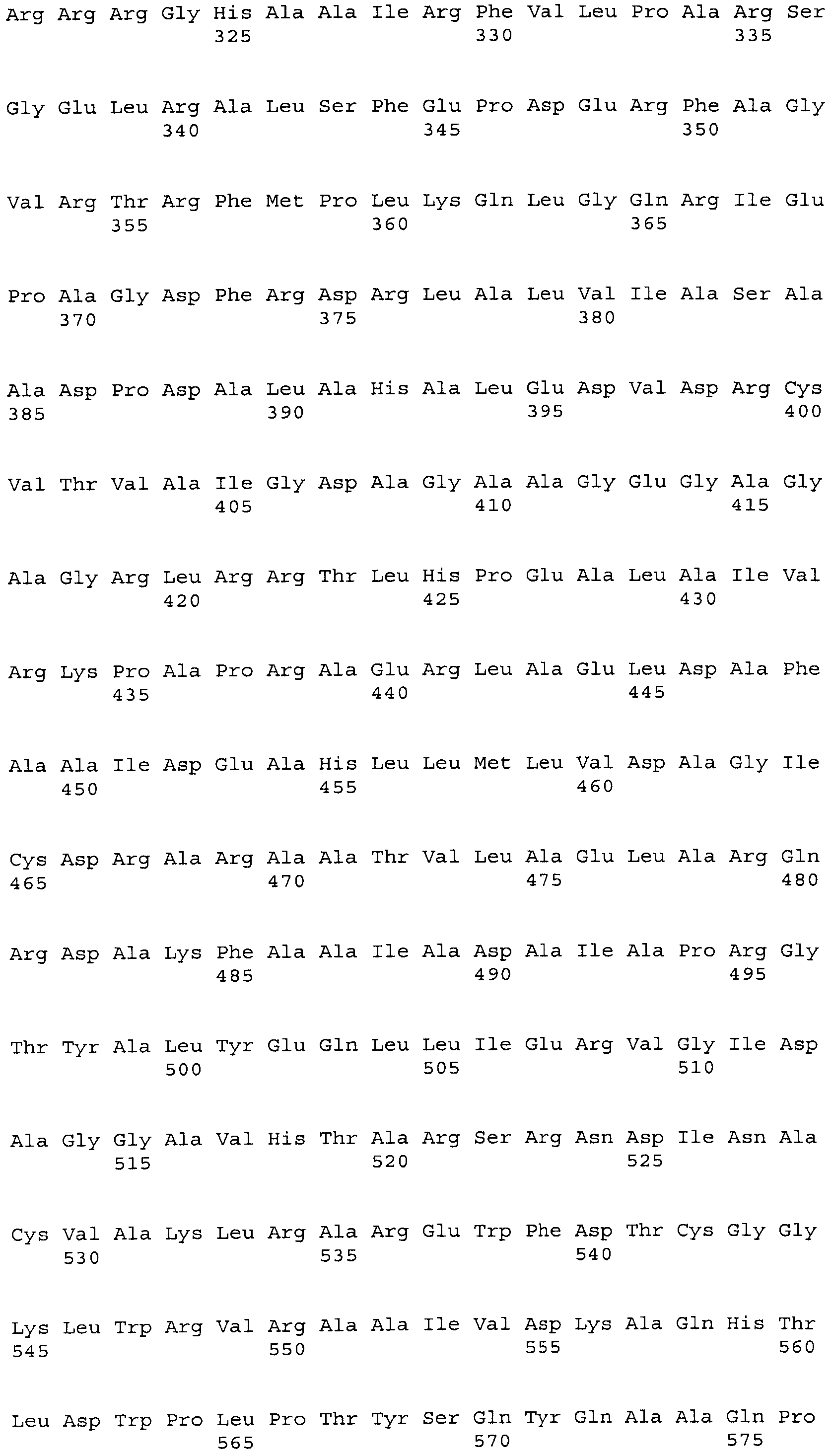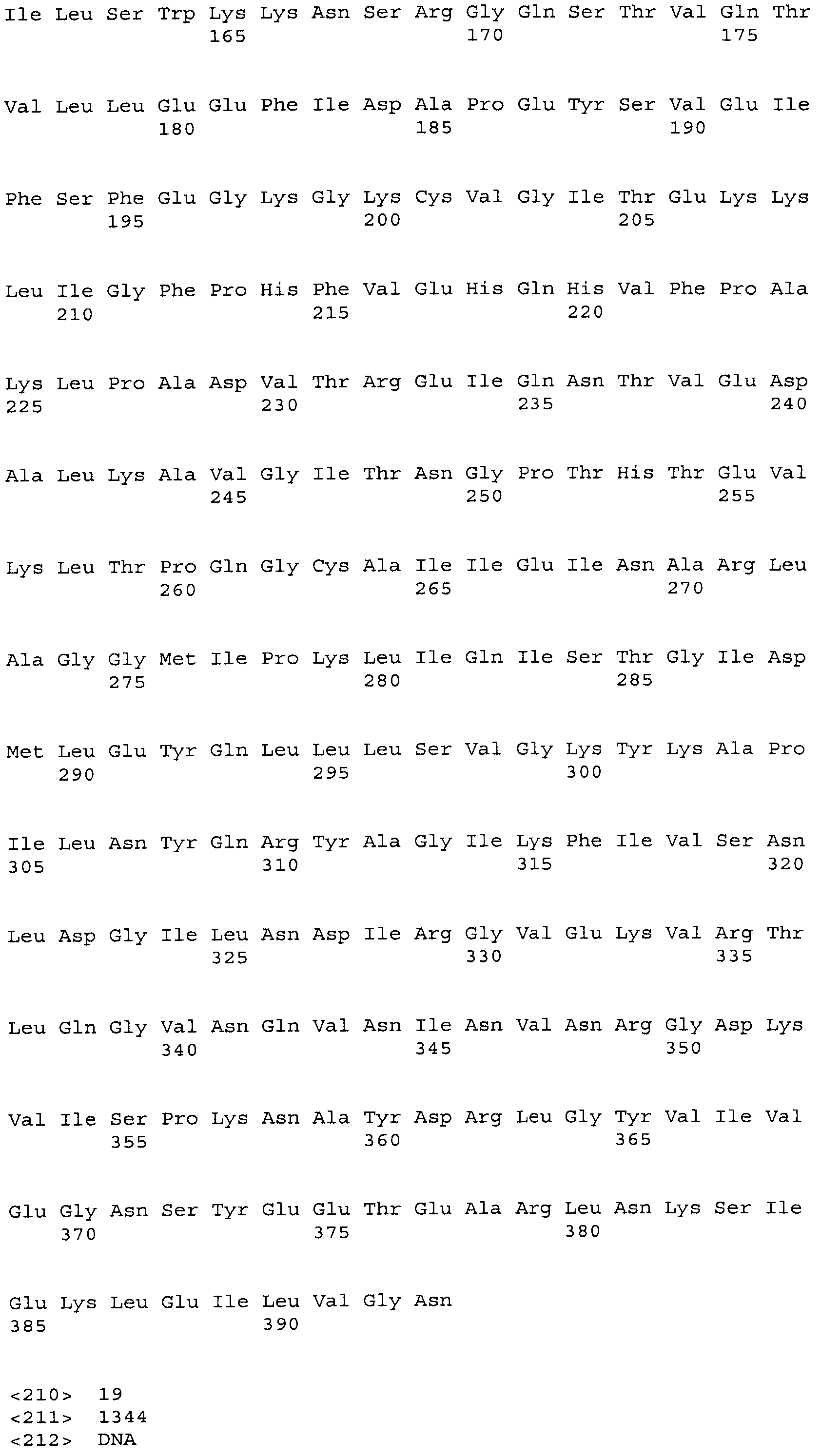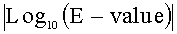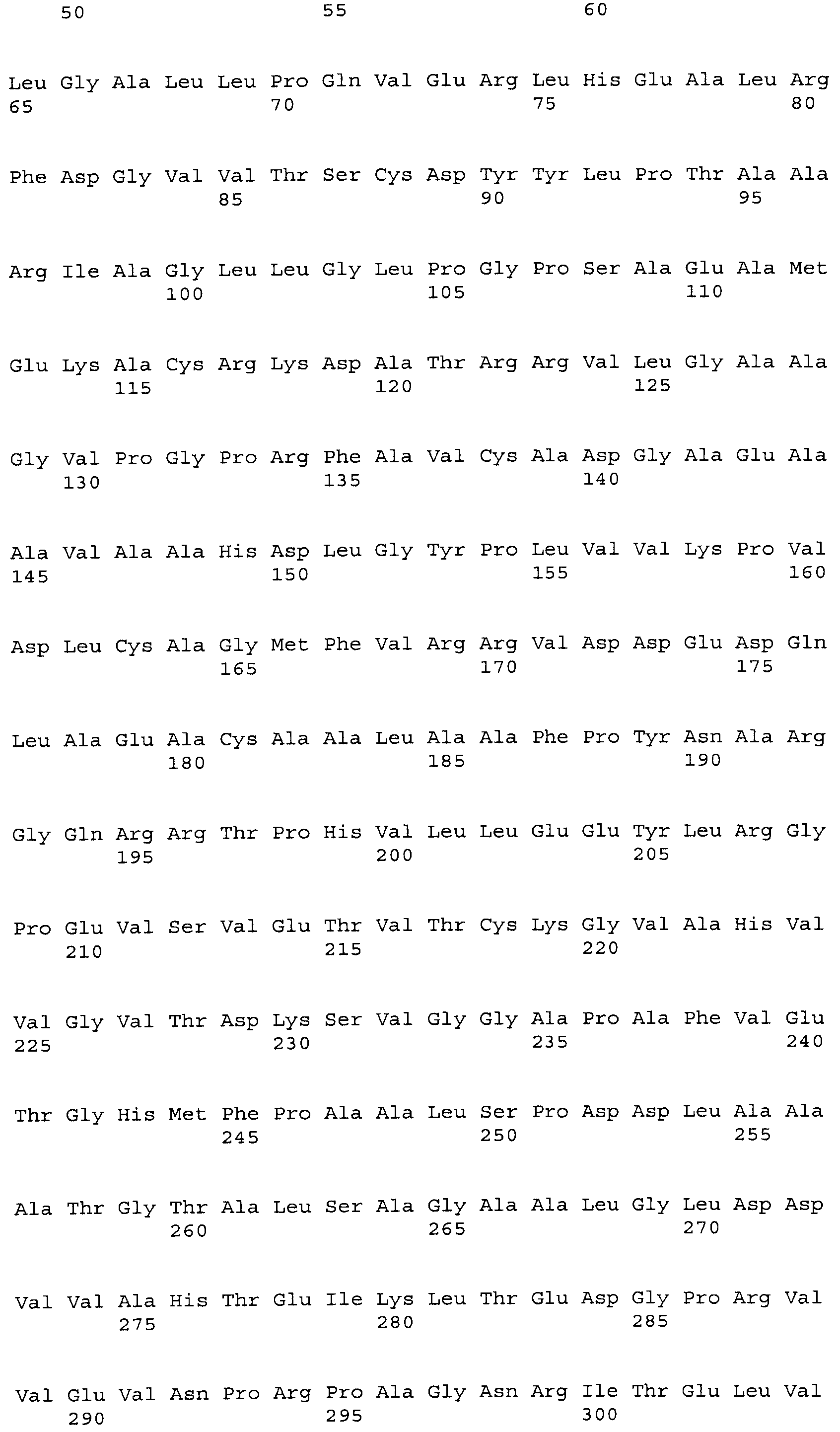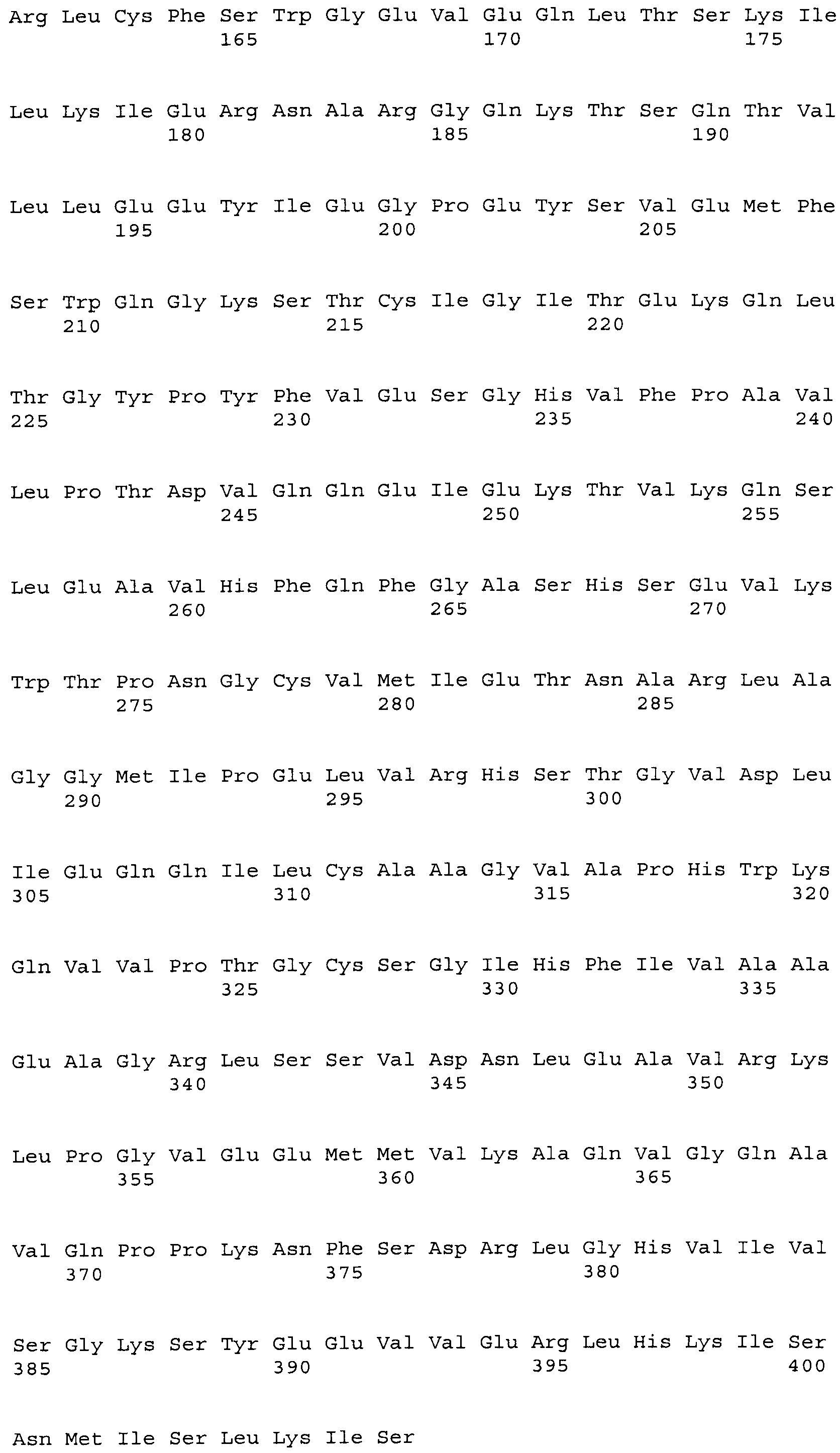Результат интеллектуальной деятельности: ДНК, КОДИРУЮЩАЯ ДИПЕПТИД-СИНТЕЗИРУЮЩИЙ ФЕРМЕНТ (ВАРИАНТЫ), БАКТЕРИЯ РОДА Escherichia И СПОСОБ ПОЛУЧЕНИЯ ДИПЕПТИДОВ С ЕЕ ИСПОЛЬЗОВАНИЕМ
Вид РИД
Изобретение
Область техники, к которой относится изобретение
Настоящее изобретение относится к биотехнологической промышленности, более точно - к новым дипептид-синтезирующим ферментам и способам получения дипептидов, в частности дипептидов, имеющих кислые L-аминокислотные остатки на N-конце.
Уровень техники
Дипептиды используются в фармакологии, пищевой промышленности и в других различных областях. Например, дипептид Asp-Glu использовался для приготовления диуретической и натрийуретической фармацевтической композиции (FR2662359 А1). Известна фармацевтическая композиция, содержащая дипептиды, имеющие агонистическое влияние на подтипы NR1/NR2A и NR1/NR2B рецептора NMDA (JP 2009209131 А). Были изучены вкусовые качества многочисленных дипептидов. Например, дипептид Asp-Val имеет кислый вкус (Sogame S. and Matsushita I., New Food Ind., 1996, 38(12):44-49 (Japanese)). Известен прекрасный усилитель вкуса соли, который получается при использовании содержащего глутаминовую кислоту дипептида, такого как Glu-Ala, Glu-Asp, Glu-Glu, Glu-Ile, Asp-Glu, His-Glu, Trp-Glu и т.д. (WO 2009113563 А1).
Известны различные способы получения дипептидов, включающие экстракцию из белковых гидролизатов, химический синтез из защищенных и/или активированных аминокислот и ферментативные способы синтеза с участием пептидаз и защищенных аминокислот (Akabori S. et al., Bull. Chem. Soc. Japan, 1961, 34:739; Monter В. et al., Biotechnol. Appl. Biochem., 1991, 14(2):183-191). Была описана генетическая конструкция, кодирующая пептид, включающий повторяющуюся аминокислотную последовательность (Asp-Phe)n, подходящую для получения бензилированных и метилированных производных дипептида Asp-Phe (Европейская патентная заявка No. 0036258).
Получение дипептидов с использованием химических и/или химико-ферментативных подходов требует введение и удаление защитных групп для функциональных групп реагирующих аминокислот и выделение требуемого продукта из рацемической смеси. Таким образом, описанные способы получения дипептидов считаются невыгодными с точки зрения цены, эффективности и необходимости утилизации сопутствующих химических реагентов, таких как органические растворители, соли и подобные им.
Было описано несколько подходов ферментативного получения дипептидов и их производных, которые включают способ, использующий обратимую реакцию пролиниминопептидазы, обладающей способностью продуцировать пептиды из L-аминокислот и их сложных эфиров (патент РФ №2279440); способ, использующий нерибосомальную пептидсинтетазу (NRPS) (патенты США №№5,795,738 и 5,652,116; Doekel S. и Marahiel M.A., Chem. Biol., 2000, 7:373-384; Dieckmann R. et al., FEBS Lett., 2001, 498:42-45); способ, использующий аминоацил-m-PHK-синтетазу (патенты Японии №№58-146539 (1983), 58-209992 (1983), и 59-106298 (1984)); и способ, использующий мутантный белок, имеющий пептид-синтезирующую активность (Российская патентная заявка 2007127719).
Ферменты, принадлежащие к суперсемейству АТФ-зависимых карбоксилат-амин/тиол-α-лигаз, широко использовались для получения дипептидов, имеющих α-пептидную связь между двумя L-аминокислотами. Например, при использовании функции поиска по гомологии в SubtiList (http://genolist.pasteur.fr/SubtiList/), являющемся базой данных геномной ДНК Bacillus subtilis 168, и аминокислотную последовательность гена D-Ala-D-Ala-лигазы из Bacillus subtilis 168, был обнаружен ген ywfE, который кодирует фермент, способный синтезировать дипептиды, имеющие на N-конце L-аминокислоту, такую как, в частности, L-Ala, L-Gly, L-Met, L-Ser и L-Thr (Tabata K. et al., J. Bacteriol., 2005, 187(15):5195-5202; патенты США №7,514,243 и №7,939,302). Несмотря на то что белок YwfE (бацилизинсинтетаза, классификационный номер фермента (ЕС) 6.3.2.28) имеет экстремально широкую субстратную специфичность, фермент не связывает аминокислоты, обладающие высоким зарядом, такие как L-Lys, L-Arg, L-Glu и L-Asp, и вторичные амины, такие как L-Pro (Tabata K. et al., J. Bacteriol., 2005, 187(15):5195-5202). Описан белок, кодируемый геном ризоктицинсинтетазы и имеющий дипептид-синтезирующую активность, который утилизирует L-аминокислоты Gly и β-Ala как субстраты (патент США №7,939,294). Как подтверждается выделившейся фосфорной кислотой (Pi), а также методами времяпролетной масс-спектроскопии (TOFMS) и ЯМР, фермент помещает L-Arg и L-Lys на N-конец дипептида. Анализ, основанный на Hidden Markov Model (НММ)-профиле, позволил обнаружить пять L-аминокислота-α-лигаз, происходящих из Treponema denticola АТСС 35405, Photorhabdus luminescence subsp. laumondii TTO1, Streptococcus mutants UA159, Streptococcus pneumoniae TIGR4 и Actinobacillus pleuropneumoniae серологический вариант 1 str. 4074, способных к образованию из L-аминокислот различных пептидных соединений, как подтверждается отщеплением фосфорной кислоты (Senoo A. et al., Biosci. Biotechnol. Biochem., 2010, 74(2):415-418). Ни одно дипептидное образование не было подтверждено в комбинации L-Glu или L-Asp с другими L-аминокислотами. Мутантный белок, имеющий пептид-синтезирующую активность, как было подтверждено методом высокоэффективной жидкостной хроматографии (ВЭЖХ) с использованием стандартных образцов, образует дипептиды, несущие L-Met на N-конце (Российская патентная заявка №2007127719). Компьютерный скрининг, выполненный с помощью сервиса BLAST в NCBI (http://www.ncbi.nlm.nih.gov/BLAST/) и основанный на аминокислотной последовательности Lal из В.subtilis (BsLal) обнаружил белок RSp1486a из Ralstonia solanacearum, который способен образовывать дипептидную связь, как подтверждено отщеплением фосфорной кислоты (Kino K. et al., Biochem. Biophys. Res. Comm., 2008, 371:536-540; Европейская патентная заявка №1870454). Структурный анализ с использованием метода ЯМР подтвердил образование дипептидов, имеющих L-Ser, L-Met, L-Gln, L-Phe, L-His, L-Ala и L-Cys на N-конце. Несмотря на то что неорганический фосфат отщеплялся, как было доказано, в смеси, содержащей RSp1486a и L-Asp с L-Phe, L-His, L-Met, L-Cys или L-Ala; или RSp1486a и L-Glu с L-Phe, L-His, L-Met, L-Cys, L-Ser или L-Ala, структурный анализ продуктов реакции не проводился. Никакого дополнительного отщепления фосфорной кислоты сверх фонового уровня в реакционной смеси, содержащей RSp1486a и L-Asp или L-Glu, не наблюдалось. Было обнаружено, что новая открытая L-аминокислоталигаза RizB из В.subtilis NBRC3134 синтезирует различные гетеропептиды и гомоолигомеры аминокислот с разветвленной цепью, состоящие из 2-5 аминокислотных остатков (Kino K., Yakugaku Zasshi, 2010, 130(11):1463-1469). Например, методом масс-спектроскопии (LC-ESI-MS анализ) было доказано образование димера, тримера и тетрамера L-Val в смеси, содержащей RizB, L-Val и L-Glu или L-Asp. Гетеропептидов обнаружено не было.
В настоящее время нет данных, описывающих синтез дипептидов, имеющих кислые L-аминокислотные остатки, такие как остатки L-Glu или L-Asp, на N-конце и любую другую L-аминокислоту или ее производное на С-конце, с использованием L-аминокислота-α-лигазы (Lal).
Раскрытие сущности изобретения
Цель настоящего изобретения - предоставление ДНК, кодирующей L-аминокислота-α-лигазу (Lal), способную синтезировать дипептид(ы), содержащий(ие) кислые L-аминокислоты, такие как L-Asp или L-Glu, на N-конце и любую другую L-аминокислоту или ее производное на C-конце, в реакционной смеси, которая содержит высокоэнергетические (макроэргические) молекулы, такие как аденозин 5′-трифосфат (АТФ) или его соль.
Другая цель настоящего изобретения - предоставление бактерии рода Escherichia, принадлежащей к виду Escherichia coli, которая модифицирована таким образом, что она содержит ДНК, кодирующую Lal, как здесь описано.
Другая цель настоящего изобретения - предоставление способа получения дипептидов, имеющих кислые L-аминокислоты, такие как L-Asp или L-Glu, на N-конце и любую другую L-аминокислоту или ее производное на С-конце, в реакционной смеси, которая содержит макроэргические молекулы, такие как аденозин 5′-трифосфат (АТФ) или его соль, с использованием фермента Lal, как описано здесь, или бактерии рода Escherichia, которая модифицирована таким образом, что она содержит ДНК, кодирующую фермент Lal, как здесь описано.
Эти цели были достигнуты благодаря обнаружению новых бактериальных L-аминокислота-α-лигаз (Lals), катализирующих образование дипептидов, имеющих кислые L-аминокислоты, такие как L-Asp или L-Glu, на N-конце.
Цель настоящего изобретения - предоставление ДНК, кодирующей белок, имеющий дипептид-синтезирующую активность, отличающейся тем, что ДНК выбрана из группы, состоящей из:
(A) ДНК, имеющей нуклеотидную последовательность SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 и 17;
(B) ДНК, гибридизующейся в жестких условиях с нуклеотидной последовательностью, комплементарной последовательности, представленной в SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 и 17, отличающейся тем, что указанные жесткие условия включают промывание один или более раз в растворе, содержащем концентрацию солей 1×SSC, 0.1% SDS или 0.1×SSC, 0.1% SDS, при 60°С или 65°С;
(C) ДНК, кодирующей белок, имеющий аминокислотную последовательность SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18;
(D) ДНК, кодирующей вариант белка, имеющего аминокислотную последовательность SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, но которая содержит замену, делецию, вставку или добавление одного или нескольких аминокислотных остатков, и имеющего дипептид-синтезирующую активность в соответствии с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18;
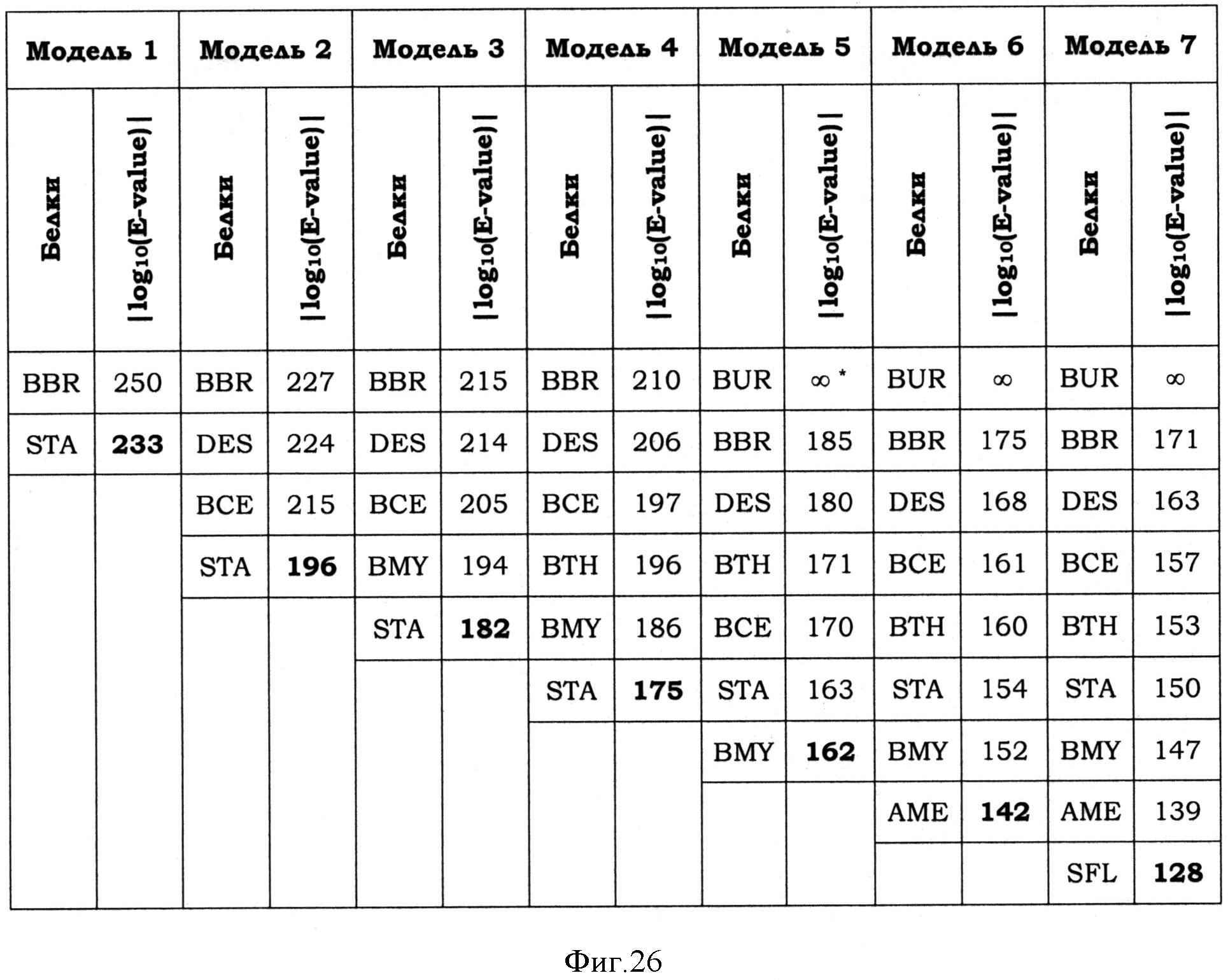
(Е) ДНК, кодирующей белок, имеющий гомологию, определенную через значение |Log10(E-value)|, не менее чем 128, не менее чем 142, не менее чем 162, не менее чем 175, не менее чем 182, не менее чем 196 или не менее чем 233 по сравнению с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18 и имеющий дипептид-синтезирующую активность в соответствии с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18.
Цель настоящего изобретения - предоставление рекомбинантной ДНК для экспрессии ДНК, как описано выше, содержащей ДНК, как описано выше.
Цель настоящего изобретения - предоставление дипептид-продуцирующей бактерии, принадлежащей к роду Escherichia и модифицированной таким образом, чтобы содержать рекомбинантную ДНК, как описано выше.
Другая цель настоящего изобретения - предоставление бактерии, как описано выше, отличающейся тем, что бактерия принадлежит к виду Escherichia coli.
Другая цель настоящего изобретения - предоставление бактерии, как описано выше, отличающейся тем, что бактерия модифицирована таким образом, чтобы иметь ослабленные или инактивированные один или более генов, кодирующих белки, обладающие пептидазой активностью.
Другая цель настоящего изобретения - предоставление бактерии, как описано выше, отличающейся тем, что гены, кодирующие белки, имеющие пептидазную активность, выбраны из группы, состоящей из рерА, рерВ, pepD, pepE, pepP, pepQ, pepN, pepT, iadA, iaaA(ybiK) и dapE.
Цель настоящего изобретения - предоставление белка, имеющего дипептид-синтезирующую активность, отличающегося тем, что указанный белок выбран из группы, состоящей из:
(F) белка, имеющего аминокислотную последовательность SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18;
(G) варианта белка, имеющего аминокислотную последовательность SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, но которая содержит замену, делецию, вставку или добавление одного или нескольких аминокислотных остатков, и имеющего дипептид-синтезирующую активность в соответствии с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18;
(Н) белка, имеющего гомологию, определенную через значениие |Log10(E-value)|, не менее чем 128, не менее чем 142, не менее чем 162, не менее чем 175, не менее чем 182, не менее чем 196 или не менее чем 233 по сравнению с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, и имеющего дипептид-синтезирующую активность в соответствии с аминокислотной последовательностью SEQ ID NOs:2, 4, 6, 8, 10, 12, 14 и 16.
Цель настоящего изобретения - предоставление способа получения белка, как описано выше, включающего:
(a) выращивание бактерии, как описано выше, в питательной среде для продукции указанного белка;
(b) накопление указанного белка в бактерии и/или культуральной жидкости и, если необходимо,
(c) выделение указанного белка из бактерии или культуральной жидкости.
Цель настоящего изобретения - предоставление способа получения дипептида или его соли, включающего:
(a) реакцию L-аминокислот, или производных L-аминокислот, или их солей в приемлемых условиях в присутствии белка, как описано выше;
(b) накопление указанного дипептида или его соли в приемлемом растворителе и, если необходимо,
(c) выделение указанного дипептида или его соли из приемлемого растворителя.
Цель настоящего изобретения - предоставление способа получения дипептида или его соли, включающего:
(a) выращивание бактерии, как описано выше, в питательной среде;
(b) накопление указанного дипептида в бактерии и/или культуральной жидкости и, если необходимо,
(c) выделение указанного дипептида из бактерии или культуральной жидкости.
Цель настоящего изобретения - предоставление способа, как описано выше, отличающегося тем, что указанные L-аминокислоты или их производные выбраны из группы, состоящей из L-аланина, L-аргинина, L-аспарагина, L-аспарагиновой кислоты, L-цистеина, L-глутаминовой кислоты, L-глутамина, глицина, L-гистидина, L-изолейцина, L-лейцина, L-лизина, L-метионина, L-фенилаланина, L-пролина, L-серина, L-треонина, L-триптофана, L-тирозина, L-валина и низшего алкильного эфира L-фенилаланина.
Цель настоящего изобретения - предоставление способа, как описано выше, отличающегося тем, что указанный дипептид представлен формулой
R1-R2,
отличающегося тем, что R1 есть остаток кислой L-аминокислоты или производное остатка кислой L-аминокислоты и R2 есть остаток L-аминокислоты или производное остатка L-аминокислоты, причем указанный остаток L-аминокислоты выбран из группы, состоящей из остатка L-аланина, L-аргинина, L-аспарагина, L-аспарагиновой кислоты, L-цистеина, L-глутаминовой кислоты, L-глутамина, глицина, L-гистидина, L-изолейцина, L-лейцина, L-лизина, L-метионина, L-фенилаланина, L-пролина, L-серина, L-треонина, L-триптофана, L-тирозина, L-валина и низшего алкильного эфира L-фенилаланина.
Цель настоящего изобретения - предоставление способа, как описано выше, отличающегося тем, что R1 есть остаток L-аспарагиновой кислоты или остаток L-глутаминовой кислоты и R2 есть остаток L-глутаминовой кислоты, L-изолейцина, L-фенилаланина, L-триптофана, L-валина или низшего алкильного эфира L-фенилаланина.
Цель настоящего изобретения - предоставление способа, как описано выше, отличающегося тем, что R1 есть остаток L-аспарагиновой кислоты или остаток L-глутаминовой кислоты и R2 есть остаток L-глутаминовой кислоты, L-изолейцина, L-фенилаланина, L-триптофана, L-валина или низшего алкильного эфира L-фенилаланина.
Цель настоящего изобретения - предоставления способа, как описано выше, отличающегося тем, что низший алкильный эфир L-фенилаланина есть метиловый, этиловый или пропиловый эфир L-фенилаланина.
Описание настоящего изобретения приведено ниже.
Краткое описание фигур
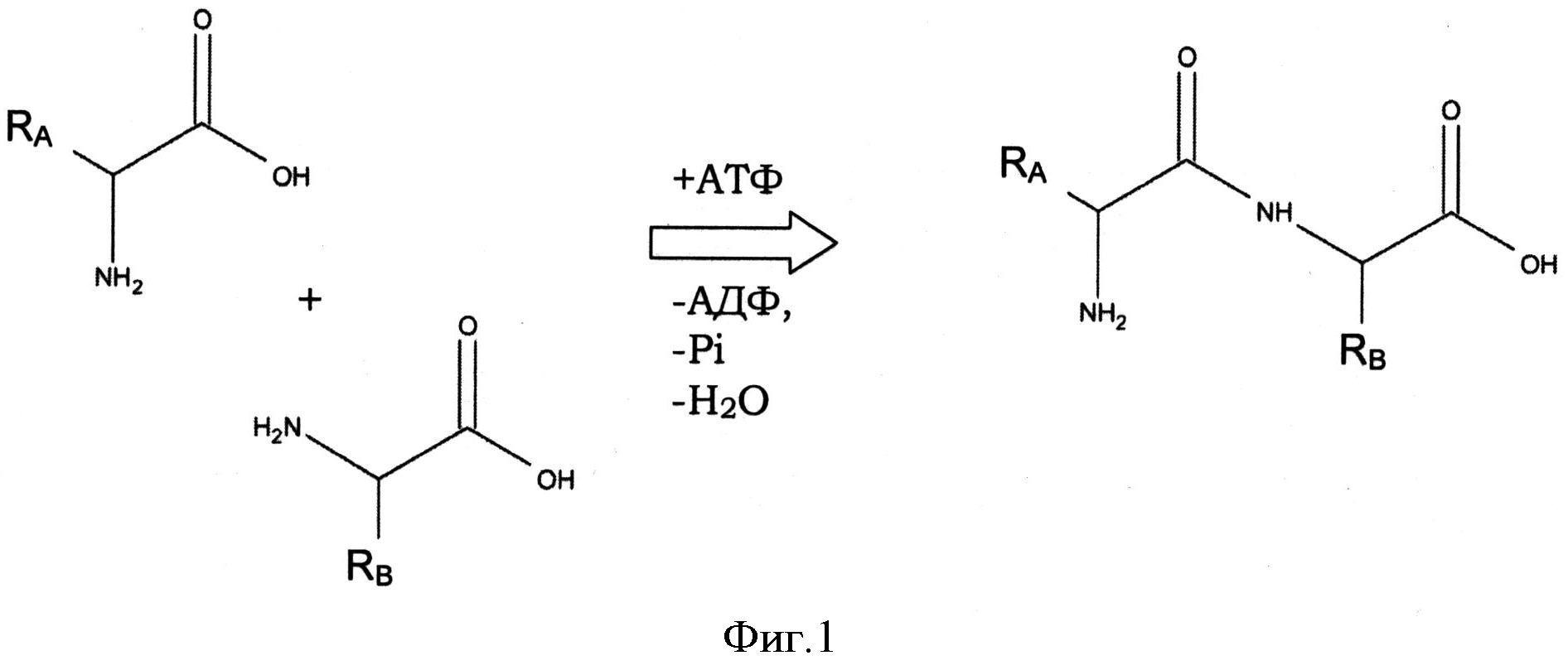
Фигура 1 показывает схему реакции лигирования, катализируемой L-аминокислота-α-лигазами (Lals). RA и RB группы боковой цепи, которые могут быть одного или разных типов. АТФ означает аденозин-5′-трифосфат, АДФ означает аденозин-5′-дифосфат и Pi означает неорганический фосфат, фосфорную кислоту или ее соль.
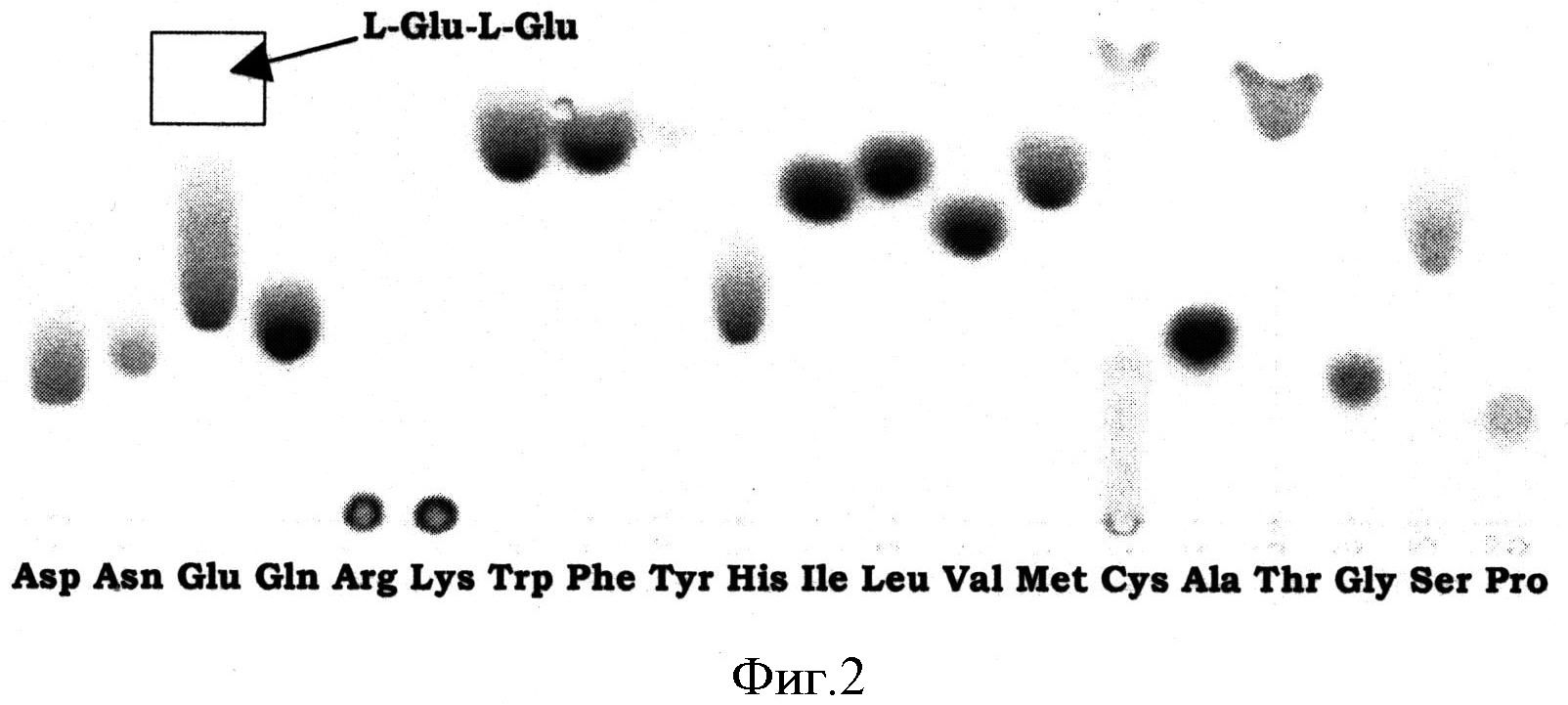
Фигура 2 показывает активность BBR47_51900 в лигировании одинаковых канонических L-аминокислот.
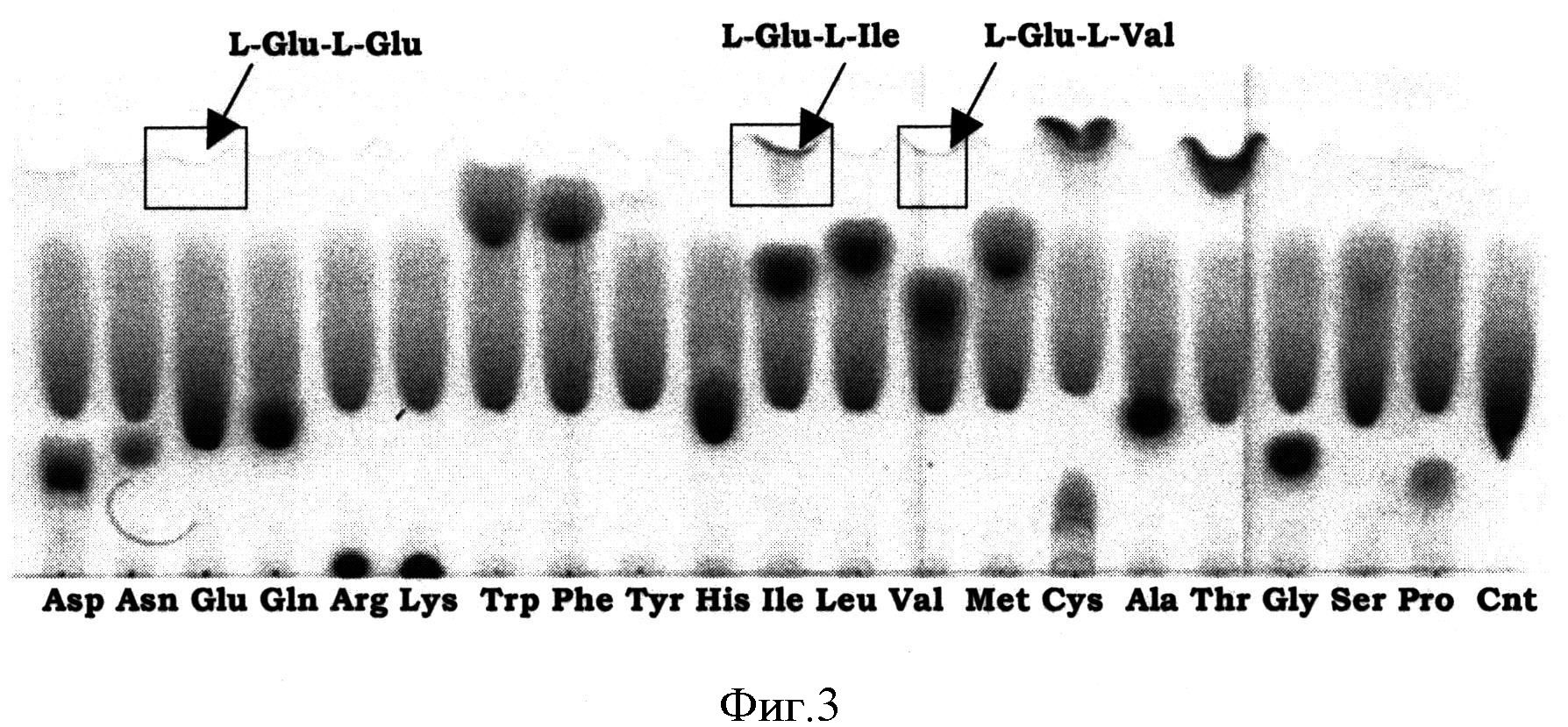
Фигура 3 показывает активность BBR47_51900 в лигировании двух различных канонических L-аминокислот, где одна L-аминокислота есть L-Glu. Cnt: контроль (L-Glu).
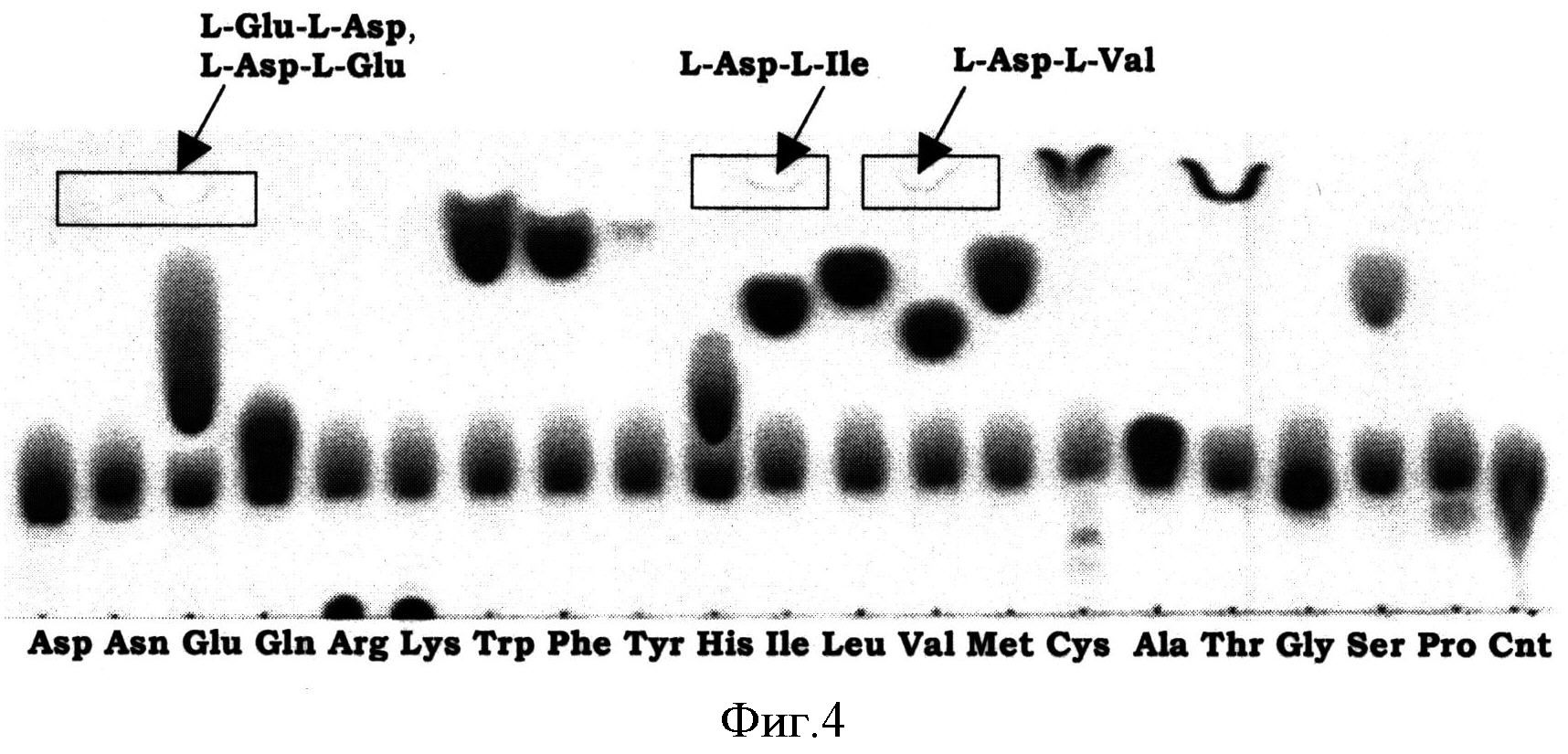
Фигура 4 показывает активность BBR47_51900 в лигировании двух различных канонических L-аминокислот, где одна L-аминокислота есть L-Asp. Cnt: контроль (L-Asp).
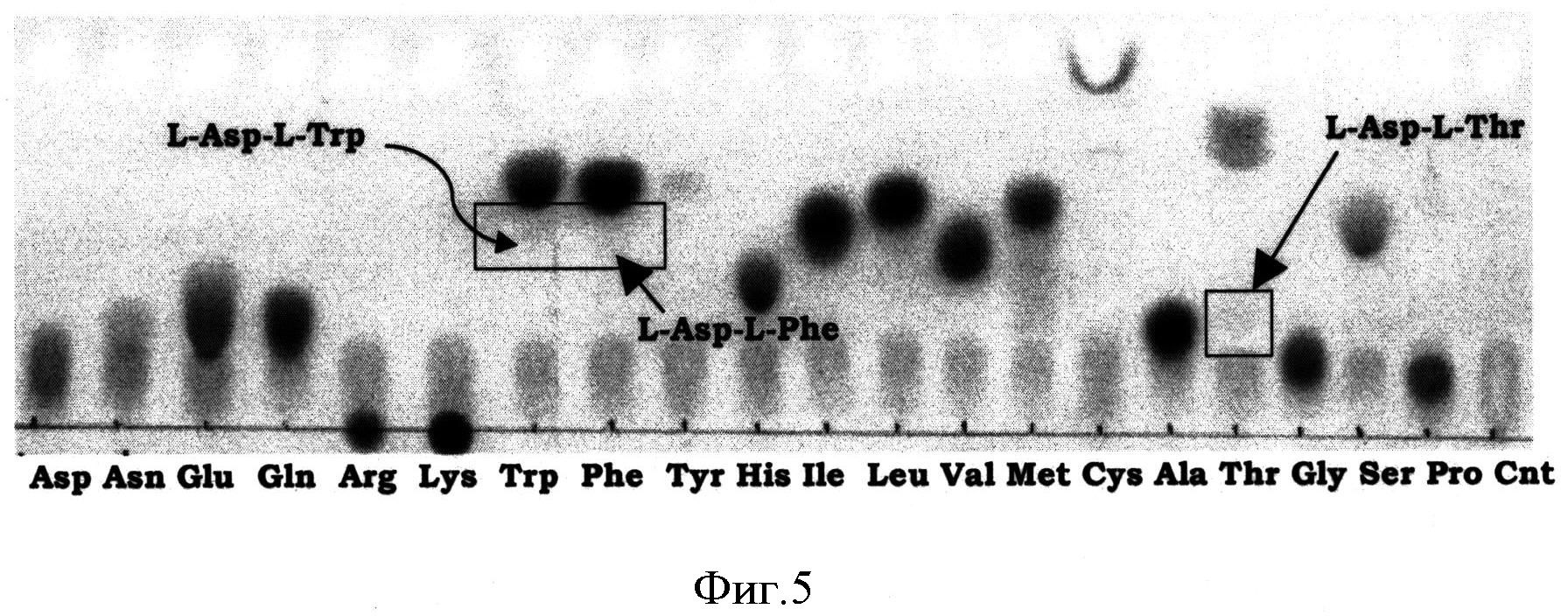
Фигура 5 показывает активность Staur_4851 в лигировании двух различных канонических L-аминокислот, где одна L-аминокислота есть L-Asp.
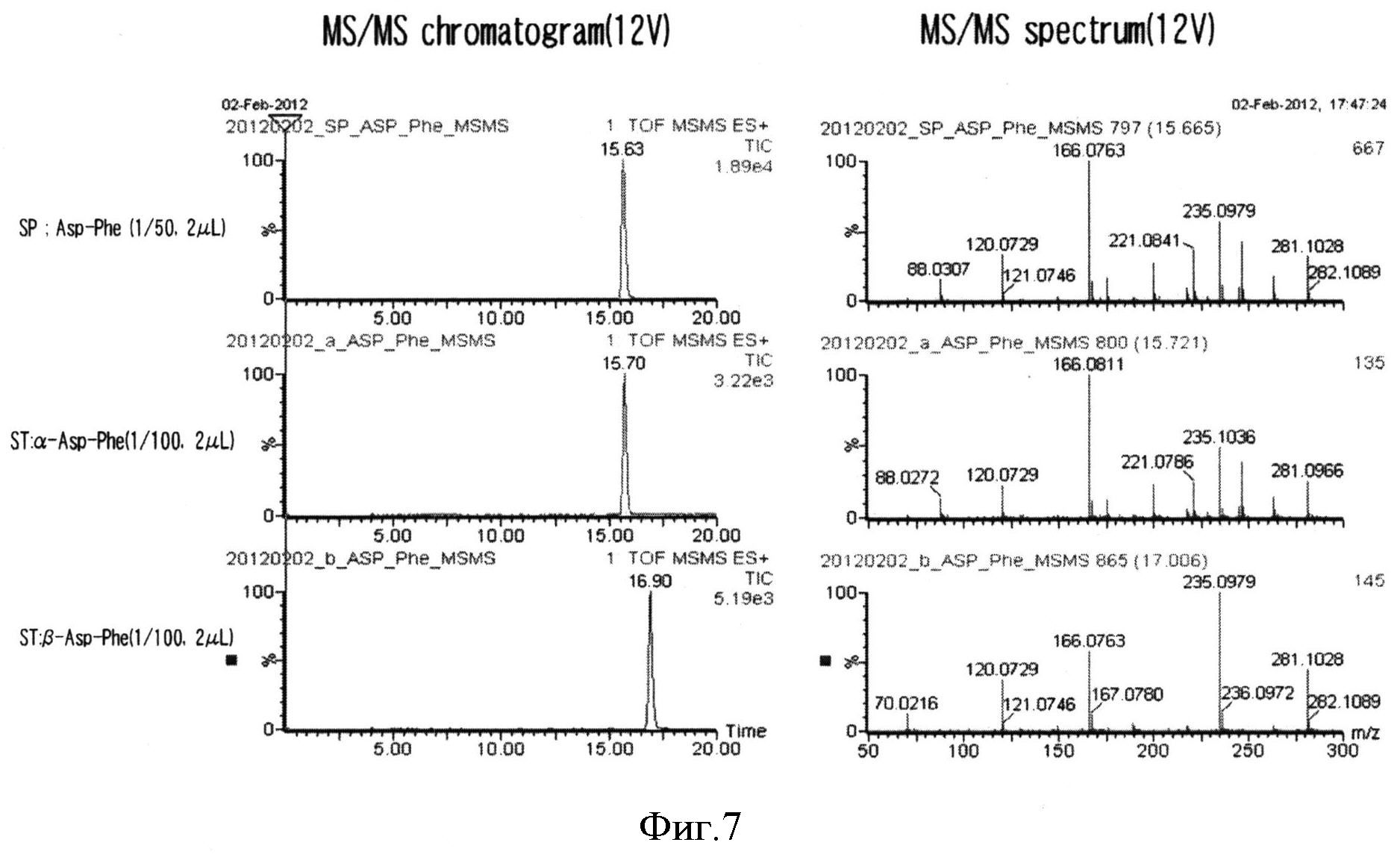
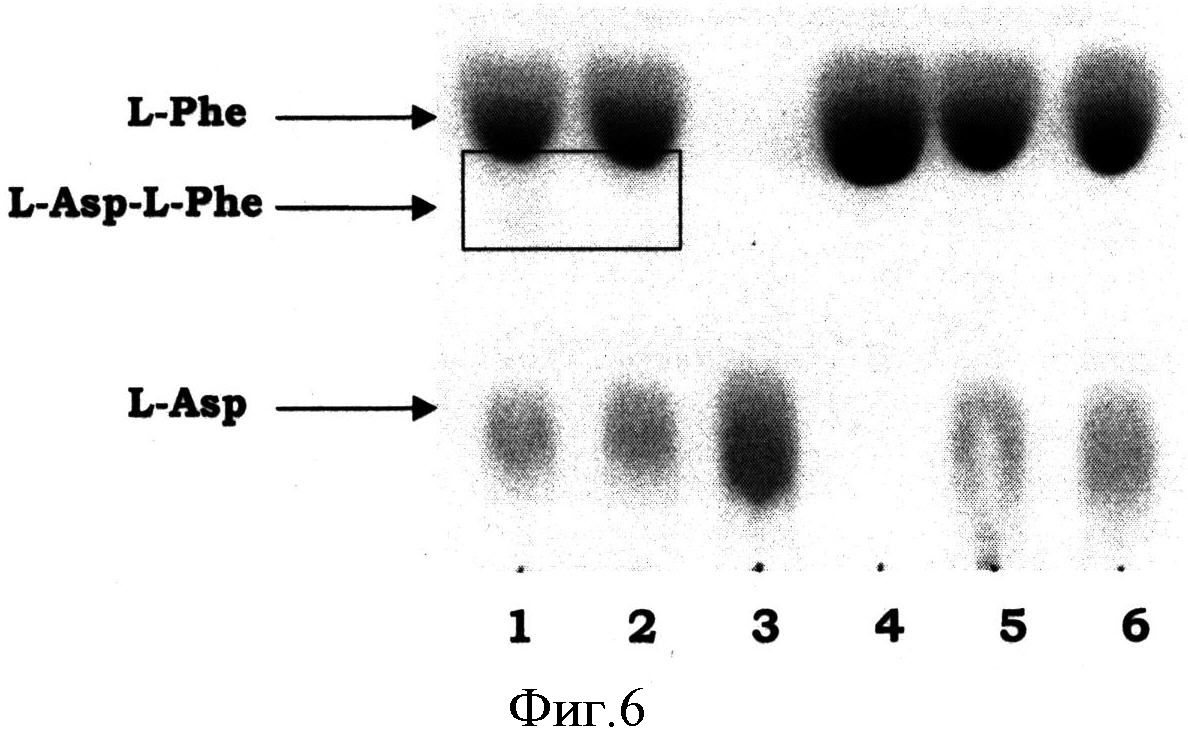
Фигура 6 показывает активность Staur_4851 в лигировании L-Asp и L-Phe, определенную методом ТСХ. 1 - (Трис-HCl рН 9.0 50 мМ, MgCl2 10 мМ, L-Asp 10 мМ, L-Phe 10 мМ, АТФ 10 мМ, Staur_4851 2 мкг); 2 - (Трис-HCl рН 8.0 50 мМ, MgCl2 10 мМ, L-Asp 10 мМ, L-Phe 10 мМ, АТФ 10 мМ, Staur_4851 2 мкг); 3 - (Трис-HCl рН 8.0 50 мМ, MgCl2 10 мМ, L-Asp 20 мМ, L-Phe 0 мМ, АТР 10 мМ, Staur_4851 2 мкг); 4 - (Трис-HCl рН 8.0 50 мМ, MgCl2 10 мМ, L-Asp 0 мМ, L-Phe 20 мМ, АТФ 10 мМ, Staur_4851 2 мкг); 5 - (Трис-HCl рН 8.0 50 мМ, MgCl2 10 мМ, L-Asp 10 мМ, L-Phe 10 мМ, АТФ 0 мМ, Staur_4851 2 мкг); 6 - (Трис-HCl рН 8.0 50 мМ, MgCl2 10 мМ, L-Asp 10 мМ, L-Phe 10 мМ, АТФ 10 мМ, Staur_4851 0 мкг).
Фигура 7 показывает активность BBR47_51900 в лигировании L-Asp и L-Phe, определенную методом LC-QTOF/MS/MS. SP: образец; ST: стандарт (αAsp-Phe и βAsp-Phe).
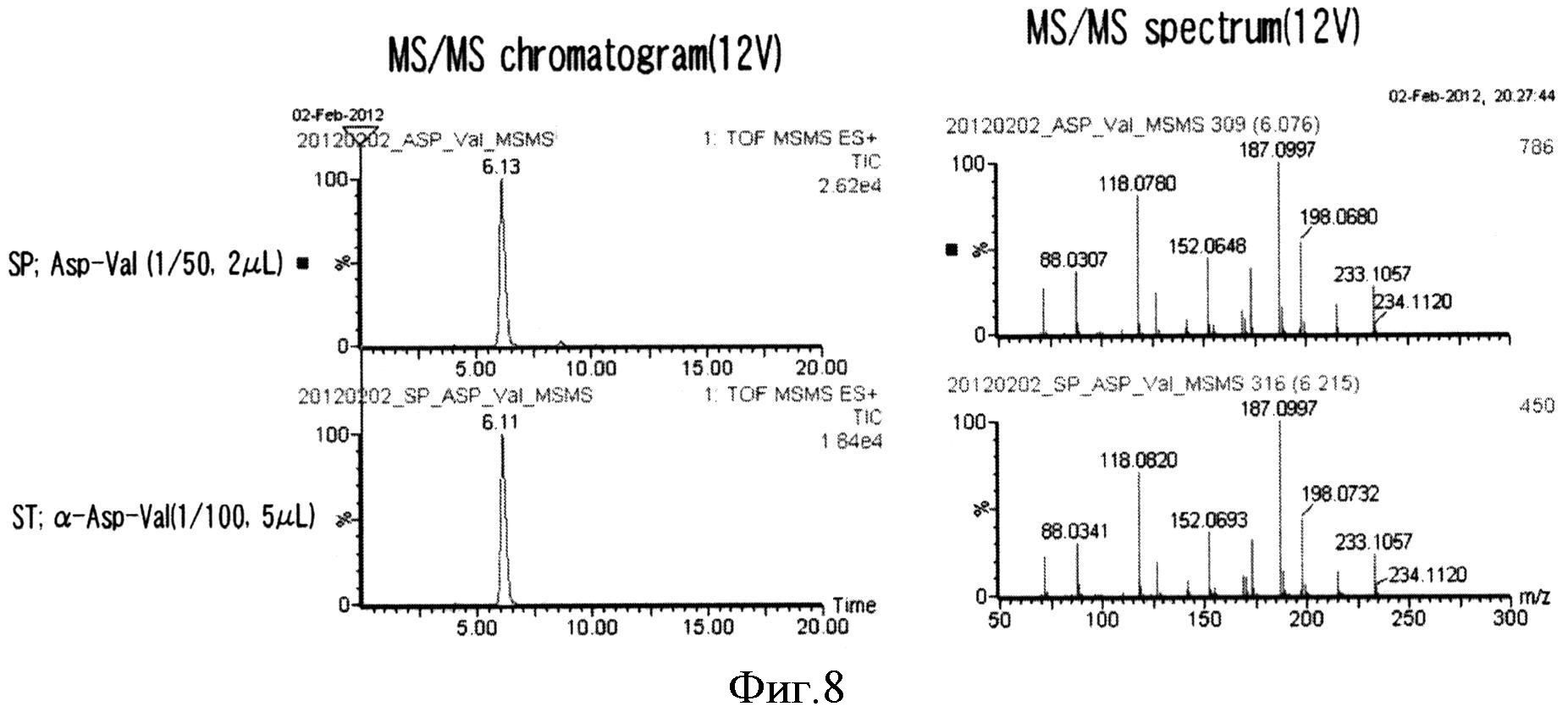
Фигура 8 показывает активность BBR47_51900 в лигировании L-Asp и L-Val, определенную методом LC-QTOF/MS/MS. SP: образец; ST: стандарт (αAsp-Val).
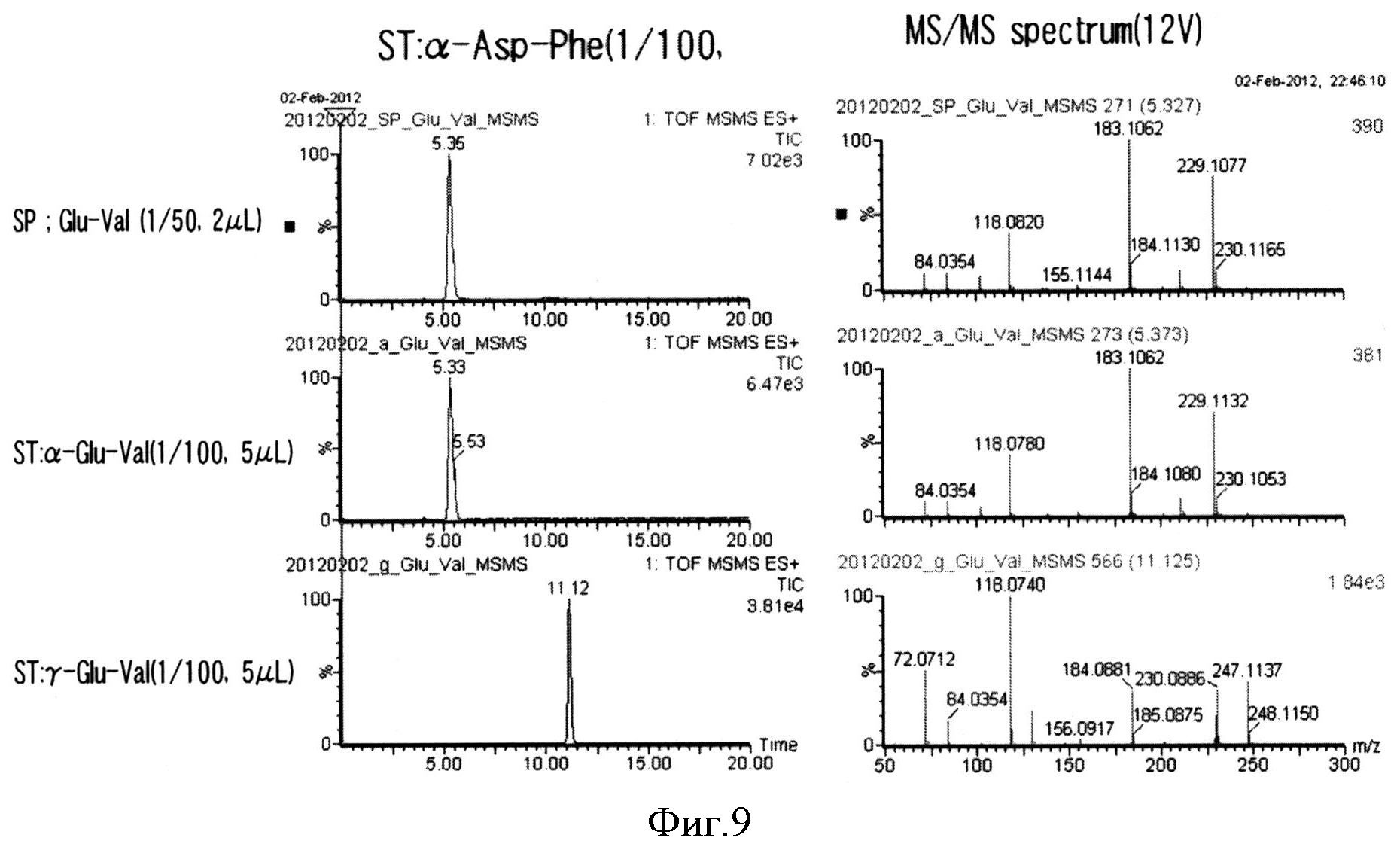
Фигура 9 показывает активность BBR47_51900 в лигировании L-Glu и L-Val, определенную методом LC-QTOF/MS/MS. SP: образец; ST: стандарт (αGlu-Val и γGlu-Val).
Фигура 10 показывает выравнивание BBR47_51900 и Staur_4851 (ClustalW, в формате PIR).
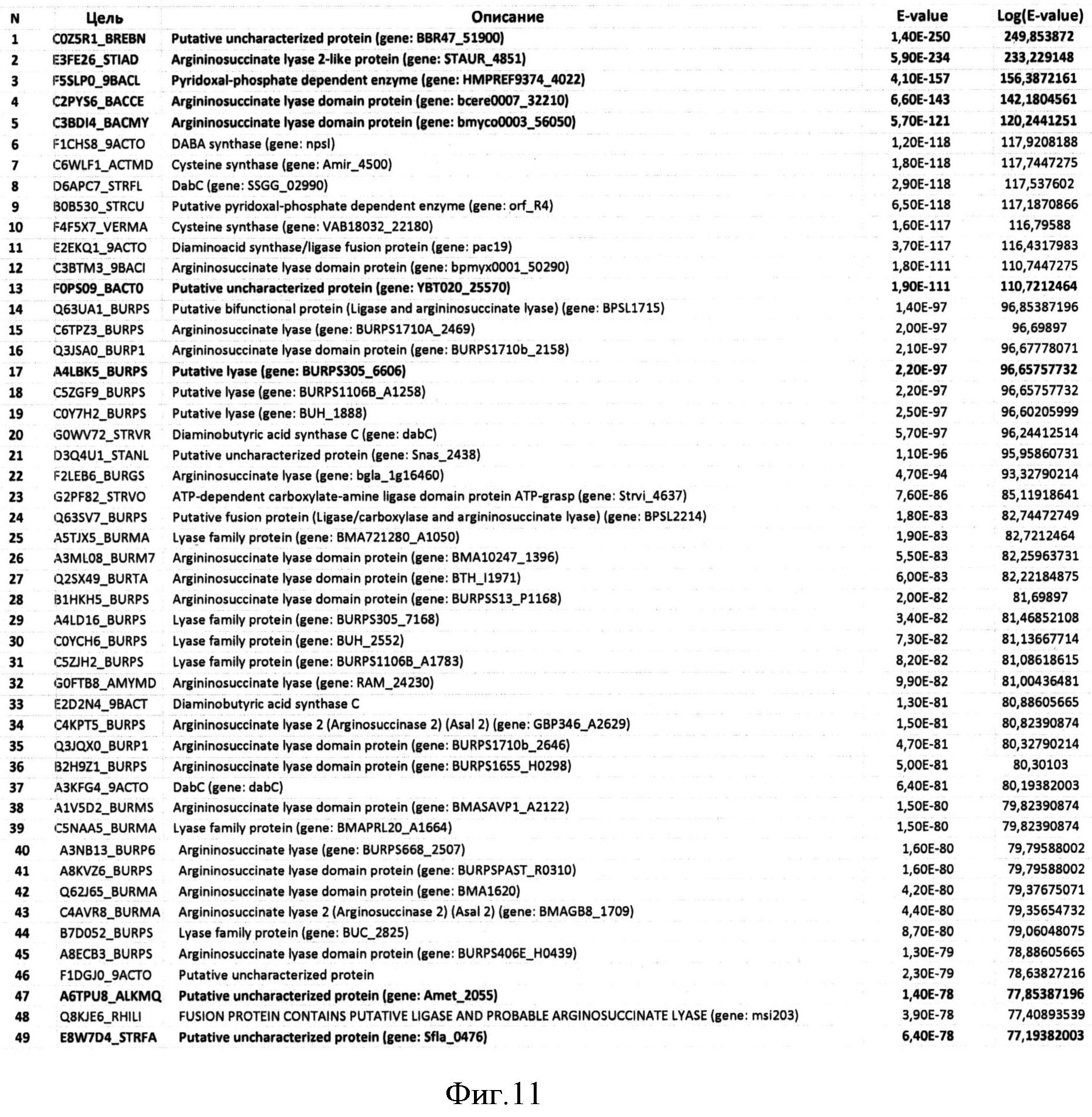
Фигура 11 показывает данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900 и Staur_4851 (представлены первые 49 хитов).
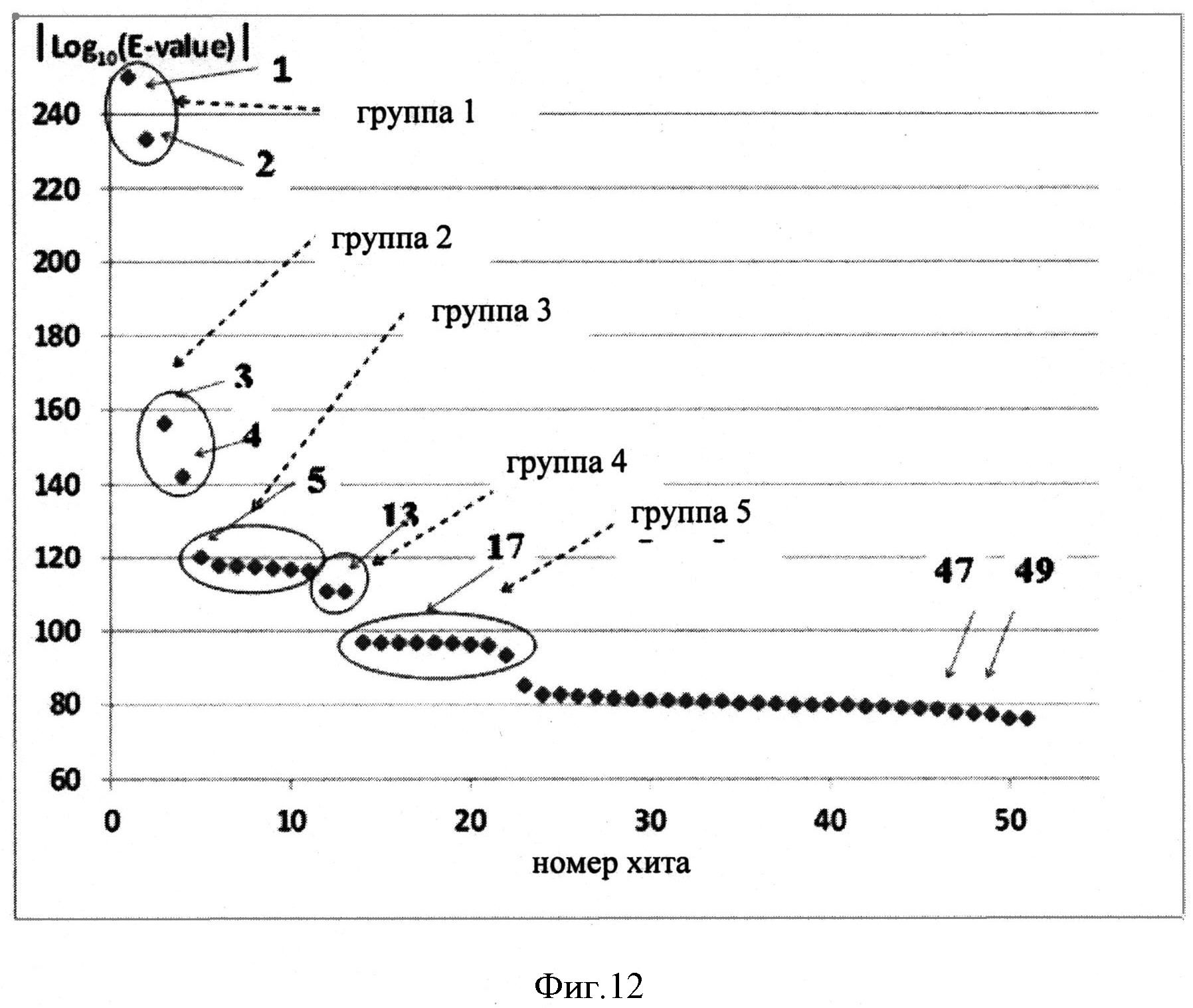
Фигура 12 показывает диаграмму распределения значений |Log10(E-value)|, полученных с помощью программы HMMsearch с использованием выравнивания BBR47_51900 и Staur_4851 (см. Фигуру 10). Следующие хиты отмечены сплошными стрелками: 1 - BBR47_51900, 2 - Staur_4851, 3 - DES, 4 - ВСЕ, 5 - BMY, 13 - BTH, 17 - BUR, 47 - AME, 49 - SFL.
Фигура 13 показывает выравнивание BBR47_51900, Staur_4851, DES и ВСЕ (ClustalW, в формате PIR).
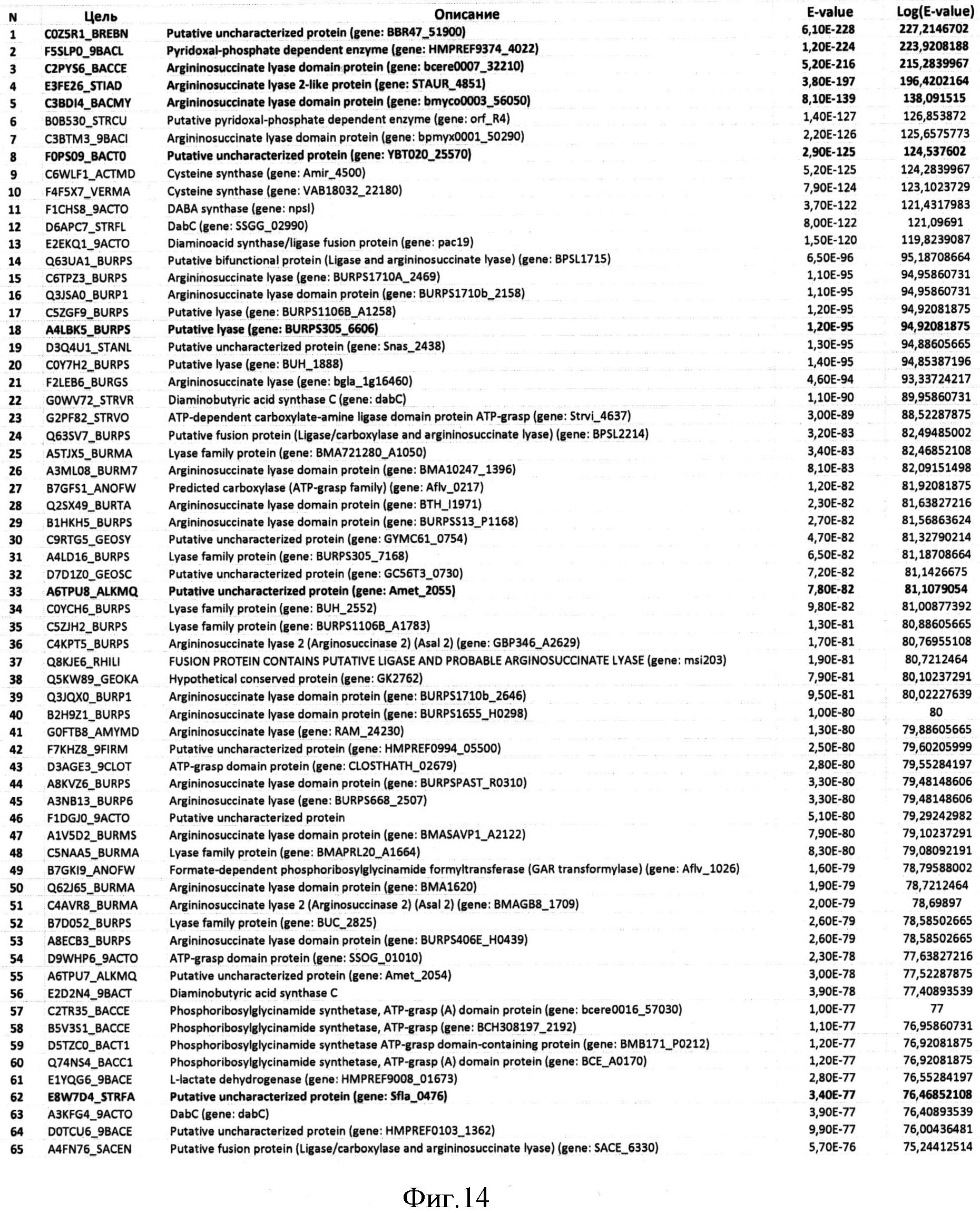
Фигура 14 показывает выходные данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES и ВСЕ (представлены первые 65 хитов).
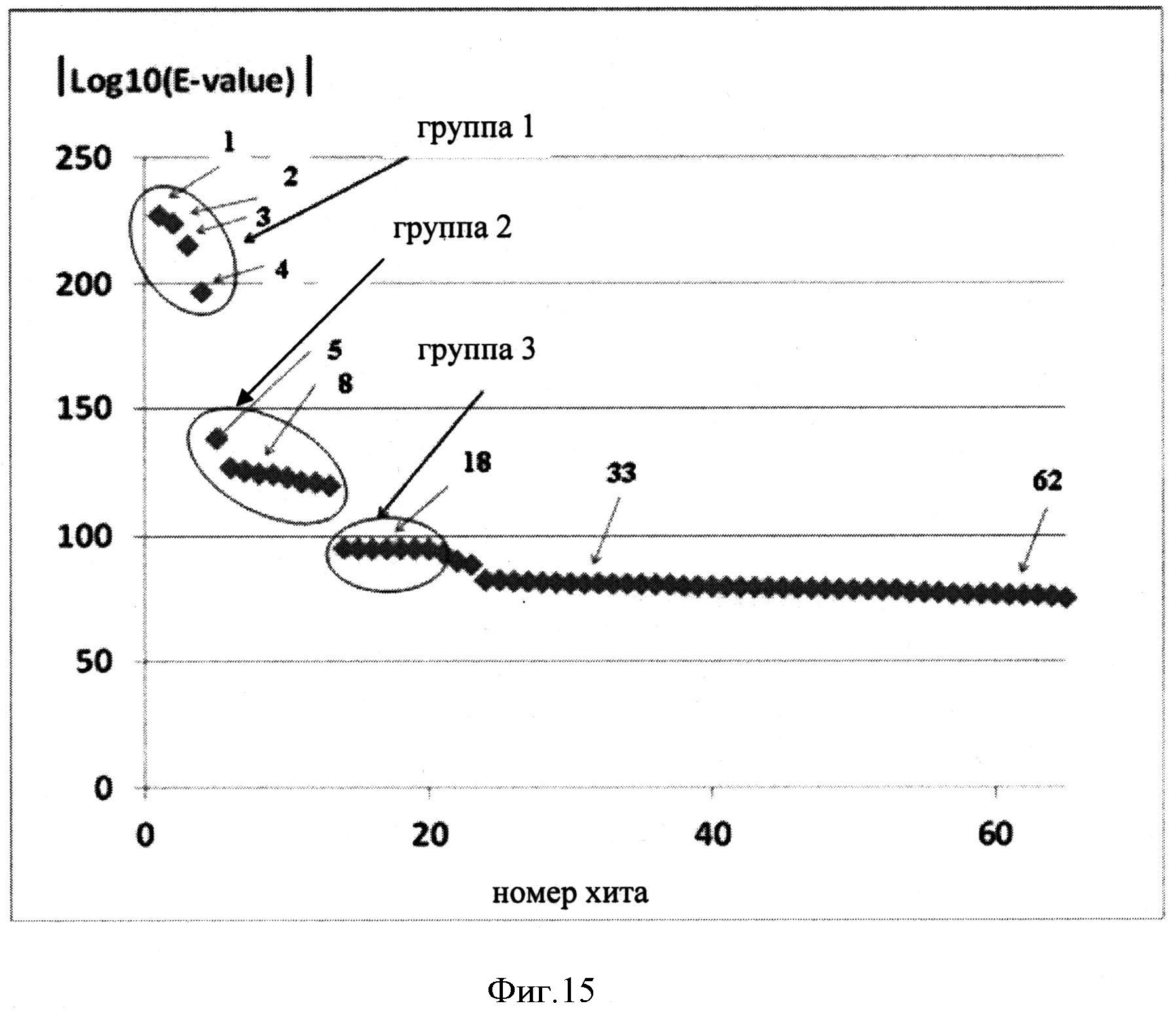
Фигура 15 показывает диаграмму распределения значений |Log10(E-value)|, полученных с помощью программы HMMsearch с использованием выравнивания BBR47_51900, Staur_4851, DES и ВСЕ (см. Фигуру 13). Следующие хиты отмечены сплошными стрелками: 1 - BBR47_51900, 2 - DES, 3 - ВСЕ, 4 - Staur_4851, 5 - BMY, 8 - BTH, 18 - BUR, 33 - AME, 62 - SFL.
Фигура 16-1 показывает выровненные BBR47_51900, Staur_4851 и DES в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ и BMY (ClustalW, в формате PIR).
Фигура 16-2 показывает выровненные ВСЕ и BMY в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ и BMY (ClustalW, в формате PIR).
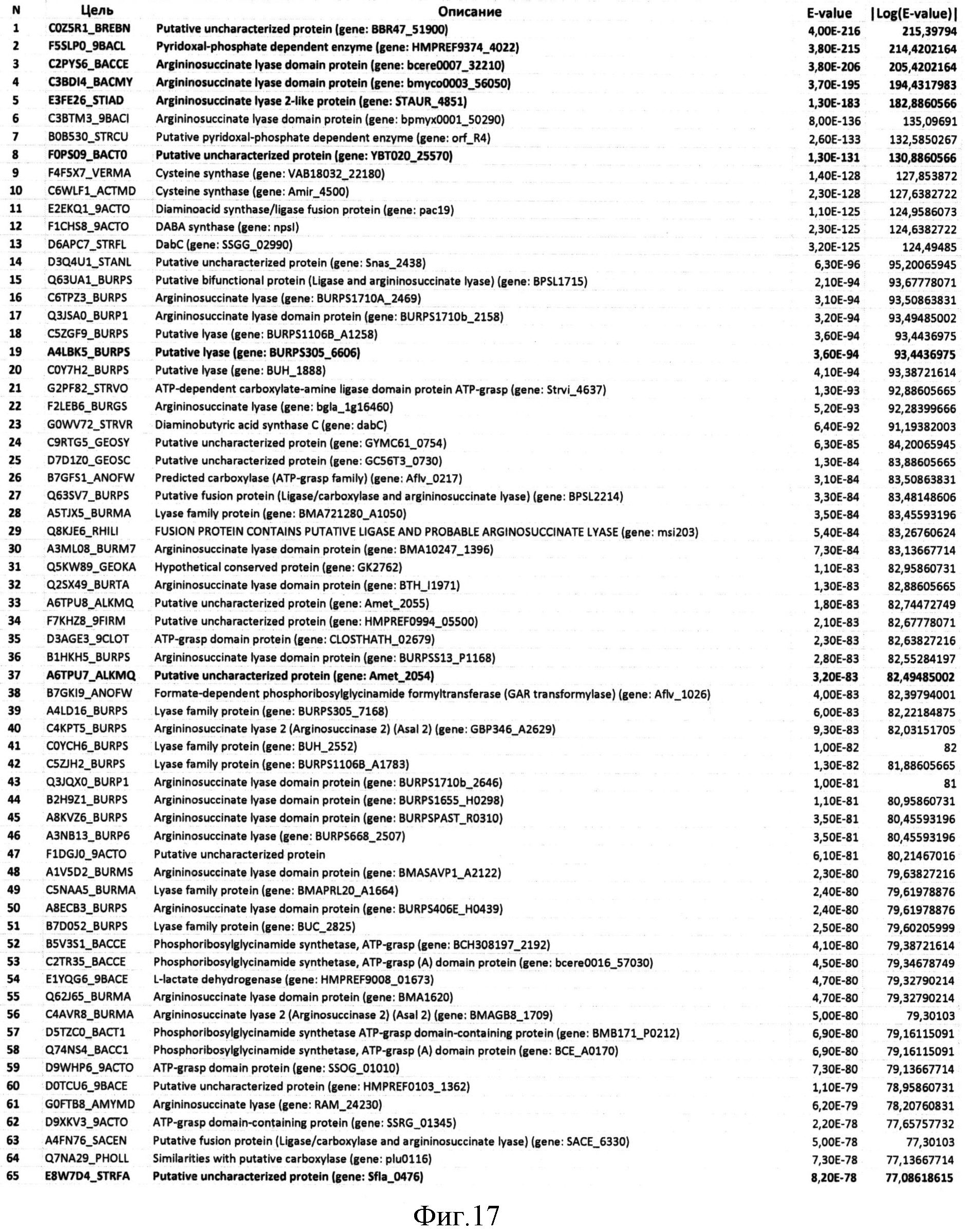
Фигура 17 показывает выходные данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES, ВСЕ и BMY (представлены первые 65 хитов).
Фигура 18-1 показывает выровненные Staur_4851, BBR47_51900 и ВСЕ в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН (ClustalW, в формате PIR).
Фигура 18-2 показывает выровненные DES, BMY и ВТН в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН (ClustalW, в формате PIR).
Фигура 19 показывает данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН (представлены первые 73 хита).
Фигура 20-1 показывает выровненные Staur_4851 и BBR47_51900 в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (ClustalW, в формате PIR).
Фигура 20-2 показывает выровненные ВСЕ и DES в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (ClustalW, в формате PIR).
Фигура 20-3 показывает выровненные BMY и ВТН в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (ClustalW, в формате PIR).
Фигура 20-4 показывает выровненный BUR в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (ClustalW, в формате PIR).
Фигура 21 показывает данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (представлены первые 104 хита).
Фигура 22-1 показывает выровненные Staur_4851 и BBR47_51900 в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR и АМЕ (ClustalW, в формате PIR).
Фигура 22-2 показывает выровненные ВСЕ и DES в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR и АМЕ (ClustalW, в формате PIR).
Фигура 22-3 показывает выровненные BMY и ВТН в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR и АМЕ (ClustalW, в формате PIR).
Фигура 22-4 показывает выровненные BUR и АМЕ в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR и АМЕ (ClustalW, в формате PIR).
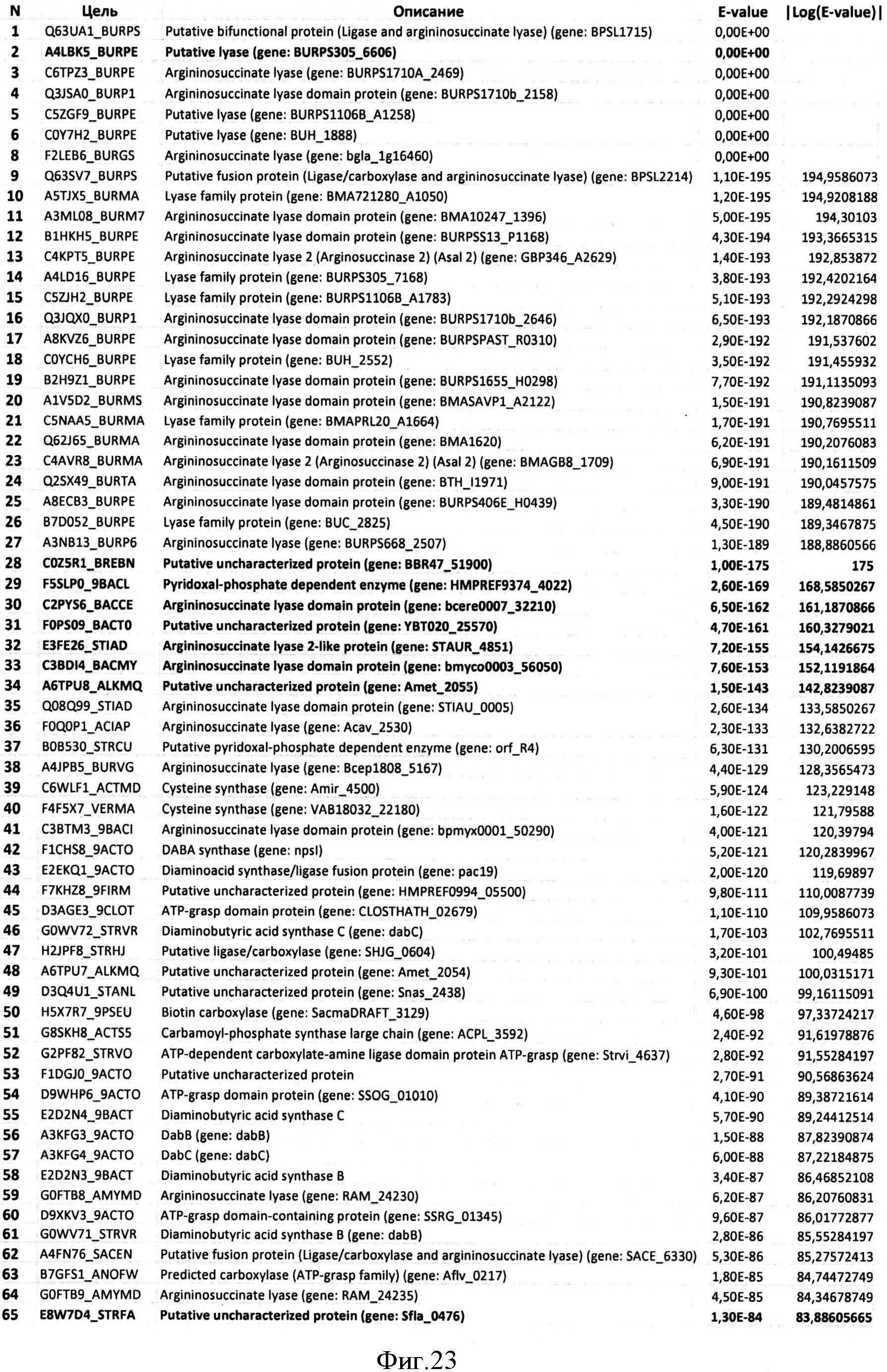
Фигура 23 показывает выходные данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR и АМЕ (представлены первые 65 хитов).
Фигура 24-1 показывает выровненные Staur_4851 и BBR47_51900 в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (ClustalW, в формате PIR).
Фигура 24-2 показывает выровненные ВСЕ и DES в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (ClustalW, в формате PIR).
Фигура 24-3 показывает выровненные BMY и BTH в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (ClustalW, в формате PIR).
Фигура 24-4 показывает выровненные BUR и АМЕ в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (ClustalW, в формате PIR).
Фигура 24-5 показывает выровненный SFL в выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (ClustalW, в формате PIR).
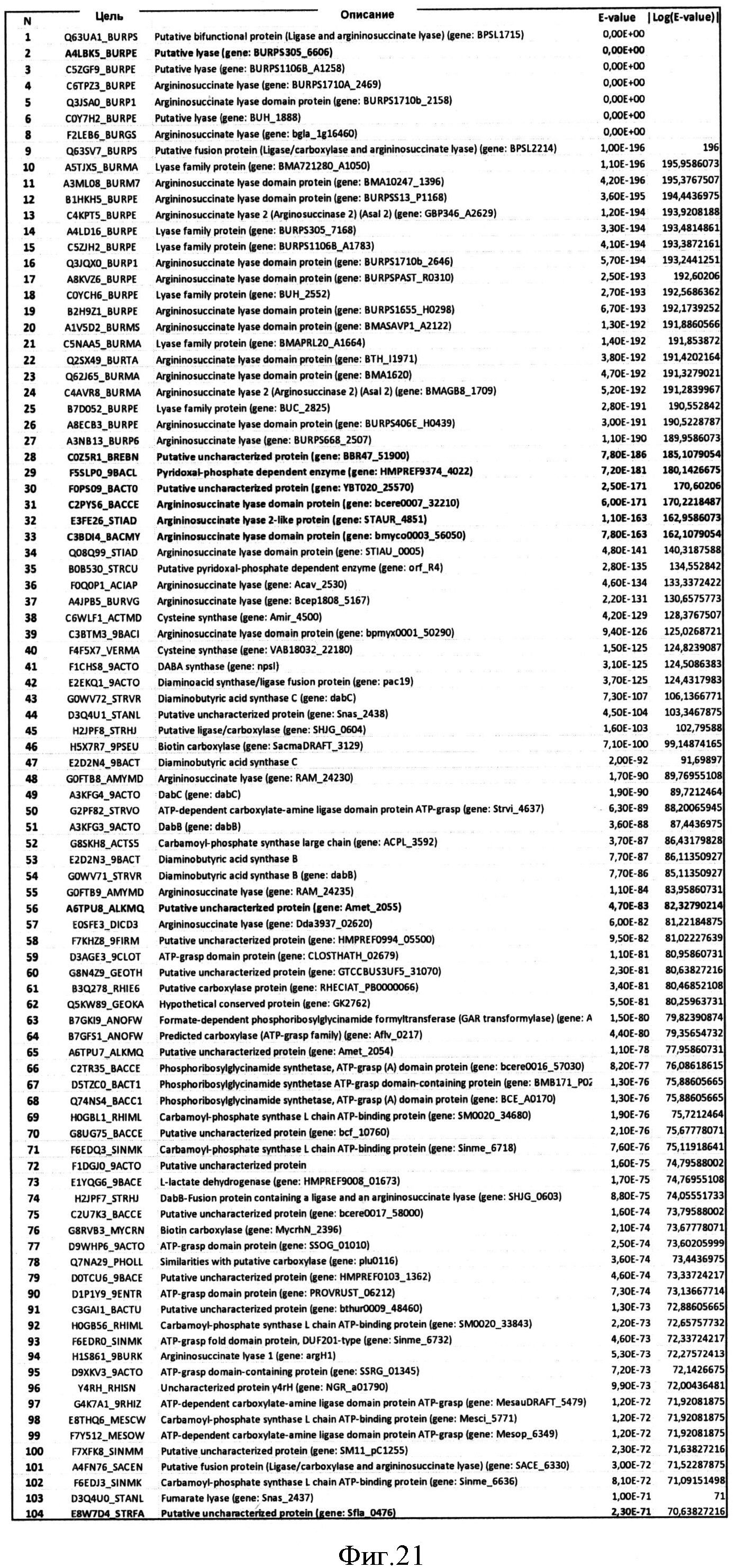
Фигура 25 показывает данные, полученные с помощью программы HMMsearch на основе выравнивания BBR47_51900, Staur_4851, DES, ВСЕ, BMY, BTH, BUR, АМЕ и SFL (представлены первые 38 хитов).
Фигура 26 показывает анализ изофункциональных Lals с использованием профиля HMMs (Модели с 1 по 7). *: E-value=0 (Фигуры 21, 23 и 25). BBR означает BBR47_51900 и STA означает Staur_4851.
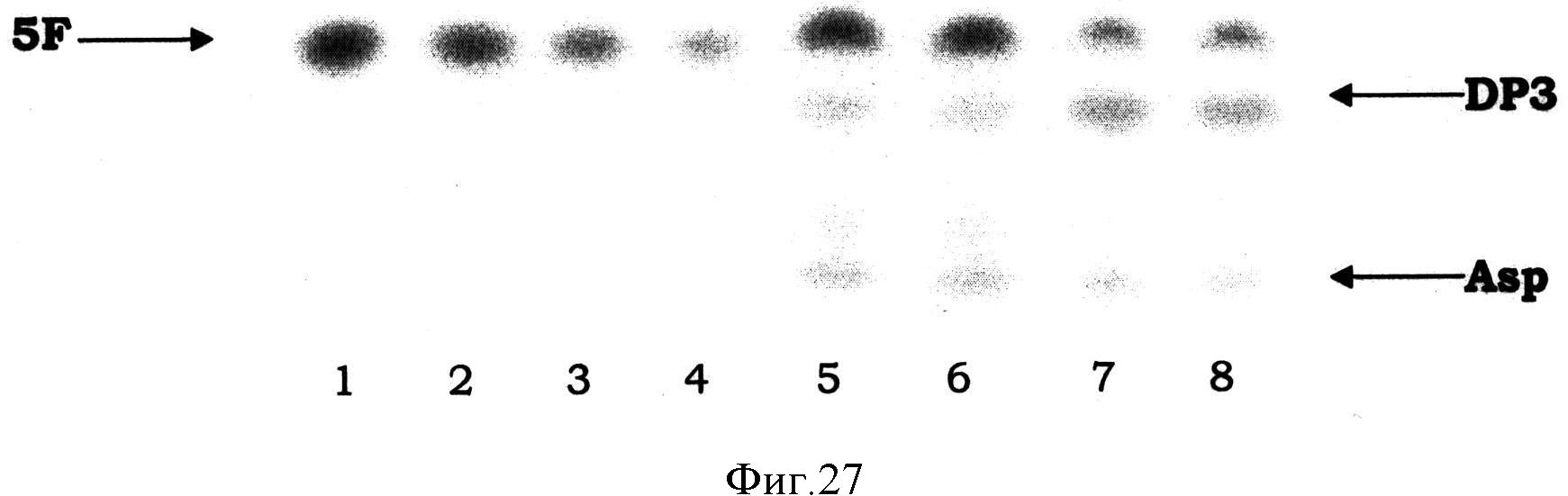
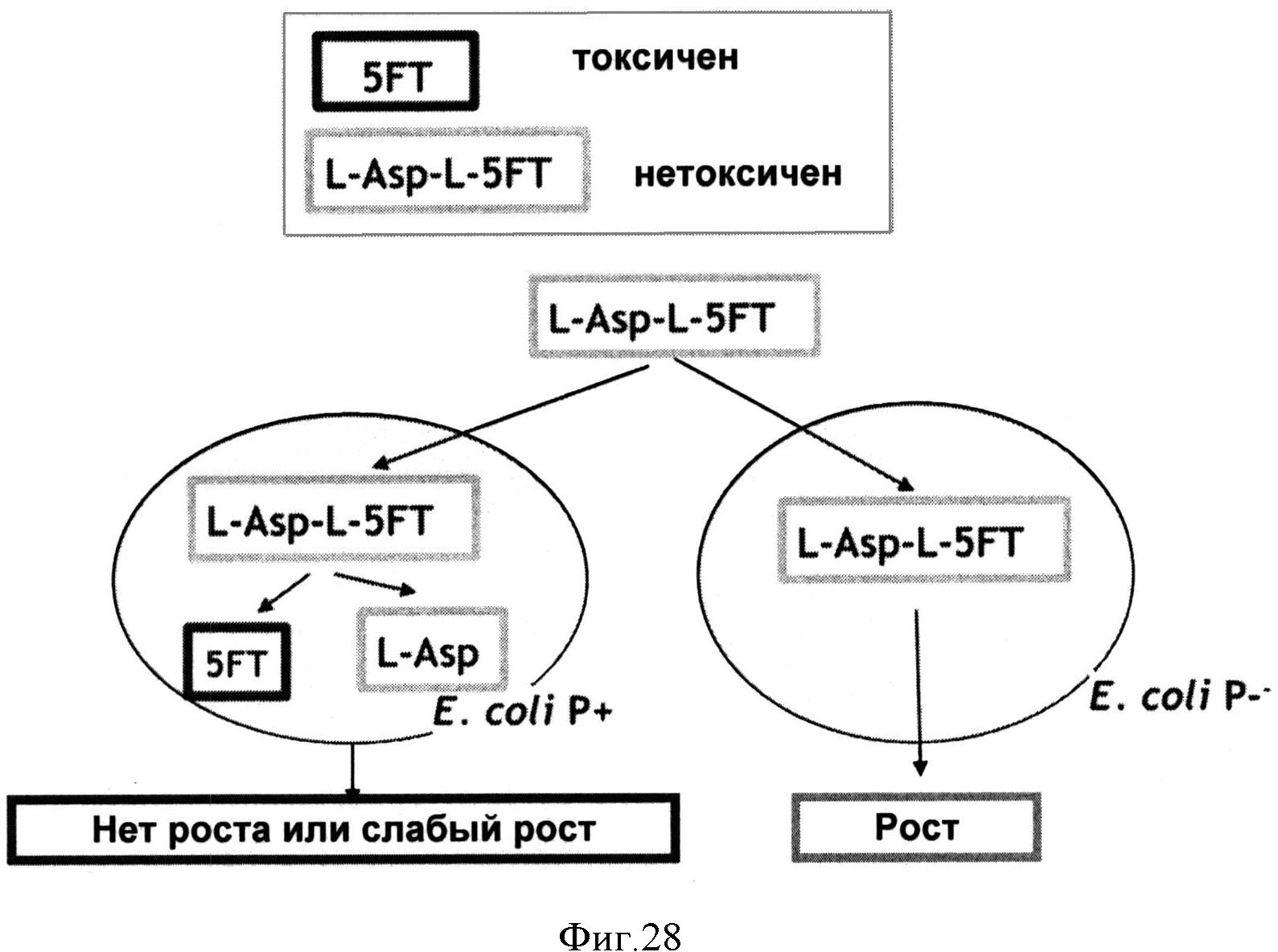
Фигура 27 показывает ТСХ-анализ специфической аспартат-пептид-гидролизующей (DP3-гидролизующей) активности в штаммах Е.coli 4-5Δ. Для калибровки использовали раствор (1 мкл) 5-фтортриптофана (стандарт): (1) 3 мМ, (2) 2 мМ, (3) 1 мМ и (4) 0,5 мМ. Аликвоту (1 мкл) реакционной смеси, содержащей Mn2+ (5, 6) или Zn2+ (7, 8), помещали на пластинку ТСХ. 5FT - 5-фтортриптофан, DP3 - L-Asp-L-5-фтортриптофан дипептид, Asp - L-аспартат.
Фигура 28 показывает схему исследования токсичности DP3 ввиду специфической аспартат-пептид-гидролизующей активности в штаммах Е.coli 1-5Δ. Штамм Е.coli выращивают в присутствии дипептида DP3. В штамме, содержащем пептидазы (Е.coli P+), DP3 гидролизуется, что приводит к образованию L-аспартата и 5-фтортриптофана (5FT). 5FT является токсическим для клетки, что, таким образом, приводит к задержке роста. Дипептид DP3 является стабильным и не влияет на клеточный рост штамма, дефицитного по пептидазам, и штамма с низкой пептидазной активностью (Е.coli P-).
Описание последовательностей
SEQ ID NO:1 показывает ген BBR47_51900
SEQ ID NO:2 показывает белок BBR47_51900
SEQ ID NO:3 показывает ген Staur_4851
SEQ ID NO:4 показывает белок Staur_4851
SEQ ID NO:5 показывает ген DES
SEQ ID NO:6 показывает белок DES
SEQ ID NO:7 показывает ген BUR
SEQ ID NO:8 показывает белок BUR
SEQ ID NO:9 показывает ген ВСЕ
SEQ ID NO:10 показывает белок ВСЕ
SEQ ID NO:11 показывает ген ВТН
SEQ ID NO:12 показывает белок ВТН
SEQ ID NO:13 показывает ген АМЕ
SEQ ID NO:14 показывает белок АМЕ
SEQ ID NO:15 показывает ген SFL
SEQ ID NO:16 показывает белок SFL
SEQ ID NO:17 показывает ген BMY
SEQ ID NO:18 показывает белок BMY
SEQ ID NO:19 показывает фрагмент XbaI-EcoRI, содержащий BBR47_51900
SEQ ID NO:20 показывает фрагмент XbaI-EcoRI, содержащий Staur_4851
SEQ ID NO:21 показывает фрагмент XbaI-EcoRI, содержащий DES
SEQ ID NO:22 показывает фрагмент XbaI-EcoRI, содержащий BUR
SEQ ID NO:23 показывает фрагмент XbaI-EcoRI, содержащий ВСЕ
SEQ ID NO:24 показывает фрагмент XbaI-EcoRI, содержащий ВТН
SEQ ID NO:25 показывает фрагмент XbaI-EcoRI, содержащий АМЕ
SEQ ID NO:26 показывает фрагмент XbaI-EcoRI, содержащий SFL
SEQ ID NO:27 показывает фрагмент XbaI-EcoRI, содержащий BMY
SEQ ID NO:28 показывает праймер P1
SEQ ID NO:29 показывает праймер Р2
SEQ ID NO:30 показывает праймер Р3
SEQ ID NO:31 показывает праймер Р4
SEQ ID NO:32 показывает праймер Р5
SEQ ID NO:33 показывает праймер Р6
SEQ ID NO:34 показывает праймер Р7
SEQ ID NO:35 показывает праймер Р8
SEQ ID NO:36 показывает праймер Р9
SEQ ID NO:37 показывает праймер Р10
SEQ ID NO:38 показывает праймер Р11
SEQ ID NO:39 показывает праймер Р12
SEQ ID NO:40 показывает праймер Р13
SEQ ID NO:41 показывает праймер Р14
SEQ ID NO:42 показывает праймер Р15
SEQ ID NO:43 показывает праймер Р16
SEQ ID NO:44 показывает праймер Р17
SEQ ID NO:45 показывает праймер Р18
SEQ ID NO:46 показывает праймер Р19
SEQ ID NO:47 показывает праймер Р20
SEQ ID NO:48 показывает праймер Р21
SEQ ID NO:49 показывает праймер Р22
SEQ ID NO:50 показывает праймер Р23
SEQ ID NO:51 показывает праймер Р24
SEQ ID NO:52 показывает праймер Р25
SEQ ID NO:53 показывает праймер Р26
SEQ ID NO:54 показывает праймер Р27
SEQ ID NO:55 показывает праймер Р28
SEQ ID NO:56 показывает праймер Р29
SEQ ID NO:57 показывает праймер Р30
SEQ ID NO:58 показывает праймер Р31
SEQ ID NO:59 показывает праймер Р32
SEQ ID NO:60 показывает праймер Р33
SEQ ID NO:61 показывает праймер Р34
SEQ ID NO:62 показывает праймер Р35
SEQ ID NO:63 показывает праймер Р36
SEQ ID NO:64 показывает праймер Р37
SEQ ID NO:65 показывает праймер Р38
SEQ ID NO:66 показывает праймер Р39
SEQ ID NO:67 показывает праймер Р40
SEQ ID NO:68 показывает оптимизированный ген, кодирующий BBR47_51900 из Brevibacillus brevis NBRC 100599
SEQ ID NO:69 показывает оптимизированный ген, кодирующий Staur_4851 из Stigmatella aurantiaca
Наилучший способ осуществления изобретения
1. Фермент
Термин «фермент» может означать L-аминокислота-α-лигазу (Lal), обладающую способностью присоединять аминокислоты высокоэнергетическим молекулярно-зависимым (макроэргическим) способом с образованием пептидной связи между аминокислотными остатками.
Ферментом, согласно настоящему изобретению, может быть L-аминокислота-α-лигаза, выбранная из группы, состоящей из BBR47_51900 (гипотетический белок), Staur_4851 (аргининсукцинатлиаза 2-подобный белок), DES (пиридоксальфосфат-зависимый фермент), BUR (предполагаемая лиаза), ВСЕ (аргининосукцинатлиаза доменный белок), ВТН (гипотетический белок YBT020_25570), AME (консервативный гипотетический белок), SFL (белок с неизвестной функцией DUF201) и BMY (аргининосукцинатлиаза доменный белок), которая не ограничивается вышеупомянутыми белками.
Нуклеотидная последовательность гена (NCBI перечень последовательности: YP_002774671.1; нуклеотиды, комплементарные нуклеотидам в положении: с 5464162 по 5465418; идентификатор гена: 7721040).
Нуклеотидная последовательность гена (NCBI перечень последовательности: YP_002774671.1; нуклеотиды, комплементарные нуклеотидам в положении с 5464162 по 5465418; идентификатор гена: 7721040) из Brevibacillus brevis NBRC 100599 (NCBI идентификатор таксона: 358681) и аминокислотная последовательность BBR47_51900, кодируемая геном, показаны в SEQ ID NO:1 и SEQ ID NO:2, соответственно.
Нуклеотидная последовательность гена (NCBI перечень последовательности: AD072629.1; нуклеотиды, комплементарные нуклеотидам в положении: с 5973963 по 5975216; идентификатор гена: 9878344) из Stigmatella aurantiaca DW4/3-1 (NCBI идентификатор таксона: 378806) и аминокислотная последовательность Staur_4851, кодируемая геном, представлены в SEQ ID NO:3 и SEQ ID NO:4, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank EGK06810.1, GI: 332967701) из Desmospora sp. 8437 (NCBI идентификатор таксона: 997346) и аминокислотная последовательность DES, кодируемая этим геном, представлены в SEQ ID NO:5 и SEQ ID NO:6, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank EBA51208.1, GI: 134251129) из Burkholderia pseudomallei 305 (NCBI идентификатор таксона: 425067) и аминокислотная последовательность BUR, кодируемая этим геном, представлены в SEQ ID NO:7 и SEQ ID NO:8, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank EEK72190.1, GI: 228615090) из Bacillus cereus AH621 (NCBI идентификатор таксона: 526972) и аминокислотная последовательность ВСЕ, кодируемая этим геном, представлены в SEQ ID NO:9 и SEQ ID NO:10, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank ADY24341.1, GI: 324329081) из Bacillus thuringiensis subsp. finitimus (штамм YBT-020) (NCBI идентификатор таксона: 930170) и аминокислотная последовательность ВТН, кодируемая этим геном, представлены в SEQ ID NO:11 and SEQ ID NO:12, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank ABR48216.1, GI: 149949688) из Alkaliphilus metalliredigens QYMF (NCBI идентификатор таксона: 293826) и аминокислотная последовательность АМЕ, кодируемая этим геном, представлены в SEQ ID NO:13 и SEQ ID NO:14, соответственно.
Нуклеотидная последовательность гена (инвентарный номер в GenBank ADWO 1942.1, GI: 320007092) из Streptomyces flavogriseus ATCC 33331 (NCBI идентификатор таксона: 591167) и аминокислотная последовательность SFL, кодируемая этим геном, представлены в SEQ ID NO:15 и SEQ ID NO:16, соответственно.
Нуклеотидная последовательность гена (NCBI перечень последовательности ZP_04160564.1, GI: 229002475) из Bacillus mycoides Rock3-17 (NCBI идентификатор таксона: 526999) и аминокислотная последовательность BMY, кодируемая этим геном, представлены в SEQ ID NO:17 and SEQ ID NO:18, соответственно.
Ввиду того что могут быть некоторые различия в последовательностях ДНК между родами или видами и штаммами указанных родов, гены, кодирующие BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, не ограничены генами, представленными в SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 или 17, но могут включать гены, которые являются вариантами нуклеотидных последовательностей или гомологичны нуклеотидным последовательностям SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 или 17, кодирующие варианты белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY. Кроме того, гены, кодирующие BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, могут быть вариантами нуклеотидных последовательностей.
Термин «вариант нуклеотидной последовательности» может означать нуклеотидную последовательность, которая кодирует «вариант белка».
Термин «вариант нуклеотидной последовательности» может означать нуклеотидную последовательность, которая кодирует «вариант белка» с использованием любых синонимичных аминокислотных кодонов в соответствии с таблицей стандартного генетического кода (см., например, Lewin В., Genes VIII, 2004, Pearson Education, Inc., Upper Saddle River, NJ 07458).
Термин «вариант нуклеотидной последовательности» также может означать нуклеотидную последовательность, которая гибридизуется в жестких условиях с нуклеотидной последовательностью, комплементарной последовательности, представленной в SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 или 17, или с зондом, который может быть синтезирован на основе указанных нуклеотидных последовательностей, при условии, что он кодирует функциональную L-аминокислота-α-лигазу. Под «жесткими условиями» понимаются такие условия, при которых образуется специфический гибрид, например гибрид, имеющий идентичность не менее чем 80%, не менее чем 90%, не менее чем 95%, не менее чем 96%, не менее чем 97%, не менее чем 98% или не менее чем 99%, и не образуется неспецифический гибрид, например гибрид, имеющий гомологию меньшую, чем указано выше. Практическим примером жестких условий может быть однократная или многократная отмывка, или, в другом случае, двух- или трехкратная отмывка при концентрации солей 1×SSC (стандарт цитрата натрия или стандарт хлорида натрия) и 0,1% SDS (додецилсульфат натрия) или, в другом случае, 0, 1×SSC и 0,1% SDS при 60°С или 65°С. Продолжительность отмывки зависит от типа используемой для блоттинга мембраны и, как правило, такова, как рекомендовано производителем. Например, рекомендуемая продолжительность отмывки для положительно заряженной нейлоновой мембраны Hybond™-N+ (GE Healthcare) при жестких условиях составляет 15 минут. Промывка может быть произведена двух- или трехкратно. В качестве зонда может быть использована часть последовательности, комплементарной последовательности, приведенной в SEQ ID NO:3, 5, 7, 9, 11, 13, 15 или 17. Подобный зонд может быть получен с помощью метода ПЦР (полимеразная цепная реакция. White T.J. et al., Trends Genet., 1989, 5:185-189) с использованием олигонуклеотидных праймеров, приготовленных на основе последовательностей, приведенных в SEQ ID NO:1, 3, 5, 7, 9, 11, 13, 15 или 17 и фрагмента ДНК, содержащего нуклеотидную последовательность в качестве матрицы. Рекомендуемая длина зонда должна быть >50 п.н., она может быть подобрана в зависимости от условий гибридизации и составляет обычно от 100 до 1000 п.н. Например, при использовании в качестве зонда фрагмента ДНК длиной около 300 п.н. условия отмывки могут быть следующие: 2×SSC, 0.1% SDS при 50°С, или при 60°С, или при 65°С. С другой стороны, жесткими условиями может быть гибридизация в 6×SCC при 45°C с последующими одно-, двух или многократными отмывками в 0,2×SCC и 0,1% SDS при температуре от 50 до 65°С.
Гены, кодирующие белки BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, известны (см. описание выше), поэтому гены, кодирующие варианты белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, могут быть получены с использованием метода ПЦР с использованием праймеров, синтезированных на основе нуклеотидной последовательности генов, кодирующих BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY. Гены, кодирующие белки BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY или их варианты белков из других микроорганизмов, могут быть получены аналогичным способом.
Термин «вариант белка» может означать белок, который содержит одно или несколько изменений в последовательности при сравнении с SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, являются ли они заменами, делениями, вставками и/или добавлениями аминокислотных остатков при условии, что активность, соответствующая активности белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, соответственно, сохранена, или третичная структура белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY изменена несущественно по сравнению с белком дикого типа или немодифицированным белком. Количество изменений в вариантах белков зависит от положения или вида аминокислотных остатков в третичной структуре белка. Оно может быть, но строго не ограничено, с 1 по 45, или с 1 по 30, или с 1 по 15, или с 1 по 10, или с 1 по 5 в SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18.
Примерами замены, делеции, вставки и/или добавления одного или нескольких аминокислотных остатков могут быть консервативная мутация(и), такая, что активность и характеристики варианта белка сохраняются и подобны белкам BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY. Примером консервативной мутации является консервативная замена. Консервативными заменами могут быть взаимные замены между Phe, Trp и Tyr, если сайт замещения является ароматической аминокислотой; между Ala, Leu, Ile и Val, если сайт замещения является гидрофобной аминокислотой; между Glu, Asp, Gln, Asn, Ser, His и Thr, если сайт замещения является гидрофильной аминокислотой; между Gln и Asn, если сайт замещения является полярной аминокислотой; между Lys, Arg и His, если сайт замещения является основной аминокислотой; между Asp и Glu, если сайт замещения является кислой аминокислотой; и между Ser и Thr, если сайт замещения является аминокислотой, имеющей гидроксильную группу. Примеры консервативных замен включают замену Ser или Thr на Ala, замену Asn, Glu или Gln на Asp, замену Ser или Ala на Cys, замену Asn, Glu, Lys, His, Asp или Arg на Gln, замену Asn, Gln, Lys или Asp на Glu, замену Pro на Gly, замену Asn, Lys, Gln, Arg или Tyr на His, замену Leu, Met, Val или Phe на Ile, замену Ile, Met, Val или Phe на Leu, замену Asn, Glu, Gln, His или Arg на Lys, замену Ile, Leu, Val или Phe на Met, замену Trp, Tyr, Met, Ile или Leu на Phe, замену Thr или Ala на Ser, замену Ser или Ala на Thr, замену Phe или Tyr на Trp, замену His, Phe или Trp на Tyr и замену Met, Ile или Leu на Val. Эти изменения в варианте белка могут происходить в участках белка, не являющимися критичными для функции белка. Это объясняется тем, что некоторые аминокислоты, имеют высокую гомологию друг к другу, такую, что третичная структура или активность белка не подвержены влиянию ввиду таких изменений. Таким образом, варианты белка, кодируемые генами, представленными в SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 или 17, могут иметь подобие или идентичность не менее чем 40%, не менее чем 50%, не менее чем 60%, не менее чем 70%, не менее чем 80%, не менее чем 90%, не менее чем 95%, не менее чем 96%, не менее чем 97%, не менее чем 98%, или не менее чем 99% по отношению к аминокислотным последовательностям, представленным в SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, до тех пор, пока функциональность белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, соответственно, остается неизменной. Также, варианты белка, кодируемые генами, представленными в SEQ ID NOs:1, 3, 5, 7, 9, 11, 13, 15 или 17, могут иметь гомологию, которая может быть определена через значение |Log10(E-value)|, рассчитанное с помощью программы HMMsearch, когда скрытый профиль модели Маркова (the profile hidden Markov model, profile HMM; профиль НММ) построен с помощью вышеупомянутой программы (Finn R.D. et al., HMMER web server: interactive sequence similarity searching, Nucleic Acids Res., 2011, 39 (Web Server issue):W29-37), как описано ниже в Примере 6, не менее чем 128, не менее чем 142, не менее чем 162, не менее чем 175, не менее чем 182, не менее чем 196, или не менее чем 233 по отношению к полным аминокислотным последовательностям, приведенным в SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, до тех пор, пока функциональность белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, соответственно, остается неизменной.
Примеры замен, делеций, вставок и/или добавлений одного или нескольких аминокислотных остатков также могут включать неконсервативную(ые) мутацию(и) при условии, что эта(эти) мутация(и) компенсирована(ы) одной или несколькими вторичными мутациями в одном или нескольких положениях аминокислотной последовательности так, что активность и характеристики варианта белка остаются неизменными и подобны белкам BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY.
Степень гомологии белка или ДНК, может быть определена с использованием нескольких известных подходов, например, компьютерных алгоритмов BLAST и FASTA и метода ClustalW. Алгоритм BLAST (Basic Local Alignment Search Tool, www.ncbi.nlm.nih.gov/BLAST/), позволяющий проводить иерархический поиск, заложен в программах blastp, blastn, blastx, megablast, tblastn и tblastx; эти программы присваювают уровень значимости найденным объектам, используя статистические методы, описанные в Samuel K. and Altschul S.F. («Methods for assessing the statistical significance of molecular sequence features by using general scoring schemes» Proc. Natl. Acad. Sci. USA, 1990, 87:2264-2268; «Applications and statistics for multiple high-scoring segments in molecular sequences». Proc. Natl. Acad. Sci. USA, 1993, 90:5873-5877). Алгоритм BLAST вычисляет три параметра: число аминокислотных остатков, идентичность и сходство. Поисковый алгоритм FASTA описан в Pearson W.R. («Rapid and sensitive sequence comparison with FASTP and FASTA», Methods Enzymol., 1990, 183:63-98). Метод ClustalW описан в Thompson J.D. et al. («CLUSTAL W: improving the sensitivity of progressive multiple sequence alignment through sequence weighting, position-specific gap penalties and weight matrix choice». Nucleic Acids Res., 1994, 22:4673-4680).
Термин «активность L-аминокислота-α-лигазы (Lal)» может означать активность фермента, катализирующего реакцию соединения аминокислот макроэргическим молекулярно-зависимым способом с образованием пептидной связи между аминокислотными остатками. Продуктом реакции, катализируемой Lal, может быть дипептид, трипептид или пептид с линейной или разветвленной структурой, состоящий из более чем трех аминокислотных остатков или их производных. Схема реакции, катализируемой Lal, может быть описана, как показано на Фигуре 1, без ограничения по типу аминокислот или их производных и условий реакции, которые применяются в следующих не ограничивающих настоящее изобретение примерах. Активность Lal может быть определена методом, приведенным в Примере 3 или в Tabata K. et al., J. Bacterial., 2005, 187(15):5195-5202. Термин «активность L-аминокислота-α-лигазы (Lal)» может быть эквивалентен, в частности, термину «дипептид-синтезирующая активность».
Кроме того, когда аминокислотная последовательность, которая содержит замену, делецию, вставку и/или добавление одного или нескольких аминокислотных остатков в аминокислотной последовательности SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18, остаточная активность L-аминокислота-α-лигазы может быть сохранена на уровне 10% или более, на уровне 30% или более, на уровне 50% или более, на уровне 70% или более, и на уровне 90% или более от активности белка, имеющего аминокислотную последовательность SEQ ID NOs:2, 4, 6, 8, 10, 12, 14, 16 и 18.
Термин «изофункциональный белок» может означать белок, который имеет активность L-аминокислота-α-лигазы (Lal), как описано выше. Например, изофункциональный белок может синтезировать дипептид, имеющий кислый L-аминокислотный остаток, такой как остаток L-Glu или L-Asp, на N-конце и любую другую L-аминокислоту или ее производное на С-конце.
2. Бактерия
Термин «бактерия-продуцент дипептида» может означать бактерию семейства Enterobacteriaceae, такую как бактерия, принадлежащая к роду Escherichia, которая способна продуцировать и вызывать накопление дипептида в культуральной жидкости, когда указанная бактерия выращивается (культивируется) в питательной среде. Бактерия может обладать способностью к продукции дипептида в среде или бактериальных клетках и вызывать накопление дипептида до такой степени, что дипептид выделяется (извлекается) из культуральной жидкости или бактериальных клеток, когда бактерия выращена в питательной среде.
Бактерия может обладать способностью к продукции дипептида изначально в соответствии со своими природными характеристиками или может быть модифицирована таким образом, чтобы она получила способность продуцировать дипептид, с использованием метода мутагенеза или технологии рекомбинантных ДНК.
Бактерия, принадлежащая к семейству Enterobacteriaceae, может быть из рода Enterobacter, Erwinia, Escherichia, Klebsiella, Morganella, Pantoea, Photorhabdus, Providencia, Salmonella, Yersinia и т.д. и может обладать способностью продуцировать дипептид. Более точно, могут быть использованы бактерии, принадлежащие к семейству Enterobacteriaceae в соответствии с таксономией, используемой в базе данных NCBI (National Center for Biotechnology Information) (http://www.ncbi.nlni.nih.gov/Taxonomy/Browser/wwwtax.cgi?id=543). Примерами могут служить бактерии, принадлежащие к роду Escherichia, Enterobacter или Pantoea.
Выбор штаммов бактерий, принадлежащих к роду Escherichia, которые могут быть модифицированы в настоящем изобретении, не ограничен каким-либо образом, однако, в качестве примеров, бактерии рода Escherichia, описанные в книге Neidhardt et al. (Bachmann, B.J., Derivations and genotypes of some mutant derivatives of E.coli K-12, p.2460-2488. In F.C. Neidhardt et al. (ed.), E.coli and Salmonella: cellular and molecular biology, 2nd ed. ASM Press, Washington, D.C., 1996) могут быть включены в число бактерий согласно настоящему изобретению. В качестве конкретного примера могут быть взяты штаммы E.coli, такие как E.coli W3110 (АТСС 27325), E.coli MG1655 (АТСС 47076) и т.д., которые происходят из исходного штамма дикого типа, т.е. штамма E.coli K-12. Этот штамм может быть получен, в частности, из Американской коллекции типовых культур «American Type Culture Collection, (АТСС)» (P.O. Box 1549, Manassas, VA 20108, United States of America). Каждому штамму присвоен индивидуальный регистрационный номер, и штаммы могут быть заказаны согласно регистрационному номеру (см. ссылку www.atcc.org). Регистрационные номера штаммов находятся в списке каталога Американской коллекции типовых культур «American Type Culture Collection, АТСС».
Примеры бактерий Enterobacter включают Enterobacter agglomerans, Enterobacter aerogenes и т.д. Примеры бактерии Pantoea включают Pantoea ananatis и т.д. Недавно некоторые виды Enterobacter agglomerans были переклассифицированы как Pantoea agglomerans, Pantoea ananatis или Pantoea stewartii на основе анализа нуклеотидной последовательности 16S рРНК и других доказательств. Бактерии, относящиеся к роду Enterobacter или Pantoea, могут быть использованы в соответствии с настоящим изобретением, так как принадлежат семейству Enterobacteriaceae. Полученные с использованием технологий генной инженерии штаммы Pantoea ananatis, такие как штамм Pantoea ananatis AJ13355 (PERM BP-6614), штамм AJ13356 (PERM BP-6615), штамм AJ13601 (PERM BP-7207) и их производные могут быть использованы в соответствии с настоящим изобретением. Эти штаммы были классифицированы как Enterobacter agglomerans при выделении, и они депонированы как Enterobacter agglomerans. Однако позднее они были классифицированы как Pantoea ananatis на основе анализа нуклеотидной последовательности 16S рРНК и других доказательств.
Бактерия-продуцент дипептида, согласно настоящему изобретению, может быть модифицирована таким образом, чтобы иметь ослабленные или инактивированные один или более гены, кодирующие один или более видов белков, имеющих пептидазную или протеолитическую активность, таким образом, чтобы активность пептидаз(ы) была снижена. Например, могут быть ослаблены и/или инактивированы гены, кодирующие пептидазы, такие как рерА (KEGG, Kyoto Encyclopedia of Genes and Genomes, входящий No. b4260), pepB (KEGG, входящий No. b2523), pepD (KEGG, входящий No. b0237), pepE (KEGG, входящий No. b4021), pepP (KEGG, входящий No. b2908), pepQ (KEGG, входящий No. b3847), pepN (KEGG, входящий No. b0932), pepT (GenBank accession No. AAC74211), iadA (KEGG, входящий No. b4328), iaaA(ybiK) (KEGG, входящий No. b0828), dapE (KEGG, входящий No. b2472) и т.д.
Вышеописанная бактерия-продуцент дипептида также может быть модифицирована таким образом, чтобы иметь ослабленный или инактивированный один или более генов одного или более типов, кодирующих белок(ки), который (е) имеет(ют) активность дипептидной пермеазы (dpp), причем активность пептидной пермеазы (пермеаз) уменьшена. Например, могут быть ослаблены и/или инактивированы один или более генов дипептидных пермеаз, такие как dppA (KEGG, входящий № b3544), dppB (KEGG, входящий № b3543), dppC (KEGG, входящий № b3542), dppD (KEGG, входящий № b3541), dppF (KEGG, входящий № b3540) и т.д. Делеция целого оперона генов dpp (dppA, dppB, dppC, dppD и dppF) также может быть предпочтительной в бактерии-продуценте дипептида.
Вышеописанная бактерия-продуцент дипептида также может быть модифицирована таким образом, чтобы иметь ослабленный или инактивированный один или более генов одного или более типов, кодирующие белки, вовлеченные в биосинтез ароматических аминокислот, причем активность таких белков снижена. Например, могут быть ослаблены и/или инактивированы один или более генов, такие как tyrR (KEGG, входящий № b1323), tryA (KEGG, входящий № b2600) и т.д.
Термин «ослабленный ген, кодирующий пептидазу» или «ослабленный ген, кодирующий белок» эквивалентен термину «пептидаза, кодируемая геном с ослабленной экспрессией» или термину «белок, кодируемый геном с ослабленной экспрессией», соответственно. В дальнейшем термин «пептидаза» может быть заменен на термин «белок», как изложено выше (например, белок, имеющий активность дипептидной пермеазы (dpp), или белок, вовлеченный в биосинтез ароматических аминокислот) для интерпретации термина «ослабленный ген, кодирующий белок», и тому подобное. Таким образом, такие заменяющие термины могут упоминаться с целью уточнения настоящего изобретения.
Термин «пептидаза, кодируемая геном с ослабленной экспрессией» может означать, что количество пептидазы в модифицированной бактерии, в которой экспрессия гена, кодирующего пептидазу, ослаблена, снижена по сравнению с немодифицированной бактерией, например штаммом дикого типа бактерии, принадлежащей к семейству Enterobacteriaceae или более точно роду Escherichia, такой как штамм Е.coli K-12.
Термин «пептидаза, кодируемая геном с ослабленной экспрессией» также может означать, что модифицированная бактерия включает модифицированный ген, который кодирует мутантный белок, имеющий сниженную активность по сравнению с белком дикого типа, или участок, функционально связанный с геном, включающий последовательности, контролирующие экспрессию гена, такие как промотеры, энхансеры, аттенюаторы, рибосома-связывающие участки (RBS), последовательности Шайна-Дальгарно (Shine-Dalgarno, SD) и т.д., модифицированные с целью снизить уровень экспрессии гена, кодирующего пептидазу, и другие примеры (см., например, WO95/34672; Carrier Т.A. and Keasling J.D., Biotechnol. Prog., 1999, 15:58-64).
Экспрессия гена, кодирующего пептидазу, может быть ослаблена путем замены последовательности, контролирующей экспрессию гена, такого как промотор на хромосомной ДНК, на более слабую. Сила промотора определяется частотой актов инициации синтеза РНК. Примеры методов для оценки силы промотора описаны в Goldstein et al., Prokaryotic promoters in biotechnology, Biotechnol. Annu. Rev., 1995, 1:105-128, и т.д. Кроме того, также возможно ввести нуклеотидную замену в промоторный участок целевого гена таким образом, чтобы ослабить силу промотора, как описано в Международной патентной публикации WO00/1835. Кроме того, известно, что замена нескольких нуклеотидов в области между SD последовательностью и стартовым кодоном на рибосома-связующем участке (RBS), в частности, в последовательности, расположенной выше и сразу за стартовым кодоном, значительно влияет на эффективность трансляции мРНК. Подобная модификация RBS может комбинироваться с уменьшением уровня транскрипции гена, кодирующего пептидазу.
Экспрессия гена, кодирующего пептидазу, также может быть ослаблена путем вставки транспозона или IS-фактора в кодирующий участок гена (патент США №5,175,107) или с помощью обычных методов, такого как мутагенез, за счет обработки микроорганизмов, например, УФ-излучением или нитрозогуанидином (N-метил-N′-нитро-N-нитрозогуанидин). Кроме того, введение сайт-направленной мутации за счет замены гена с использованием гомологичной рекомбинации, как указано выше, также может быть осуществлено с помощью плазмиды, которая неспособна реплицироваться в штамме-хозяине.
Термин «ферментативная активность понижена» может означать, что ферментативная активность пептидазы в модифицированном штамме ниже, чем в немодифицированном штамме, например в штамме дикого типа бактерии, принадлежащей к семейству Enterobacteriaceae или более точно роду Escherichia. Например, ферментативная активность пептидазы, кодируемой геном, может быть снижена за счет инактивации гена.
Термин «активность пептидазы понижена» также может означать, что активность пептидазы в реакции расщепления пептида снижена по сравнению с пептидазой дикого типа, кодируемой генами дикого типа, такими как рерА, рерВ, pepD, pepE, pepP, pepQ, pepN, pepT, iadA, iaaA(ybiK), dapE и т.д.
В модифицированной бактерии активность пептидазы может быть снижена по крайней мере на не менее чем 10% или более, не менее чем 30% или более, не менее чем 50% или более, не менее чем 70% или более, не менее чем 90% или более по сравнению с пептидазой, кодируемой геном дикого типа в немодифицированной бактерии, принадлежащей к семейству Enterobacteriaceae или более точно к роду Escherichia.
Термин «активность пептидазы» или «протеолитическая активность» может означать активность фермента, катализирующего реакцию внутримолекулярного расщепления пептидной связи (R. Beynon (ed.) and J.S. Bond (ed.), «Proteolytic Enzymes: A Practical Approach», 2nd ed., Oxford University Press, USA (2001)).
Пептидная расщепляющая активность микроорганизма может быть измерена помещением пептида в качестве субстрата и клеток микроорганизма в среду с целью проведения реакции пептидной деградации с последующим определением количества оставшегося пептида известным методом, например методом ВЭЖХ или как описано в Kristjansson M.M., Activity measurements of proteinases using synthetic substrates (UNIT C2.1) или Akpinar O. and Penner M.H., Peptidase activity assays using protein substrates (UNIT C2.2) in Current Protocols in Food Analytical Chemistry (UNIT C2, Proteolytic Enzymes), John Wiley 85 Sons, Inc. (2002).
Ферментативная активность пептидазы также может быть уменьшена за счет введения мутации в ген на хромосоме так, чтобы внутриклеточная активность пептидазы была уменьшена по сравнению с немодифицированным штаммом. Такой мутацией в гене(ах) может быть замена одного или нескольких оснований, приводящая к замене аминокислотного остатка в белке, кодируемом геном(ами) («миссенс»-мутация), введение стоп-кодона («нонсенс»-мутация), делеция одного или нескольких оснований, приводящая к сдвигу рамки считывания, вставка гена, сообщающего устойчивость к антибиотику, частичное или полное удаление генов в геноме (Qiu Z. and Goodman M.F., J. Biol. Chem. 1997, 272:8611-8617; Kwon D.H. et al., J. Antimicrob. Chemother. 2000, 46:793-796).
Термин «инактивированный ген, кодирующий пептидазу» может означать, что модифицированный ген кодирует полностью неактивную или нефункциональную пептидазу. Также возможно, что модифицированный участок ДНК не способен к естественной экспрессии гена благодаря делеции части гена или целого гена, сдвигу рамки считывания гена, введению миссенс/нонсенс мутации(ий) или модификации смежного участка гена, включению последовательностей, контролирующих экспрессию гена, таких как промотор(ы), энхансер(ы), аттенуатор(ы), рибосома-связывающий(ие) сайт(ы) (RBS, ribosome-binding site) и т.д. Инактивация гена также может быть проведена обычными способами, такими как мутагенез, путем обработки микроорганизмов, например, УФ-облучением или нитрозогуанидином (N-метил-N′-нитро-N-нитрозогуанидин), сайт-направленный мутагенез, нарушение структуры гена с использованием гомологичной рекомбинации и/или мутагенеза за счет вставки-делеции (Yu D. et al., Proc. Natl. Acad. Sci. USA, 2000, 97(12):5978-83; Datsenko K.A. и Wanner B.L., Proc. Natl. Acad. Sci. USA 2000, 97(12):6640-45), также называемого как «λRed-зависимая интеграция».
Термин «бактерия, модифицированная таким образом, что она содержит рекомбинантную ДНК» может означать, что бактерия модифицирована обычными способами так, что она содержит экзогенную ДНК. К обычным способам относятся, например, трансформация, трансфекция, инфекция, конъюгация и мобилизация. Трансформация, трансфекция, инфекция, конъюгация или мобилизация бактерии молекулой ДНК, кодирующей белок, может сообщить бактерии способность бактерии синтезировать белок, кодируемый молекулой ДНК. Способы трансформации, трансфекции, инфекции, конъюгации и мобилизации включают любые известные ранее описанные способы. Например, известен способ эффективной трансформации и трансфекции ДНК путем обработки клеток-реципиентов Escherichia coli K-12 хлоридом кальция с целью увеличения проницаемости клеток для ДНК (Mandel М. and Higa A., Calcium-dependent bacteriophage DNA infection, J. Mol. Biol., 1970, 53:159-162). Описаны способы специализированной и/или общей трансдукции (Morse M.L. et al., Transduction in Escherichia coli K-12, Genetics, 1956, 41(1):142-156; Miller J.H., Experiments in Molecular Genetics. Cold Spring Harbor, N.Y.: Cold Spring Harbor La. Press, 1972). Могут быть применены другие способы для случайной и/или направленной интеграции ДНК в геном хозяина, например, «Mu-зависимая интеграция/амплификация» (Akhverdyan et al., Appl. Microbiol. Biotechnol., 2011, 91:857-871), «Red/ET-зависимая» или «λRed/ET-зависимая интеграция» (Datsenko K.A. and Wanner B.L., Proc. Natl. Acad. Sci. USA 2000, 97(12):6640-45; Zhang Y., et al., Nature Genet., 1998, 20:123-128). Более того, для многократных вставок желаемых генов в дополнение к Mu-зависимой репликативной транспозиции (Akhverdyan et al., Appl. Microbiol. Biotechnol., 2011, 91:857-871) и химически индуцированной хромосомной эволюции, основанной на recA-зависимой гомологичной рекомбинации, приводящей к амплификации желаемых генов (Туо K.E.J. et al., Nature Biotechnol., 2009, 27:760-765), могут быть использованы другие подходы, основанные на различных комбинациях транспозиции, сайт-специфичной и/или гомологичной Red/ET-зависимой рекомбинации, и/или P1-зависимой общей трансдукции (см., например, Minaeva et al., BMC Biotechnology, 2008, 8:63; Koma D. et al., Appl. Microbiol. Biotechnol., 2012, 93(2):815-829).
Бактерия, описанная в настоящем изобретении, может быть модифицирована далее таким образом, что уровень экспрессии гена, кодирующего L-аминокислота-α-лигазу (Lal) или одного или более генов, кодирующих один или более белков, вовлеченных в биосинтез фенилаланина, повышен. Примеры такого белка включают pheA, aroG4 и aroL, кодирующие хоризматмутаза-префенатдегидратазу (CM-PD), 3-деокси-D-арабиногептулозонат-7-фосфатсинтетазу (DAHP синтетаза) и шикиматкиназу (SK), соответственно (см., например, патент Японии №3225597). В дальнейшем, термин «Lal» может быть заменен на белок, вовлеченный в биосинтез фенилаланина, для интерпретации термина «ген, кодирующий белки, вовлеченные в биосинтез фенилаланина» или подобные. Таким образом, такие заменяющие термины могут упоминаться с целью уточнения настоящего изобретения.
Термин «повышенная экспрессия гена, кодирующего Lal» может означать, что количество молекул белка, кодируемых Lal-кодирующим геном, в пересчете на одну клетку увеличено или активность в пересчете на молекулу белка (может быть названа как специфическая активность), кодируемого этим геном, повышена по сравнению с немодифицированным штаммом, таким как штамм дикого типа или родительский штамм. Примером немодифицированного штамма-сравнения могут быть штаммы дикого типа микроорганизма семейства Enterobacteriaceae, такие как штамм Е.coli MG1655 (АТСС 47076), штамм W3110 (АТСС 27325), штамм Pantoea ananatis AJ13335 (PERM BP-6614) и т.д.
Термин «повышенная экспрессия гена, кодирующего Lal» также может означать, что уровень экспрессии Lal-кодирующего гена в модифицированном штамме выше, чем уровень экспрессии в немодифицированном штамме, например штамме дикого типа или родительском штамме.
Способы, которые могут применяться для повышения экспрессии Lal-кодирующего гена, включают, но не ограничиваются данными примерами, увеличение числа копий Lal-кодирующего гена в бактериальном геноме на хромосоме и/или на автономно реплицирующейся плазмиде и/или введение Lal-кодирующего гена в вектор таким образом, что он становится способным увеличивать число копий и/или уровень экспрессии Lal-кодирующего гена в бактерии рода Escherichia в соответствии с методами генной инженерии, известными для специалистов в данной области.
В качестве примеров таких векторов можно привести, но не ограничиться этим, векторы с широким кругом хозяев (broad-host-range vectors), например, pCMHO, pRK310, pVK101, pBBR122, pBHR1 и подобные им. Множественные копии Lal-кодирующего гена могут быть введены в хромосомную ДНК бактерии посредством, например, гомологичной рекомбинации, Mu-зависимой интеграции или подобными методами. Гомологичная рекомбинация может быть проведена с использованием многокопийной последовательности в хромосомной ДНК. Многокопийные последовательности в хромосомной ДНК включают, но не ограничиваются данными примерами, повторяющие участки ДНК или обращенные повторы на концах перемещающегося генетического элемента. Также, возможно введение Lal-кодирующего гена в переносимый транспозон с целью введения в хромосомную ДНК множества копий Lal-кодирующего гена. При использовании Mu-зависимой интеграции более чем 3 копии могут быть вставлены в хромосомную ДНК за один акт (Akhverdyan V.Z. et al., Biotechnol. (Russian), 2007, 3:3-20).
Повышение уровня экспрессии Lal-кодирующего гена также может быть достигнуто путем модификации регуляторного участка, смежного с Lal-кодирующим геном, или введением нативных и/или модифицированных чужеродных регуляторных участков. В качестве примеров регуляторных участков или последовательностей можно привести промоторы, энхансеры, аттенюаторы, сигналы терминации и анти-терминации транскрипции, рибосома-связывающие сайты (RBS) и другие элементы контроля экспрессии (например, участки, с которыми связываются репрессоры или индукторы, и/или участки связывания транскрипционных и трансляционных регуляторных белков, например, в составе мРНК). Такие регуляторные последовательности описаны, например, в Sambrook J., Fritsch E.F. and Maniatis Т., «Molecular Cloning: A Laboratory Manual», 2nd ed., Cold Spring Harbor Laboratory Press (1989). Модификации участков, контролирующих экспрессию гена(ов) могут комбинироваться с увеличением числа копий модифицированного гена(ов) в бактериальном геноме с использованием известных методов (см., например, Akhverdyan V.Z. et al., Appl. Microbiol. Biotechnol., 2011, 91:857-871; Tyo K.E.J. et al., Nature Biotechnol., 2009, 27:760-765).
Примерами промоторов, усиливающих экспрессию Lal-кодирующего гена, могут быть сильные промоторы. Например, lac промотер, trp промотер, trc промотер, tac промотер, PR или PL промотеры λ-фага известны как сильные промоторы. Сильные промоторы, обеспечивающие высокий уровень экспрессии гена в бактерии, принадлежащей к семейству Enterobacteriaceae, могут использоваться. Также, влияние промотора может быть усилено, например, введением мутации в участок промотора Lal-кодирующего гена с целью получения промотора с более сильной функцией, таким образом, приводя к увеличению уровня транскрипции Lal-кодирующего гена, расположенного под промотором. Кроме того, известно, что замена нескольких нуклеотидов в области между сайтом связывания рибосомы и стартовым кодоном, особенно в последовательности непосредственно перед стартовым кодоном, существенно влияет на трансляционную способность мРНК. Например, обнаружен 20-кратный разброс в уровне экспрессии гена в зависимости от природы трех нуклеотидов, предшествующих стартовому кодону (Gold L. et al., Annu. Rev. Microbiol., 1981, 35:365-403; Hui A. et al., EMBOJ., 1984, 3:623-629).
Усиление гетерологичной экспрессии Lal-кодирующего гена в клетке микроорганизма-хозяина также может быть осуществлено путем замены редких и/или мало используемых кодонов на синонимичные средне или часто используемые кодоны, при этом термин «использование кодона» может быть определен как число раз (частота) трансляции кодона в клетке организма в единицу времени или как средняя частота встречаемости кодона в сиквенированных белок-кодирующих рамках считывания (Zhang S.P. et al., Gene, 1991, 105(1):61-72). Использование кодонов в организме может быть найдено в базе данных использования кодонов Codon Usage Database, которая является расширенной веб-версией CUTG (Codon Usage Tabulated from GenBank) (http://www.kazusa.or.jp/codon/, Nakamura Y. et al., Codon usage tabulated from the international DNA sequence databases: status for the year 2000, Nucl. Acids Res., 2000, 28(1):292). В Е.coli такие мутации могут включать, не ограничиваясь этим, замену редких Arg-кодонов AGA, AGG, CGG, CGA на CGT или CGC; редкого Ile-кодона АТА на АТС или АТТ; редкого Leu-кодона СТА на CTG, СТС, СТТ, ТТА или TTG; редкого Pro-кодона ССС на CCG или ССА; редкого Ser-кодона TCG на ТСТ, ТСА, ТСС, AGC или AGT; редких Gly-кодонов GGA, GGG на GGT или GGC; и т.д. Замена мало используемых кодонов на синонимичные часто используемые кодоны предпочтительна. Замена редких и/или мало используемых кодонов на синонимичные средне или часто используемые кодоны может комбинироваться с со-экспрессией генов, кодирующих редкие m-РНК, распознающие редкие кодоны.
Количество копий, присутствие или отсутствие гена и/или генов оперона может быть измерено, например, рестрикцией хромосомной ДНК с последующим блоттингом по-Саузерну (Southern blotting), используя зонд, подобранный на основе последовательности гена, флуоресцентную гибридизацию in situ (fluorescence in situ hybridization, FISH) и подобные им методы. Уровень экспрессии гена и/или генов оперона можно определить с помощью различных известных методов, включая Нозерн-блоттинг, количественную ОТ-ПЦР (RT-PCR) и т.п. Дополнительно, уровень экспрессии гена может быть определен измерением количества транскрибируемой мРНК с применением хорошо известных методов, включающих, например, Нозерн-блоттинг, количественную ОТ-ПЦР (RT-PCR) и т.п. Количество белков, кодируемых геном, можно определить известными методами, включая ДДС-ПААГ (SDS-PAGE) с последующим иммуноблоттингом (Вестерн-блоттинг) (Western blotting analysis), или масс-спектрометрический анализ образцов белка и т.п.
Методы приготовления плазмидной ДНК, расщепления, лигирования и трансформации ДНК, выбор олигонуклеотидов в качестве праймеров и т.п. могут осуществляться обычными способами, известными специалисту в данной области. Эти методы описаны, например, в Sambrook J., Fritsch E.F. и Maniatis Т., «Molecular Cloning: A Laboratory Manual, 2nd ed.», Cold Spring Harbor Laboratory Press (1989). Методы молекулярного клонирования и экспрессии гетерологичных генов описаны в Bernard R. Glick, Jack J. Pasternak и Cheryl L. Patten, «Molecular Biotechnology: principles and applications of recombinant DNA», 4th ed., Washington, D.C: ASM Press (2009); Evans Jr., T.C. and Xu M.-Q., «Heterologous gene expression in E.coli», 1st ed., Humana Press (2011).
Термин «функционально связанный с геном» может означать, что одна или несколько регуляторных последовательностей связаны с нуклеотидной последовательностью молекулы нуклеиновой кислоты или заданного гена таким образом, что возможна экспрессия (например, усиленная, увеличенная, конститутивная, базальная, ослабленная, сниженная или репрессивная экспрессия) нуклеотидной последовательности, предпочтительно экспрессии продукта гена, кодируемого нуклеотидной последовательностью.
Бактерия, описанная в настоящем изобретении, может быть получена путем сообщения требуемых свойств бактерии, изначально способной продуцировать дипептид. Аналогично, бактерия может быть получена путем сообщения способности продуцировать дипептид бактерии, которая уже имеет требуемые свойства.
Помимо упомянутых выше свойств, бактерия также может обладать другими специфическими свойствами, не выходя за рамки данного изобретения, такими как: нуждаться в различных питательных веществах, обладать чувствительностью, устойчивостью и зависимостью от антибиотиков.
3. Способы получения дипептидов
Способами настоящего изобретения могут быть способы получения дипептида, более точно дипептида, имеющего кислую L-аминокислоту на N-конце, с использованием L-аминокислота-α-лигазы (Lal) или бактерии, принадлежащей к семейству Enterobacteriaceae, модифицированной таким образом, чтобы содержать указанную Lal, и, таким образом, названные как ферментативный способ и способ бактериальной ферментации, соответственно.
Термин «аминокислота» может означать обычную аминокислоту, известную для специалиста данной области, производную аминокислоты или ее соль. Например, аминокислота может быть α-аминокислотой и β-аминокислотой, имеющей Cα или Cβ хиральный атом углерода, соответственно, к которому присоединены аминогруппа, карбоксильная группа и группа боковой цепи. Примером β-аминокислоты может быть βAla. α-Аминокислоты могут быть протеиногенными и непротеиногенными аминокислотами. Протеиногеными аминокислотами могут быть L-аминокислоты, такие как L-аланин, L-аргинин, L-аспарагин, L-аспарагиновая кислота, L-цистеин, глицин, L-глутаминовая кислота, L-глутамин, L-гистидин, L-изолейцин, L-лейцин, L-лизин, L-метионин, L-фенилаланин, L-пролин, L-серин, L-треонин, L-триптофан, L-тирозин и L-валин, или их соли, имеющие Cα хиральный атом углерода. Аминокислоты могут использоваться в защищенной и незащищенной форме. Защищенная форма аминокислот, в отличие от незащищенной формы, может означать наличие одного или более заместителей, связанных с аминогруппой, карбоксигруппой и/или группой боковой цепи. Аминокислоты, имеющие заместитель(и), могут быть отнесены к производным аминокислот. Примерами производных аминокислот могут быть низшие алкильные эфиры аминокислот, такие как низший эфир L-фенилаланина. К низшим эфирам относятся метиловый эфир, этиловый эфир, пропиловый эфир и т.п.
Термин «аминокислота» эквивалентен термину «субстрат реакции, катализируемой Lal» или просто «субстрат».
Термин «кислая L-аминокислота» может означать аспарагиновую кислоту (Asp) или глутаминовую кислоту (Glu) L-формы или их соли.
Термин «дипептид» может означать органическую молекулу или ее соль, состоящую из двух аминокислотных остатков, или остатков двух производных аминокислот, или их сочетание, как указано выше. Например, термин «дипептид» может означать дипептид, образованный двумя протеиногенными L-аминокислотными остатками таким образом, что остаток кислой L-аминокислоты, такой как L-Asp или L-Glu, расположен на N-конце дипептида, а другой остаток L-аминокислоты такого же или другого вида расположен на С-конце дипептида. Также принято, что производные аминокислоты, например низший алкильный эфир L-аминокислоты, такой как метиловый эфир L-Phe, может быть расположен на С-конце дипептида.
Дипептид, описанный здесь, не ограничивается дипептидом, имеющим кислые аминокислотные остатки на N-конце. Дипептид может быть представлен формулой R1-R2, где R1 и R2 могут означать аминокислотные остатки или их производные, расположенные на N- и С-концах соответствующих дипептидов и соединенные посредством пептидной связи. Примерами R1 и R2 могут быть L-аминокислоты, такие как L-Ala, L-Arg, L-Asp, L-Asn, L-Cys, Gly, L-Glu, L-Gln, L-His, L-Ile, L-Leu, L-Lys, L-Met, L-Phe, L-Pro, L-Ser, L-Thr, L-Trp, L-Tyr и L-Val, или их производные, такие как L-PheOMe, или их соли. R1 и R2 могут быть одинаковыми или отличаться.
Термин «пептидная связь» может означать ковалентную химическую связь -C(O)NH-, образующуюся между двумя молекулами, когда карбоксильная часть одной молекулы, карбокси компонент, реагирует с аминной частью, аминный компонент, вследствие чего образуется молекула. Например, аминокислоты могут образовывать пептидные связи, взаимодействуя друг с другом и с образованием молекулы воды.
Любой карбоксильный компонент может использоваться при условии, что он способен образовывать пептид при конденсации с аминной компонентой другого субстрата. Примеры карбоксильного компонента включают эфиры L-аминокислот, эфиры D-аминокислот, амиды L-аминокислот и амиды D-аминокислот, а также эфиры органических кислот, не имеющие незащищенную аминогруппу. Примеры эфиров аминокислот включают не только эфиры аминокислот, соответствующих природным аминокислотам, но также эфиры аминокислот, соответствующие неприродным аминокислотам или их производные. Также, примеры эфиров аминокислот включают эфиры α-аминокислот, равно как и эфиры β-, γ- и ω-аминокислот и подобные им, имеющие различные сайты, связывающие аминогруппы. Типичные примеры эфиров аминокислот включают метиловые эфиры, этиловые эфиры, изо-пропиловые эфиры, н-бутиловые эфиры, изобутиловые эфиры, трет-бутиловые эфиры, фениловые эфиры аминокислот. Также, карбоксильная часть карбоксильного компонента может быть представлена карбоксильной группой СООН или ее производной COR, где R может означать галогенид-группу, такую как, например, хлор.
Любой аминный компонент может использоваться при условии, что он способен образовывать пептид при конденсации с карбоксильным компонентом другого субстрата. Примеры аминной компоненты включают L-аминокислоты, C-защищенные L-аминокислоты, D-аминокислоты, C-защищенные D-аминокислоты и амины. Также, примеры аминокислот включают не только природные аминокислоты, но и аминокислоты неприродного происхождения или их производные. Они включают α-аминокислоты, равно как и β-, γ- или ω-аминокислоты и подобные им, имеющие сайты, связывающие карбоксигруппы.
3.1 Ферментативный способ
Ферментативный способ может включать по меньшей мере одну стадию, позволяющую контактировать L-аминокислота-α-лигазе или Lal-содержащему веществу с одной или несколькими аминокислотой(ами) одного вида или различных видов, или их производных, или их солей, в приемлемых условиях, чтобы получить продукт реакции в соответствии с активностью Lal как описано выше.
Способ, допускающий воздействие Lal или Lal-содержащего вещества согласно настоящему изобретению, может быть смешение друг с другом Lal или Lal-содержащего вещества, молекулы с карбоксильной частью и молекулы с аминной частью. Более точно, может быть использован способ добавления Lal или Lal-содержащего вещества к раствору, содержащему карбоксильный и аминный компоненты, допускающий протекание реакции между компонентами для образования дипептида. Также, в случае использования бактерии, которая продуцирует Lal, данный способ может включать выращивание бактерии, которая продуцирует Lal, продукция и накопление Lal в бактерии или культуральной жидкости и затем добавление молекулы с карбоксильным компонентом и молекулы с аминной компонентой в среду. Полученный дипептид может быть выделен известными способами и очищен, если необходимо.
Термин «Lal-содержащее вещество» может означать любое вещество при условии, что оно содержит Lal. Примеры Lal-содержащего вещества включают культуру бактерий, которые продуцируют Lal, бактериальные клетки, изолированные из культуры, и продукт, полученный в результате обработки бактериальных клеток (также можно употребить как «обработанный бактериальный клеточный продукт»). Культура бактерий может означать то, что получается при выращивании бактерии и, более точно, смесь бактериальных клеток, среды, используемой для выращивания бактерий, вещества, продуцируемого выращиваемыми бактериями, и т.д. Бактериальные клетки могут быть промыты и использованы в форме промытых клеток. Обработанный бактериальный клеточный продукт включает продукты разрушения, лизиса или лиофилизированные бактериальные клетки и подобные им, а также неочищенный фермент, получаемый в результате обработки бактериальных клеток, и т.п., а также очищенный фермент, получаемый при очистке неочищенного фермента, и т.п. Частично очищенный фермент, полученный различными способами очистки, может быть использован в качестве очищенного фермента; или иммобилизованные ферменты могут быть использованы, которые были иммобилизованы способом ковалентного связывания, адсорбции, захвата и подобными им. Ввиду того что некоторые бактерии частично разрушаются в процессе выращивания в зависимости от используемого микроорганизма, внеклеточная жидкость (супернатант) также может использоваться как фермент-содержащее вещество.
Штаммы дикого вида могут использоваться в качестве бактерий, которые содержат Lal, или же могут использоваться рекомбинантные штаммы, экспрессирующие Lal как описано выше. Бактерии не ограничены интактными бактериальными клетками, но также могут использоваться бактериальные клетки, обработанные ацетоном, лиофилизированные бактериальные клетки или обработанные другим способом бактериальные клетки. Также могут использоваться иммобилизованные бактериальные клетки или продукт иммобилизованных обработанных бактериальных клеток, полученный иммобилизацией бактериальных клеток, или продукт обработанных бактериальных клеток, полученный путем ковалентного связывания, адсорбции, захвата или другими способами, а также иммобилизованные обработанные бактериальные клетки.
Когда используются клеточные культуры, выращиваемые бактериальные клетки, промытые бактериальные клетки или продукт промытой бактериальной клетки, который был получен при разрушении или лизисе бактериальных клеток, часто случается, что полученные пептиды расщепляются содержащимися в них ферментами, которые не вовлечены образование пептида. В этой ситуации может быть предпочтительно в некоторых случаях добавлять ингибитор металлопротеаз, такой как этилендиаминтетрауксусную кислоту (ЭДТА). Добавляемое количество может быть в области от 0,1 мМ до 300 мМ или более предпочтительно в области от 1 мМ до 100 мМ.
Примером ферментативного способа настоящего изобретения является способ, в котором трансформированные клетки, описанные здесь, выращены в культуральной жидкости с целью накопления пептид-образующего фермента (Lal) в культуральной жидкости и/или трансформированных клетках. Так как пептид-образующий фермент может быть легко получен в больших количествах с использованием трансформанта, дипептиды также могут быть получены быстро и в большом количестве.
Количество используемого Lal или Lal-содержащего вещества может быть достаточным, если это такое количество, при котором проявляется ожидаемый эффект (эффективное количество), и это эффективное количество может быть легко определено специалистом, знакомым с данной областью техники, в простом предварительном эксперименте. В случае использования Lal, например, количество, которое может быть использовано, составляет от 0,1 г/л до 10 г/л (см. Пример 3), в то время как в случае использования промытых бактериальных клеток использующееся количество может быть выше, что определяется количеством Lal в бактериальной клетке.
Термин «приемлемые условия» может означать условия, при которых может протекать Lal-катализируемая реакция; т.е. продукт реакции, например дипептид, может образовываться из карбоксильного компонента и аминного компонента. Термин «приемлемые условия» может включать, без ограничений, термины «фермент», «аминокислота», «субстрат», «приемлемый растворитель», «макроэргическая молекула», «приемлемые температурные условия» и т.д.
Термин «макроэргическая молекула» может означать любую органическую или неорганическую молекулу, необходимую для протекания Lal-катализируемой реакции в приемлемых условиях. Традиционно, кофакторы могут быть примером макроэргических молекул. Более точно, примером макроэргической молекулы может быть аденозин-5′-трифосфат (АТФ) или его соль. Могут использоваться соли натрия, калия, аммония и подобные им в любых их комбинациях.
Термин «приемлемый растворитель» может означать любой растворитель, в котором протекает Lal-катализированная реакция так, что продукт реакции, например дипептид, может быть образован. Органические и водные растворители или их смеси в различных соотношениях могут быть примерами приемлемого растворителя. Приемлемый растворитель может содержать Lal-фермент согласно настоящему изобретению; кофактор, такой как АТФ и т.п.; катионы, такие как ионы натрия, калия, аммония, кальция и магния и подобные им; анионы, такие как сульфат, хлорид, фосфат ионы и подобные им; другие неорганические и/или органические молекулы, необходимые для активности L-аминокислоты-α-лигазы. Трис(гидроксиметил)-аминометан (трис), N-трис(гидроксиметил)-метилглицин (трицин) или N,N-бис(2-гидроксиэтил)глицин (бицин) или подобные им, описанные в Carmody W.R. J. Chem. Educ., 1961, 38(11):559-560, могут быть добавлены в реакционную смесь в качестве буферных агентов. Кислотность реакционной смеси (рН) может находиться в интервале между 6.5 и 10.5, или между 7.0 и 10.0, или между 7.5 и 9.5, или между 8 и 9. Приемлемый растворитель может находиться в приемлемых температурных условиях.
Термин «приемлемые температурные условия» может означать температурные условия, при которых может протекать Lal-катализируемая реакция так, что может быть образован продукт реакции, например дипептид. Приемлемые температурные условия могут быть между 0 и 60°С, или 20 и 40°С, или 25 и 37°С, или 28 и 35°С.
Концентрация карбоксильного компонента и аминного компонента, использующихся в качестве исходных соединений, может быть от 1 мМ до 10 М и предпочтительно от 50 мМ до 2 М, соответственно; однако есть случаи, в которых предпочтительно добавлять аминный компонент в эквимолярном количестве или избытке по отношению к карбоксильному компоненту. В тех случаях, когда высокая концентрация субстратов ингибирует реакцию, они могут быть добавлены последовательно в ходе реакции до тех пор, пока концентрация этих субстратов не начнет вызывать ингибирование.
После того, как дипептид получен и накоплен в приемлемом растворителе в необходимом количестве, твердые частицы, такие как клетки, клеточный дебрис и денатурированные белки могут быть удалены из культуральной жидкости центрифугированием или мембранной фильтрацией, и затем целевой дипептид может быть извлечен из приемлемого растворителя с помощью любой комбинации известных способов, таких как концентрация, ионообменная хроматография, высоко-эффективная жидкостная хроматография (ВЭЖХ), кристаллизация и т.п.
Выделение и очистка фермента
L-Аминокислота-α-лигаза может быть выделена из бактерии, принадлежащей к роду Escherichia, например, к виду Е.coli. Способ накопления, выделения и очистки Lal из бактерии может быть обычным способом, известным для специалиста данной области.
Бактерия выращивается в культуральной жидкости, как описано здесь, чтобы продуцировать Lal. Экстракт бактериальных клеток может быть приготовлен из клеток путем разрушения клеток с использованием физических способов, таких как ультразвуковое разрушение, или ферментативного способа с использованием ферментов, разрушающих клетки, и удалением нерастворимой фракции центрифугированием и т.п. Пептид-образующие белки могут быть очищены далее фракционированием раствора экстракта бактериальных клеток, полученного вышеуказанным способом с сочетанием обычных способов очистки белка, таких как анионообменная хроматография, катионообменная хроматография или гель-фильтрация.
Примерами носителей для анионообменной хроматографии могут быть Q-Sepharose HP или ДЭАЭ агароза (GE Healthcare) и т.п. Фермент может быть извлечен из несвязанной фракции в условиях нейтрального показателя рН (7-8), когда клеточный экстракт, содержащий ферменты, пропускают через колонку, заполненную носителем. В зависимости от носителя могут использоваться различные элюенты. Например, если при катионообменной хроматографии применяется MonoS HR (GE Healthcare), клеточный экстракт, содержащий фермент, пропускают сквозь колонку, наполненную носителем, при этом для элюирования фермента колонку промывают буферным раствором, имеющим высокую концентрацию соли. В то же время концентрация соли может последовательно увеличиваться, или может быть применен градиент концентрации. В качестве физиологического раствора может использоваться NaCl от 0 до 0.5 М. Примерами носителей для гель-фильтрации могут быть Superdex 200 HR или Sephadex 200 (GE Healthcare).
В вышеупомянутом способе очистки каждая фракция, содержащая фермент, может быть проверена на Lal-активность в соответствии со способом, указанным в описанных ниже Примерах.
3.2. Способ бактериальной ферментации
Способ, в соответствии с настоящим изобретением, также может быть способ для получения дипептида, более точно, дипептида, имеющего кислую L-аминокислоту на N-конце, путем выращивания бактерии настоящего изобретения в культуральной жидкости с целью продукции, экскреции и накопления дипептида в культуральной жидкости, и выделения дипептида из культуральной жидкости.
Выращивание бактерии согласно настоящему изобретению, накопление и очистка дипептида от среды и т.п. может быть проведено способом, подобным традиционным способам ферментации, отличающимся тем, что дипептид или аминокислота продуцируются с использованием микроорганизма. Питательная среда для получения дипептида может быть типичной средой, которая содержит источник углерода, источник азота, неорганические ионы и другие необходимые органические компоненты. В качестве источника углерода могут использоваться сахара, такие как глюкоза, лактоза, галактоза, фруктоза, арабиноза, мальтоза, ксилоза, трегалоза, рибоза и гидролизаты крахмала; спирты, такие как глицерин, маннитол и сорбитол; органические кислоты, такие как глюконовая кислота, фумаровая кислота, лимонная кислота, яблочная кислота и янтарная кислота и т.п. В качестве источника азота могут использоваться неорганические аммониевые соли, такие как сульфат аммония, хлорид аммония, фосфат аммония; органические питательные вещества, такие как гидролизаты соевых бобов; аммиачный газ; водный раствор аммиака и т.п. Витамины, такие как витамин В1, необходимые вещества, например органические питательные вещества, такие как нуклеиновые кислоты, такие как аденин и РНК или экстракт дрожжей и подобные им могут присутствовать в соответствующих количествах или в виде следов. Помимо перечисленных, небольшие количества фосфата кальция, сульфата магния, ионов железа, ионов марганца и подобные им могут быть добавлены при необходимости.
Чтобы увеличить дипептид-продуцируюшую способность бактерии, представленной в настоящем изобретении, питательная среда может быть дополнительно насыщена аминокислотами, производными аминокислот, кофакторами и другими (био)химическими веществами. Например, чтобы увеличить способность бактерии продуцировать дипептид Asp-Phe, в питательную среду могут быть добавлены дополнительные количества L-Phe и L-Asp.
Выращивание осуществляют предпочтительно в аэробных условиях от 16 до 72 часов, температура выращивания, в интервале которой выращивание может контролироваться - от 15 до 45°С, или в интервале от 28 до 37°С. Кислотность среды (рН) поддерживают в интервале 5-8, или в интервале 6,5-7,2 с использованием неорганических или органических кислых или щелочных веществ, таких как газообразный аммиак.
После выращивания твердые частицы, такие как клеточный дебрис, могут быть удалены из жидкой среды центрифугированием или мембранной фильтрацией и затем целевой дипептид может быть выделен из ферментативной смеси сочетанием известных способов, таких как концентрация, ионообменная хроматография, высокоэффективная жидкостная хроматография (ВЭЖХ), кристаллизация и т.п.
Примеры
Настоящее изобретение более подробно будет описано ниже со ссылкой на следующие, не ограничивающие настоящее изобретение Примеры.
Пример 1.
Клонирование BBR47_51900 из Brevibacillus brevis NBRC 100599 и Staur_4851 из Stigmatella aurantiaca DW4/3-1.
Первичная структура генов, кодирующих гипотетические белки BBR47_51900 и Staur_4851, была оптимизирована для экспрессии в Е.coli. Гены, кодирующие BBR47_51900 из Brevibacillus brevis NBRC 100599 и Staur_4851 из Stigmatella aurantiaca DW4/3-1 были синтезированы с помощью сервиса синтеза генов SlonoGene™ (http://www.sloning.com/) и доставлены в качестве набора плазмид pSlo.X, содержащих синтезированный фрагмент XbaI-EcoRI, который включает целевые гены, имеющие оптимизированные последовательности. Фрагменты XbaI-EcoRI, содержащие гены с оптимизированными последовательностями, кодирующими белки BBR47_51900 и Staur_4851, представлены в SEQ ID NOs:19 и 20, соответственно.
Для конструкции плазмид pET-HT-BBR и pET-HT-STA, соответствующие фрагменты XbaI-EcoRI плазмид pSlo.X были удалены путем расщепления с помощью XbaI и EcoRI и затем лигированы с вектором pET15(b+) (Novagen, USA), расщепленным такими же рестриктазами.
Пример 2.
Экспрессия и очистка His6-меченного BBR47_51900 и Staur_4851.
Плазмиды pET-HT-BBR и pET-HT-STA вводили в штамм BL21 (DE3) (Novagen, USA) методом Ca2+-зависимой трансформации для конструкции штаммов BL21 (DE3) [pET-HT-BBR] и BL21 (DE3) [pET-HT-STA]. Электротрансформацию проводили с использованием электропоратора "Bio-Rad" (USA) (№165-2098, версия 2-89) в соответствии с инструкциями производителя.
Клетки каждого из штаммов BL21 (DE3) [pET-HT-BBR] и BL21 (DE3) [pET-HT-STA] выращивали в среде LB (также упоминается как лизогенная среда, как описано в Sambrook, J. and Russell, D.W. (2001) Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor Laboratory Press) при 37°С и 150 об/мин, до OD540 ~1. Изопропил-β-D-тио-галактозил (ИПТГ) добавляли в конечной концентрации 1 мМ, и клеточную культуру инкубировали 2 часа при 37°С и 150 об/мин. Индуцированные клетки извлекали из 1 л ферментационной среды, ресуспендировали в 60-80 мл НТ-I-буфера (20 мМ NaH2PO4, 0,5 М NaCl, 20 мМ имидазола, рН поддерживали в районе 7,4 раствором KOH) и расщепляли при 2000 Psi (~140 бар) с использованием French-press (Thermo Spectronic). Дебрис удаляли центрифугированием в две стадии: при 4°С и 13000 об./мин, затем следовала фильтрация через фильтр 0,45 мкм (CHROMAFIL Xtra СА-45/25, MACHEREY-NAGEL GmbH). Раствор неочищенного белка загружали на колонку HiTrap Chelating (GE Healthcare), заполненную хромато графическим сорбентом (IMAC), обладающим иммобилизованным сродством к металлу, на 1 мл общего объема и откалиброванную HT-I-буфером. IMAC использовали в соответствии с рекомендациями производителя. Активные фракции собирали, объединяли и обессоливали с использованием колонки PD10 (GE Healthcare), откалиброванной SB-буфером (20 мМ Трис-HCl, 120 мМ NaCl, 1 мМ β-меркаптоэтанол, 15% глицерина, рН 7,5). Собранный белок разделяли на аликвоты объемом 200 мкл и сохраняли при -70°С. Концентрацию белка определяли с использованием BIO-RAD PROTEIN ASSAY (BIO-RAD, USA).
Пример 3.
Предварительный анализ на субстратную специфичность BBR47_51900 и Staur_4851.
Субстратную специфичность BBR47_51900 и Staur_4851 изучали в реакционной среде, содержащей фермент и одинаковые или две различные канонические L-аминокислоты, такие как L-аланин, L-аргинин, L-аспарагин, L-аспарагиновую кислоту, L-цистеин, глицин, L-глутаминовую кислоту, L-глутамин, L-гистидин, L-изолейцин, L-лейцин, L-лизин, L-метионин, L-фенилаланин, L-пролин, L-серин, L-треонин, L-триптофан, L-тирозин и L-валин или их соли.
Состав реакционной смеси общим объемом 50 мкл был, как показано ниже, если не указано иное:
|
Реакцию проводили при 32°С в течение 15 часов. Порции реакционной смеси объемом 1-2 мкл исследовали с помощью тонкослойной хроматографии (ТСХ) с использованием в качестве подвижной фазы смесь 2-пропанол:ацетон:250 мМ аммиак:H2O как 100:100:12:28. Раствор (0,3%, масс./об.) нингидрина в ацетоне применяли в качестве окрашивающего реактива. Определение проводили при 540 нм. Пластины ТСХ проявляли после обработки реакционных смесей, содержащих:
1) BBR47_51900 и L-Glu/L-Glu, или L-Glu/L-Asp, или L-Glu/L-Val, или L-Glu/L-Ile, или L-Asp/L-Ile, или L-Asp/L-Val (Фигуры 2-4);
2) Staur_4851 и L-Asp/L-Phe или L-Asp/L-Trp или L-Asp/L-Thr) (Фигуры 5 и 6).
Полученные результаты указывают на то, что BBR47_51900 и Staur_4851 катализируют лигирование кислых L-аминокислот, таких как L-Glu и L-Asp, с другими L-аминокислотами.
Пример 4.
Определение методом ВЭЖХ дипептидов, синтезированных с помощью BBR47_51900 и Staur_4851.
Полученные при помощи BBR47_51900 и Staur_4851 дипептиды определяли с использованием метода ВЭЖХ в реакционной смеси общим объемом 400 мкл, которая содержала:
|
где Me означает метильную группу.
Реакции проводили при 32°С в течение 15 часов. Затем 0,5 мл реакционной смеси отфильтровывали через Amicon Ultra-0.5 мл, 3K Centrifugal Filters (Millipore, #UFC500396) и анализировали при помощи ВЭЖХ.
Условия анализа были следующие:
Оборудование: HITACHI L-2000 series.
Колонка: Inertsil ODS-3 4,6×250 мм, 5 мм (GL Sciences Inc.).
Температура: 40°С.
Буферы:
А (для смеси L-Asp/L-Val и L-Glu/L-Val): 0,1 M KH2PO4 (pH 2,2) + 5 мМ октансульфонат натрия: CH3CN как 4:1 (об./об.),
В (для смеси L-Asp/L-Phe): 0,1 M KH2PO4 (pH 2,2) + 5 мМ октансульфонат натрия: CH3CN как 7:3 (об./об.),
С (для смеси L-Asp/L-PheOMe, L-Asp/L-Trp и L-Val/L-Val): 0,1 M KH2PO4 (pH 2,2) + 5 мМ октансульфонат натрия: CH3CN как 3:2 (об./об.),
D (для смеси L-Phe/L-Phe): 0,1 M KH2PO4 (pH 2,2) + 5 мМ октансульфонат натрия: CH3CN как 1:1 (об./об.).
Профиль градиента: изократический.
Скорость потока: 1,5 мл/мин.
Вводимый объем: 10 мкл.
Область определения: УФ 210 нм.
Химические реактивы, которые применялись для метода ВЭЖХ:
L-Asp (L-аспарагиновая кислота, натриевая соль): Nacalai Tesque, Inc. #03504-75
L-Phe (L-фенилаланин): Nacalai Tesque, Inc. #26901-35
L-Val (L-валин): Ajinomoto Co., Inc. #317LG13
L-Trp (L-триптофан): Ajinomoto Co., Inc. #0000002205
L-PheOMe (гидрохлорид метилового эфира L-фенилаланина): Tokyo Chemical Industry Co., Ltd. #P1278
АТФ: Oriental yeast Co., Ltd. #45142000
MgSO4×7H2O: Junsei Chemical Co., Ltd. #83580-0301
αAsp-Phe (H-Asp-Phe-OH): Bachem G-1620
βAsp-Phe (H-Asp(Phe-OH)-OH): Bachem G-4750
αAsp-Asp (H-Asp-Asp-OH): Bachem G-1565
Phe-Asp (H-Phe-Asp-OH): Bachem G-2870
Phe-Phe (H-Phe-Phe-OH): Bachem G-2925
αAsp-Val (H-Asp-Val-OH): Bachem G-1635
Val-Asp (H-Val-Asp-OH): Bachem G-3510
Val-Val (H-Val-Val-OH): Bachem G-3595
αGlu-Val (H-Glu-Val-OH): Bachem G-2010
γGlu-Val (H-Glu(Val-OH)-OH): Bachem G-2015
Val-Glu (H-Val-Glu-OH): Bachem G-3520
αGlu-Glu (H-Glu-Glu-OH): Bachem G-1915
αAspartame (H-Asp-Phe-OMe): Bachem G-1545
βAspartame: (H-Asp(Phe-OMe)-OH): Bachem G-3725
Asp-Trp (H-Asp-Trp-OH): Bachem G-3705.
Были приготовлены растворы αGlu-Val, γGlu-Val, αAsp-Val (10 мМ каждого) и αAsp-Phe, βAsp-Phe (5 мМ каждого) для анализа методом ВЭЖХ. Концентрацию полученного дипептида определяли с использованием соответствующих калибровочных кривых. Для метода LC-QTOF/MS/MS каждый раствор со стандартным образцом растворяли 50-кратно или 100-кратно (Example 5).
Результаты метода ВЭЖХ для реакционной смеси представлены в Таблице 1. Как видно из Таблицы 1, BBR47_51900 и Staur_4851 катализируют образование дипептида, имеющего кислые L-аминокислоты, такие как L-Asp и L-Glu, на N-конце.
Пример 5.
Определение методом LC-QTOF/MS/MS дипептидов, полученных с помощью BBR47_51900.
Образцы реакционных смесей и стандартные растворы, полученные, как описано в Примере 4, исследовали методом LC-QTOF/MS/MS.
Условия анализа были следующие:
Оборудование: LC (Agilentl200SL), MS (Micromass Q-TOF Premier)
LC условия:
Колонка: Develosil C30 2.0×250 мм, 3 мкм (Nomura Chemical).
Температура: 20°С.
Буферы:
А: H2O (0,025% муравьиная кислота),
В: CH3CN (0,025% муравьиная кислота).
Профиль градиента:
|
Скорость потока: 0,3 мл/мин.
Вводимый объем: 2-5 мкл.
MS условия:
Капиллярное напряжение: 3,0 кВ.
Напряжение на игле: 20 В.
Напряжение столкновения: 4 В (MS/MS 12B).
Температурный источник: 80°С.
Температура испарения: 120°С.
Скорость газового потока на игле: 50 л/ч.
Скорость газового потока десольвации: 700 л/ч.
Результаты анализа реакционной смеси методом LC-QTOF/MS/MS представлены на Фигурах 7-9. Как видно из Фигур 7-9, BBR47_51900 катализирует образование дипептидов αAsp-Phe, αAsp-Val, и αGlu-Val.
Пример 6.
Поиск ферментов, которые являются изофункциональными к BBR47_51900 и Staur_4851.
Для поиска в базе данных гомологичных белковых последовательностей и для выравнивания белковых последовательностей использовали метод HMMER. Метод основан на вероятностной модели, называемой как профиль скрытой модели Маркова (Hidden Markov model profile, профиль HMM) (Finn R.D. et al., HMMER web server: interactive sequence similarity searching, Nucleic Acids Res., 2011, 39 (Web Server issue):W29-37).
По сравнению с компьютерными алгоритмами BLAST, FASTA и другими программами выравнивания последовательностей и поиска в базах данных последовательностей, основанных на устаревшей методологии, метод HMMER достиг значительно более точного и более надежного определения сильно отличающихся гомологии, таких как изофункциональные белки, благодаря эффективности математических моделей, лежащих в основе метода. Ранее эффективность достигалась при значительных вычислительных затратах, но в новом проекте HMMER3, HMMER стал значительно быстрее, как и BLAST (Finn R.D. et al., HMMER web server: interactive sequence similarity searching, Nucleic Acids Res., 2011, 39 (Web Server issue):W29-37).
Для того чтобы найти ферменты, которые являются изофункциональными BBR47_51900 и Staur_4851, проводили выравнивание BBR47_51900 и Staur_4851 (Фигура 10) с помощью программы поиска HMMsearch из набора программ HMMER3, которые позволяют искать один или более профилей против базы данных белковых последовательностей (http://hmmer.janelia.org/). Был создан профиль НММ, основанный на выравнивании последовательностей BBR47_51900 и Staur_4851 (Модель 1), который использовался для поиска по гомологии. Список найденных ближайших изофункциональных белков (hits) показан на Фигуре 11. Анализ диаграммы распределения значений |Log(E-value)| показал, что значения |Log(E-value)| распределены по пяти группам белков, причем указанные значения между членами групп были ниже, чем те же значения между группами (Фигура 12). Первая группа включала BBR47_51900 и Staur_4851, вторая группа включала DES и ВСЕ, третья группа включала BMY, четвертая группа включала ВТН и пятая группа включала BUR. Оставшиеся белки являются набором несгруппированных гомологов, имеющих стохастические значения |Log(E-value)|. Белки АМЕ и SFL были выбраны из несгруппированных гомологов как отрицательный контроль.
При условии, что белки из первой (BBR47_51900, Staur_4851) и второй (DES и ВСЕ) групп являются изофункциональными, может быть получен новый профиль НММ (Модель 2), основанный на выравнивании BBR47_51900, Staur_4851, DES и ВСЕ (Фигура 13); он может быть использован для поиска изофункциональных белков с применением программы HMMsearch как описано выше. Таким образом, может быть получен новый перечень изофункциональных белков (Фигура 14), который описывается диаграммой распределения значений |Log10(E-value)|, отличающейся от начальной диаграммы (Фигура 15). Новый перечень изофункциональных белков может включать три группы, такие как первая группа, включающая BBR47_51900, Staur_4851, DES и ВСЕ; вторая группа, включающая BMY и ВТН; третья группа, включающая BUR, отличающаяся тем, что АМЕ находится ближе к первой группе (номер положения изменился с №47 (Фигура 12) на №33 (Фигура 15)) и SFL находится далее от первой группы (номер положения изменился с №49 (Фигура 12) на №62 (Фигура 15)).
При условии, что белки из первой группы (BBR47_51900, Staur_4851, DES и ВСЕ) и второй группы (такой как BMY) являются изофункциональными, новый профиль НММ (Модель 3), основанный на выравнивании BBR47_51900, Staur_4851, DES, ВСЕ и BMY (Фигура 16), может быть получен; он может использоваться для поиска изофункциональных белков, с применением программы HMMsearch, как описано выше. Таким образом, может быть получен новый перечень изофункциональных белков (Фигуры 17). Новый перечень изофункциональных белков может включать три группы, такие как первая группа, включающая BBR47_51900, Staur_4851, DES, ВСЕ и BMY; вторая группа, включающая ВТН; третья группа, включающая BUR, отличающаяся тем, что АМЕ присутствует в положении №37 и SFL присутствует в положении №65 (Фигура 17).
При условии, что белки из первой группы (BBR47_51900, Staur_4851, DES, ВСЕ и BMY) и второй группы (ВТН) являются изофункциональными, может быть получен новый профиль НММ (Модель 4), основанный на выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН (Фигура 18), который может использоваться для поиска изофункциональных белков, с применением программы HMMsearch как описано выше. Таким образом, новый перечень изофункциональных белков может быть получен (Фигура 19). Новый перечень изофункциональных белков может включать три группы, такие как первая группа, которая включает BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН; вторая группа, которая включает BUR; третья группа, которая включает АМЕ, отличающаяся тем, что SFL находится в положении №73 (Фигура 19).
При условии, что белки из первой группы (BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН) и второй группы (BUR) являются изофункциональными, может быть получен новый профиль НММ (Модель 5), основанный на выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR (Фигура 20), который может использоваться для поиска изофункциональных белков с использованием программы HMMsearch как описано выше. Таким образом, может быть получен новый перечень изофункциональных белков (Фигура 21). Новый перечень изофункциональных белков может включать три группы, такие как первая группа, включающая BUR; вторая группа, включающая BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН; третья группа, включающая АМЕ, отличающаяся тем, что SFL находится в положении №104 (Фигура 21).
При условии, что белки из первой группы (BUR), второй группы (BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН) и третьей группы (АМЕ) являются изофункциональными, может быть получен новый профиль НММ (Модель 6), основанный на выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR и АМЕ (Фигура 22), который может использоваться для поиска изофункциональных белков с использованием программы HMMsearch как описано выше. Таким образом, может быть получен новый перечень изофункциональных белков (Фигура 23). Новый перечень изофункциональных белков может включать две группы, такие как первая группа, включающая BUR; вторая группа, включающая BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и АМЕ, отличающаяся тем, что SFL находится в положении №65 (Фигура 23).
При условии, что белки из первой группы (BUR) и второй группы (BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и АМЕ) и белок SFL являются изофункциональными, может быть получен новый профиль НММ (Модель 7), основанный на выравнивании BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR, АМЕ и SFL (Фигура 24), который можно использовать для поиска изофункциональных белков с использованием программы HMMsearch как описано выше. Таким образом, новый перечень изофункциональных белков может быть получен (Фигура 25). Новый перечень изофункциональных белков может включать три группы, такие как первая группа, включающая BUR; вторая группа, включающая BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и АМЕ; и третья группа, включающая SFL, который находится в положении №38 (Фигура 25).
Аналогичным способом могут быть получены новые перечни изофункциональных L-аминокислота-α-лигаз, способных синтезировать дипептид, имеющий кислую L-аминокислоту, такую как L-Asp или L-Glu, на N-конце и любую другую L-аминокислоту или ее производную на С-конце. Если белки BBR47_51900 и Staur_4851 используются для построения профиля НММ (Model 1), то значение |Log10(E-value)|≥233 может использоваться для поиска новых изофункциональных Lals; если для построения профиля НММ (Модель 2) используются белки BBR47_51900, Staur_4851, DES и ВСЕ, то значение |Log10(E-value)|≥196 может использоваться для поиска новых изофункциональных Lals; если для построения профиля НММ (Модель 3) используются белки BBR47_51900, Staur_4851, DES, ВСЕ и BMY, то значение |Log10(E-value)|≥182 может использоваться для поиска новых изофункциональных Lals; если для построения НММ профиля (Модель 4) используются белки BBR47_51900, Staur_4851, DES, ВСЕ, BMY и ВТН, то значение |Log10(E-value)|≥175 может использоваться для поиска новых изофункциональных Lals; если для построения НММ профиля (Модель 5) используются белки BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН и BUR, то значение |Log10(E-value)|≥162 может использоваться для поиска новых изофункциональных Lals; если для построения НММ профиля (Модель 6) используются белки BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR и АМЕ, то значение |Log10(E-value)|≥142 может использоваться для поиска новых изофункциональных Lals; и если для построения НММ профиля (Модель 7) используются белки BBR47_51900, Staur_4851, DES, ВСЕ, BMY, ВТН, BUR, АМЕ и SFL, то значение |Log10(E-value)|≥128 может использоваться для поиска новых изофункциональных Lals (Фигура 26), где E-value (Е-значение) является параметром программы HMMsearch (Finn R.D. et al., HMMER web server: interactive sequence similarity searching, Nucleic Acids Res., 2011, 39 (Web Server issue):W29-37).
Пример 7.
Клонирование, экспрессия и очистка ферментов DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY.
Первичная структура гена, кодирующего белки DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, была оптимизирована для экспрессии в Е.coli с использованием функции "Back translation" программы Gene Designer (Villalobos A. et al., Gene Designer: a synthetic biology tool for constructing artificial DNA segments, BMC Bioinformatics, 2006, 7:285). Все конструкции были синтезированы с помощью сервиса синтеза генов SlonoGene™ (http://www.sloning.com/) и доставлены в качестве плазмид pSlo.X, содержащих синтезированный фрагмент XbaI-EcoRI, который включает целевой ген, имеющий оптимизированные последовательности. Фрагменты XbaI-EcoRI, содержащие гены с оптимизированными последовательностями, кодирующими белки DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY, представлены в SEQ ID NOs:21, 22, 23, 24, 25, 26 и 27, соответственно.
Белки DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY могут быть экспрессированы в Е.coli, очищены, а их активность может быть исследована, как описано для BBR47_51900 и Staur_4851 в Примерах 1-5.
Пример 8.
Конструкция пептидаз-дефицитных штаммов Е.coli 1-6Δ.
8.1. Конструкция пептидаз-дефицитных штаммов Е.coli 1-6Δ.
Ген iadA удаляли в штамме Е.coli BW25113 (KEIO collection, штамм № МЕ9062), имеющем мутацию Δpep-В (KEIO collection, штамм № JW2507; The Е.coli Genetic Stock Center, Yale University, New Haven, USA, инвентарный номер CGSC9995) (Е.coli 1Δ штамм). Для этой цели фрагмент ДНК, несущий кассету λattL-cat-λattR, получали методом ПЦР (полимеразная цепная реакция) с использованием праймеров P1 (SEQ ID NO:28) и Р2 (SEQ ID NO:29) и плазмиды pMW118-λattR-cat-λattL в качестве матрицы (Katashkina Zh.I. et al., Mol. Biol. (Mosk.), 2005, 39(5):823-831). Полученный ДНК-фрагмент вводили в штамм Е.coli BW25113(ΔpepB)/pKD46 методом электротрансформации с использованием электропоратора "Bio-Rad" (USA) (№165-2098, версия 2-89) в соответствии с инструкцией производителя. Рекомбинантная плазмида pKD46 (Datsenko K.A. and Wanner B.L., Proc. Natl. Acad. Sci. USA, 2000, 97:6640-6645) с репликоном, обладающим температурной чувствительностью, была использована в качестве донора генов фага λ, ответственных за λRed-зависимую рекомбинантную систему. Плазмида pKD46 может быть интегрирована в Е.coli BW25113(ΔpepB) для получения штамма Е.coli BW25113(ΔpepB)/pKD46 описанным методом (Datsenko K.A. and Wanner B.L., Proc. Natl. Acad. Sci. USA, 2000, 97:6640-6645). Кроме того, штамм Е.coli BW25113(ΔpepB), содержащий рекомбинантную плазмиду pKD46, может быть получен из Е.coli Genetic Stock Center, Yale University, New Haven, USA, инвентарный номер CGSC7739.
Трансформант Е.coli BW25113 (ΔpepB, iadA::λattR-cat-λattL) обладает устойчивостью к хлорамфениколу и вместо iadA содержит на хромосоме маркер устойчивости к хлорамфениколу (CmR-marker), кодируемому геном cat. Маркер CmR вырезали, как описано в работе (Katashkina Zh.I. et al., Mol. Biol. (Mosk.), 2005, 39(5):823-831), для конструкции штамма Е.coli BW25113(ΔpepB, iadA::λattB) (штамм Е.coli 2Δ).
Ген рерЕ удаляли в штамме Е.coli BW25113(ΔрерВ, iadA::λattB). Фрагмент ДНК, несущий кассету λattL-cat-λattR, получали методом ПЦР с использованием праймеров Р3 (SEQ ID NO:30) и Р4 (SEQ ID NO:31) и плазмиды pMW118-λattR-cat-λattL в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli BW25113(ΔpepB, iadA::λattB)/pKD46 методом электротрансформации, как описано выше. Маркер CmR вырезали из трансформанта Е.coli BW25113 (ΔрерВ, iadA::λattB, pepE::λattR-cat-λattL) для конструкции штамма Е.coli BW25113(ΔрерВ, iadA::λattB, рерЕ::λattB) (штамм Е.coli 3Δ).
Ген ybiK удаляли в штамме Е.coli BW25113(ΔрерВ, iadA::λattB, pepE::λattB). Фрагмент ДНК, несущий кассету λattL-cat-λattR, получали методом ПЦР с использованием праймеров Р5 (SEQ ID NO:32) и Р6 (SEQ ID NO:33) и плазмиды pMW118-λattR-cat-λattL в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli BW25113(ΔрерВ, iadA::λattB, pepE::λattB)/pKD46 методом электротрансформации, как описано выше. Маркер CmR вырезали из трансформанта Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattR-cat-λattL) для конструкции штамма Е.coli BW25113(ΔрерВ, iadA::λattB, pepE::λattB, ybiK::λattB) (штамм Е.coli 4Δ).
Ген dapE удаляли в штамме Е.coli BW25113(ΔрерВ, iadA::λattB, pepE::λattB, ybiK::λattB). Фрагмент ДНК, несущий кассету λattL-cat-λattR, получали методом ПЦР с помощью праймеров Р7 (SEQ ID NO:34) и Р8 (SEQ ID NO:35) и плазмиды pMW118-λattR-cat-λattL в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli BW25113(ΔрерВ, iadA::λattB, pepE::λattB, ybiK::λattB)/pKD46 методом электротрансформации, как описано выше. Маркер CmR вырезали из трансформанта Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattB, dapE::λattR-cat-λattL) для конструкции штамма Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattB, dapE::λattB) (штамм Е.coli 5Δ). Таким образом, были получены штаммы Е.coli, имеющие от одного до пяти удаленных генов (штаммы Е.coli 1-5Δ), кодирующих пептидазы.
8.2 Анализ специфической аспартат-пептид-гидролизующей активности в штамме Е.coli 5Δ.
Для анализа специфической аспаратат-пептид-гидролизующей активности был синтезирован искусственный дипептид DP3 (L-Asp-L-5-фтортриптофан) (филиал Института Биоорганической Химии, РАН, Пущино, Российская Федерация). Пептид-гидролизующая активность была исследована in vitro и in vivo.
Для исследований in vitro клетки штаммов Е.coli BW25113 и Е.coli 5Δ выращивали в средах LB и M9-salts + Глюкоза (0.2%, масс./об.) (Sambrook, J. and Russell, D.W. (2001) Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor Laboratory Press) при 37°С до плотности клеток OD595нм ~1. Выращенные клетки высаживали центрифугированием (4°С, 10000 об/мин), ресуспедировали в буфере Е (50 мМ Трис-HCl рН 8,0, 20 мМ NaCl), разрушали ультразвуком, затем центрифугировали (14000 г, 4°С, 20 мин) для удаления клеточного дебриса. Концентрация неочищенного белка может быть определена методом Брэдфорд (Bradford M.M., Anal. Biochem., 1976, 72:248-254), используя в качестве стандарта бычий сывороточный альбумин.
Для исследования DP3-гидролизующей активности использовали неочищенные белки.
Реакционная смесь содержала:
|
Реакционные смеси инкубировали при температуре 37°С в течение различного времени. DP3-Гидролизуюшую активность измеряли методом количественной ТСХ, измеряя количество выделившегося 5-фтортриптофана (Фигура 27, Таблица 4). В качестве подвижной фазы использовали смесь 2-пропанол:ацетон:H2O как 25:25:4. Раствор (0,3%, масс./об.) нингидрина в ацетоне использовали в качестве окрашивающего реагента. Полученные результаты показали, что аспартат-пептид-гидролизующая активность может быть определена в штамме 5Δ, что предполагает наличие неизвестных пептидаз, имеющих DP3-гидролизующую активность в штамме Е.coli 5Δ.
Для изучения in vivo, исследовали токсичность DP3-дипептида для сконструированных пептидаз-дефицитных штаммов Е.coli 1-5Δ (Фигура 28). Клетки штаммов Е.coli BW25113 и Е.coli 1-5Δ выращивали в среде M9-salts, содержащей D-глюкозу или глицерин (0,4%, масс./об.) до плотности OD555нм ~2. Клеточную биомассу растворяли и около 106 клеток помещали на агар с M9-salts, D-глюкозой или глицерином (0,4%, масс./об.) и DP3-дипептидом. Чашки инкубировали при 37°С в течение 48 часов (для D-глюкозы) или 72 часов (для глицерина) (Фигура 29, Таблица 5). Визуальный анализ показал, что делеция пяти известных аспарагин-пептид-гидролизующих ферментов, кодируемых генами рерВ, iadA, рерЕ, ybiK и dapE, понижает токсичность DP3 в штаммах Е.coli 1-5Δ (с 6 мкМ для BW25113 до 30 мкМ для штамма 5Δ, выращенного в среде M9-salts + D-глюкоза). Увеличение концентрации DP3 до 50 мкМ приводило к задержке роста штаммов Е.coli 1-5Δ, что предполагает остаточную внутриклеточную DP3-пептидазную активность.
8.3. Определение остаточных пептидаз в штамме Е.coli 5Δ, имеющих специфическую аспартат-пептид-гидролизующую активность.
Чтобы определить остаточные внутриклеточные пептидазы, имеющие специфическую аспартат-пептид-гидролизующую активность, использовали следующую методику. Клетки штамма Е.coli 5Δ выращивали при 37°С в течение ночи в 4 л среды M9-salts, содержащей D-глюкозу (0,4%, масс./об.). Выращенные клетки высаживали центрифугированием (4°С, 10000 об/мин) и ресуспендировали в 100 мл буфера F (20 мМ Трис-HCl рН 7.5, 20 мМ NaCl).
Очистку проводили следующим способом:
Шаг 1. Клетки расщепляли двукратным пропусканием через French-press (Thermo Spectronic), затем центрифугировали (14000 г, 4°С, 20 мин) для удаления клеточного дебриса. Полученные неочищенные белки загружали на колонку DEAE FF 16/10 (20 мл) (GE Healthcare), откалиброванную буфером F (20 мМ Трис-HCl рН 7,5, 20 мМ NaCl). Элюирование проводили при скорости потока 1 мл/мин с применением линейного градиента NaCl (от 20 до 600 мМ в 20 объемах колонки) в буфере F. Фракции (10 мл каждая) отбирали и анализировали, как описано в Примере 8.2. Было обнаружено, что фракции 16-21 активны.
Шаг 2. Белки из отобранных фракций 16-21 осаждали насыщенным раствором (60%) (NH4)2SO4, помещали в 2 мл буфера F и загружали на стандартную колонку Superdex 200 HR 10/30А (GE Healthcare), откалиброванную буфером G (20 мМ Трис-HCl рН 7). Изократическое элюирование проводили при скорости потока 0,5 мл/мин с применением буфера G. Фракции, объемом 0,5 мл (0,5 мл каждая) собирали и анализировали, как описано в Примере 8.2. Были обнаружены активные фракции (12-13).
Шаг 3. Белки из отобранных фракций 12-13 (Шаг 2) загружали на колонку Sourse15Q (1,6 мл) (GE Healthcare), откалиброванную буфером G (20 мМ Трис-HCl рН 7). Элюирование проводили при скорости потока 0,5 мл/мин с применением линейного градиента NaCl (от 0 до 400 мМ в 20 объемах колонки) в буфере G. Фракции (0,5 мл каждая) отбирали и анализировали, как описано в Примере 8.2. Были обнаружены активные фракции (15-17). Обобщенные данные по очистке приведены в Таблице 6.
Шаг 4. Чтобы идентифицировать пептидазы, имеющие специфическую аспартат-пептид-гидролизующую активность, белки нескольких фракций (15-17) подвергали методу денатурирующего гель-электрофореза (ДДС/ПААГ) (Laemmli U.K., Cleavage of structural proteins during the assembly of the head of bacteriophage T4, Nature, 1970, 227:680-685). Профиль элюирования активности сравнивали с профилем элюирования белков. Был обнаружен только один белок, для которого профили активности и элюирования были идентичными.
Очищенный белок экстрагировали из ДДС-геля и расщепляли трипсином (Govorun V.M. et al., Biochemistry (Mosc.), 2003, 68(1):42-49).
Полученную смесь анализировали с использованием метода масс-спектрометрии MALDI-TOF, как описано в работе (Govorun V.M. et al., Biochemistry (Mosc.), 2003, 68(1):42-49). Данные масс-спектра изолированного белка совпали с полученными данными для аминопептидазы A/I (РерА) Е.coli. Таким образом, в штамме Е.coli 5Δ была найдена шестая пептидаза.
8.4. Получение пептидаз-дифицитного штамма Е.coli 6Δ.
Ген рерА удаляли в штамме Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattB, dapE::λattB), как описано в Примере 8.1. Фрагмент ДНК, несущий кассету λattL-cat-λattR, поучали методом ПЦР с использованием праймеров Р9 (SEQ ID NO:36) и Р10 (SEQ ID NO:37) и плазмиды pMW118-λattR-cat-λattL в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattB, dapE::λattB)/pKD46 методом электротрансформации как описано выше для получения штамма Е.coli BW25113(ΔpepB, iadA::λattB, pepE::λattB, ybiK::λattB, dapE::λattB, pepA::λattL-cat-λattR) (штамм Е.coli 6Δ).
8.5. Анализ специфической пептид-гидролизующей активности в штамме Е.coli 6Δ.
Специфическую аспартат-пептид-гидролизующую активность анализировали in vitro, как описано в Примере 8.2. Полученные результаты (Таблица 4) показали, что аспартат-пептид-гидролизующая активность может быть определена в штамме 6Δ и которая оказалась ниже, чем в штаммах 4-5Δ.
Пример 9.
Ферментативное получение дипептидов, имеющих кислую L-аминокислоту, такую как L-Asp или L-Glu, на N-конце, с использованием модифицированных штаммов Е.coli 5Δ и 6Δ, имеющих Lal-активность.
Дипептиды, содержащие кислую L-аминокислоту, такую как L-Asp или L-Glu, на N-конце, получаются с использованием бактерии семейства Enterobacteriaceae, более точно, бактерии, принадлежащей к роду Escherichia, имеющей способность к продукции дипептидов в среде, содержащей или лишенной, например, не ограничиваясь этим, необходимые аминокислоты. Дипептид-продуцирующая бактерия есть штамм Е.coli 5Δ или 6Δ как описано выше, со сниженной пептидазной активностью, который был модифицирован далее, чтобы иметь активность L-аминокислота-α-лигазы. Ген(ы), кодирующий(е) Lal(s), выбран(ы) из группы, состоящей из BBR47_51900, Staur_4851, DES, BUR, ВСЕ, BTH, АМЕ, SFL и BMY, которые введены в хромосому Е.coli или введены в бактериальную клетку на плазмиде, имеющей ген, кодирующий Lal. Ген(ы) кодирующий(е) Lal(s), помещен(ы) под промотор.
Модифицированные штаммы Е.coli 5Δ или 6Δ, содержащие ген(ы), кодирующий(е) Lal(s), и контрольные штаммы 5Δ или 6Δ выращиваются каждый при 28-37°С в течение 18-72 часов в среде Luria-Bertani (также называемой лизогенная среда, как описано в Sambrook, J. and Russell, D.W. (2001) Molecular Cloning: A Laboratory Manual (3rd ed.). Cold Spring Harbor Laboratory Press). Штамм Е.coli 5Δ или 6Δ, содержащий ген(ы), кодирующий(е) Lal(s), вносят в 2 мл питательной среды в пробирках 20×200 мм и выращивают при 28-37°С в течение 18-72 часов на роторной качалке при 250 об./мин.
Состав ферментативной среды (г/л):
|
Ферментационную среду стерилизуют при 116°С в течение 30 мин, глюкозу и СаСО3 стерилизуют отдельно при следующих условиях: глюкозу - при 110°С в течение 30 мин, СаСО3 - при 116°С в течение 30 мин. рН устанавливают при 5-8 раствором KOH.
После выращивания накопленный дипептид измеряют с использованием метода тонкослойной хроматографии (ТСХ). Пластинки ТСХ (10×20 см), покрытые слоем силикагеля (0,11 мм), содержат нефлюоресцирующий индикатор (Sorbpolymer, Krasnodar, Russian Federation). Пластинки ТСХ помещают в подвижную фазу, содержащую 2-пропанол:ацетон:250 мМ аммиак:Н20 как 100:100:12:28. Раствор нингидрина в ацетоне (0,3%, масс./об.) используют в качестве окрашивающего реагента. Определение выполняют при 540 нм.
Пример 10.
Ферментативное получение Asp-Phe с помощью BBR47_51900 и Staur_4851 с использованием системы регенерации АТФ
С целью предотвратить ингибирования ферментов при высокой концентрации АТФ, выход продукта Asp-Phe изучали в реакционной смеси, содержащей BBR47_51900 и Staur_4851, а также фосфоенолпируват и пируваткиназу для регенерации АТФ. Состав реакционной смеси был следующим:
|
Реакции проводили при 37°С в течение 1-48 часов. Отбирали 100 мкл из 1 мл реакционной смеси при каждом времени реакции. Каждую реакционную смесь, в которую добавляли 10 мкл 1 М ЭДТА (рН 9,0) с целью остановки реакции, исследовали методом ВЭЖХ. Условия приведены в Примере 4. Было показано, что BBR47_51900 и Staur_4851 продуцируют Asp-Phe в условиях системы регенерации АТФ (Таблица 9).
Пример 11.
Анализ специфической Asp-Phe-гидролизующей активности в штамме Е.coli 7Δ
11.1. Конструирование штаммов Е.coli 7Δ, дефицитных по Asp-Phe-гидролизующим пептидазам
Ген pepD удаляли в штамме Е.coli JM109. Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р11 (SEQ ID NO:38) и Р12 (SEQ ID NO:39) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB).
Ген рерЕ удаляли в штамме Е.coli JM109 (pepD::ΔattB). Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р13 (SEQ ID NO:40) и Р14 (SEQ ID NO:41) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepB::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB).
Ген iadA удаляли в штамме Е.coli JM109 (pepD::ΔattB, pepE::ΔattB). Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р15 (SEQ ID NO:42) и Р16 (SEQ ID NO:43) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB).
Ген рерА удаляли в штамме Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB). Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р17 (SEQ ID NO:44) и P18 (SEQ ID NO:45) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB).
Ген рерВ удаляли в штамме Е.coli JM109 [pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB). ДНК-фрагмент, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р19 (SEQ ID NO:46) и Р20 (SEQ ID NO:47) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB).
Ген iaaA удаляли в штамме Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB). ДНК-фрагмент, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р21 (SEQ ID NO:48) и Р22 (SEQ ID NO:49) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 {pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB).
Оперон генов dpp (dppA, dppB, dppC, dppD, dppF] удаляли в штамме Е.coli JM109 [pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB). ДНК-фрагмент, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р23 (SEQ ID NO:50) и Р24 (SEQ ID NO:51) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Конечный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB)/pKD46, как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA:::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB).
11.2. Анализ специфической Asp-Phe-гидролизующей активности в штамме Е.coli 7Δ
Клетки Е.coli JM109 и штамма Е.coli 7Δ выращивали в среде с LB-агаром при 37°С в течение 16 часов. Выращенные клетки инокулировали в 20 мл MS среды и выращивали при 37°С до оптической плотности OD610нм ~20. Затем, в среду добавляли Asp-Phe (конечная концентрация 2 мМ) и клетки выращивали далее в течение 32 часов. Конечную культуру при каждом времени выращивания центрифугировали для получения супернатанта культуры. Остаток Asp-Phe в супернатанте культуры анализировали методом ВЭЖХ. Результаты представлены в Таблице 10. Гидролизующая активность по отношению к Asp-Phe в культуре штамма Е.coli 7Δ была гораздо меньше по сравнению с такой активностью в штамме Е.coli JM109.
Состав MS среды (г/л):
|
Ферментационную среду стерилизовали при 121°С в течение 20 минут, за исключением того, что глюкозу, MgSO4×7H2O и СаСО3 стерилизовали раздельно при следующих условиях: глюкоза и MgSO4×7H2O - при 121°С в течение 20 мин, СаСО3 - при 180°С в течение 2 часов. Поддерживали рН до 7 раствором KOH. Специфическая Asp-Phe-гидролизующая активность была ниже в штамме Е.coli 7Δ при сравнении с такой активностью в штамме Е.coli JM109 (Таблица 10).
Пример 12.
Определение способности к продукции Asp-Phe штаммом Е.coli 7Δ, сверхэкспрессирующим ген Lal
Первичную структуру генов, кодирующих BBR47_51900 и Staur_4851, оптимизировали для экспрессии в Е.coli. Гены, кодирующие BBR47_51900 из Brevibacillus brevis NBRC 100599 и Staur_4851 из Stigmatella aurantiaca DW4/3-1, синтезировали с помощью GenScript и получали в качестве набора плазмид pUC57 (pUC57-cBBR и pUC57-cSTA). Нуклеотидные последовательности полученных cBBR и cSTA представлены SEQ ID NO:68 и SEQ ID NO:69, соответственно.
12.1. Конструирование pSF12-cBBR и pSF12-cSTA
Фрагмент ДНК, несущий BBR47_51900, получали с помощью ПЦР с использованием праймеров Р25 (SEQ ID NO:52) и Р2б (SEQ ID NO:53) и плазмиды pUC57-cBBR в качестве матрицы. ПЦР проводили с использованием следующих стадий: 98°С, 30 секунд; (98°С, 15 секунд; 58°С, 10 секунд; 72°С, 1 минута) × 30 циклов; 72°С, 5 минут - в 50 мкл реакционной смеси, включающей 0,04 мкг плазмидной ДНК, 0,2 мкмоль/л каждого праймера, 1,0 Ед. высокоточной ДНК-полимеразы (Phusion High-Fidelity DNA Polymerase, New England Labs), 10 мкл 5 × Phusion HF буфера и 0,2 ммоль/л каждого dNTPs. Очистку полученного ДНК-фрагмента проводили с использованием MinElute PCR Purification Kit (Qiagen).
Фрагмент ДНК, несущий Staur_4851, получали с помощью ПЦР с использованием праймеров Р27 (SEQ ID NO:54) и Р28 (SEQ ID NO:55) и плазмиды pUC57-cSTA в качестве матрицы. Условия ПЦР и метод очистки описаны выше.
Полученные таким образом растворы обрабатывали ферментами рестрикции Nde I и Pst I с целью расщепления полученной ДНК, и затем каждый из ДНК-фрагментов (1,3 т.п.н.) очищали с помощью MinElute Reaction Cleanup Kit (Qiagen).
Вектор pSF12-ggt был сконструирован из вектора pUC18, и содержит промотор rpoH и ген ggt, кодирующий гамма-глутамилтранспептидазу из штамма Е.coli W3110 (WO2013051685 A1). Вектор pSF12-ggt расщепляли с помощью Nde I и Pst I. Фрагменты ДНК разделяли агарозным гель-электрофорезом выделяли ДНК-фрагмент (3,0 т.п.н.) с помощью QIAquick Gel Extraction Kit (Qiagen).
Фрагмент ДНК (1,3 т.п.н.), содержащий ген BBR47_51900 или Staur_4851, и фрагмент ДНК (3,0 т.п.н.), полученный выше, подвергали реакции лигирования с использованием TaKaRa Ligation Kit Ver.2.1 (TaKaRa) при 16°С в течение 30 минут. Компетентные клетки Е.coli JM109 (TaKaRa) трансформировали методом теплового шока с использованием лигирующей реакционной смеси, помещали в среду с LB-агаром, содержащую 100 мкг/мл ампициллина, и выращивали при 30°С в течение ночи. Плазмиду выделяли из колонии трансформанта, выросшего в среде, в соответствии с известным методом. Так были получены pSF12-cBBR и p8F12-cSTA. Последовательность ДНК-векторов подтверждали использованием 3130 Genetic Analyzer (Applied Biosystems).
12.2. Ферментативная продукция Asp-Phe с использованием модифицированных штаммов Е.coli 7Δ, имеющих активность Lal
Бактерия-продуцент дипептида есть вышеописанный штамм Е.coli 7Δ, дефицитный по пептидазе и дипептид-пермеазной активности, далее модифицированный таким образом, чтобы иметь активность L-аминокислота-α-лигазы. Штаммы-продуценты дипептидов содержат ген, кодирующий BBR47_51900 или Staur_4851, введенный в бактериальную клетку на плазмиде pSF12-cBBR или pSF12-cSTA, соответственно. Каждый из генов, кодирующих Lals, помещен под промотор rpoH. Модифицированные штаммы Е.coli 7Δ, содержащие ген, кодирующий Lal, и контрольный штамм 7Δ выращивали при 25°С в течение 24 часов в среде с LB-агаром (содержащей 100 мкг/мл ампициллина для модифицированных штаммов Е.coli 7Δ, содержащих ген, кодирующий Lal). Штаммы Е.coli 7Δ, содержащие ген, кодирующий Lal, и контрольный штамм 7Δ инокулировали в 20 мл MS среды, содержащей 100 мМ L-Asp и 100 мМ L-Phe, в 500 мл колбе Sakaguchi и выращивали при 25°С в течение 32 часов на возвратно-поступательной качалке при 120 об/мин. Полученную культуру центрифугировали для получения супернатанта культуры. Накопленный в супернатанте культуре Asp-Phe анализировали методом ВЭЖХ. Результаты представлены в Таблице 11. Как видно из Таблицы 11, Asp-Phe не был получен с использованием штамма 7Δ, тогда как Asp-Phe продуцировался штаммом 7Δ, содержащим ген, кодирующий Lal.
Пример 13.
Определение способности к продукции Asp-Phe штаммом Е.coli 9Δ/pMGAL1/pHSG-cLal
13.1. Конструирование штамма Е.coli 7Δ, дефицитного по обоим генами tyrR и tyrA.
Сначала, для дерепрессии синтеза ферментов, вовлеченных в биосинтез ароматических аминокислот, удаляли ген tyrR в штамме Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB). Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р29 (SEQ ID NO:56) и Р30 (SEQ ID NO:57) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Полученный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB)/pKD46, как описано в Примере 11.1. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattB).
Затем, удаляли ген tyrA в штамме Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattB), так что префенат, общий интермедиат в биосинтезе Phe и Tyr, не смог бы быть утилизирован в биосинтезе Tyr. Фрагмент ДНК, несущий кассету ΔattL-cat-ΔattR, получали с помощью ПЦР с использованием праймеров Р31 (SEQ ID NO:58) и Р32 (SEQ ID NO:59) и плазмиды pMW118-ΔattL-cat-ΔattR в качестве матрицы. Конечный ДНК-фрагмент вводили в штамм Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattB)/pKD46 как описано выше. CmR-Маркер вырезали из трансформанта Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattB, tyrA::ΔattR-cat-ΔattL) для конструкции штамма Е.coli JM109 (pepD::ΔattB, pepE::ΔattB, iadA::ΔattB, pepA::ΔattB, pepB::ΔattB, iaaA::ΔattB, dpp::ΔattB, tyrR::ΔattB, tyrA::ΔattB).
13.2. Конструкция pHSG-cBBR и pHSG-cSTA
Для конструкции плазмиды pHSG-cBBR вырезали соответствующий фрагмент EcoRI - SphI, содержащий промотор rpoH и ген BBR47_51900, из плазмиды pSF12-cBBR расщеплением с помощью EcoRI и SphI и затем лигировали с вектором pHSG396 (TaKaRa), обработанным такими же рестриктазами.
Для конструкции плазмиды pHSG-cSTA, ДНК-фрагмент, несущий Staur_4851 под промотором rpoH, получали с помощью ПЦР с использованием праймеров РЗЗ (SEQ ID NO:60) и Р34 (SEQ ID NO:61) и плазмиды pSF12-cSTA в качестве матрицы. ПЦР проводили, используя следующую программу: 98°С, 30 секунд; (98°С, 15 секунд; 58°С, 10 секунд; 72°С, 1 минута) × 30 циклов; 72°С, 5 минут - в 50 мкл реакционной смеси, включающей 0,04 мкг плазмиды ДНК, 0,2 мкмоль/л каждого праймера, 1,0 Ед. высокоточной ДНК-полимеразы (Phusion High-Fidelity DNA Polymerase, New England Labs), 10 мкл 5 × Phusion HF буфера и 0,2 ммоль/л каждого dNTPs. Затем ДНК-фрагмент (1,5 т.п.н.), расщепленный с помощью BamHI и XhoI, лигировали с вектором pHSG396, обработанным такими же рестриктазами.
13.3. Трансформация векторов pMGAL1 и pHSG-cLal в штамм Е.coli 9Δ
Вектор pMGAL1 был сконструирован из pMW19 (Wako), и содержит три гена, вовлеченных в биосинтез Phe в Е.coli: pheA, aroG4 и aroL, кодирующие хоризматмутаза-префенатдегидратазу (CM-PD), 3-деокси-D-арабиногептулозонат-7-фосфатсинтетазу (DAHP синтетаза) и шикимат киназу (SK), соответственно (JP3225597). Каждый из нативных генов pheA и aroG4 был мутирован с целью снятия ингибирования по типу обратной связи Phe.
Векторы pMGAL1 и pHSG-cLal одновременно вводили в штамм Е.coli 9Δ методом электропорации с использованием среды с LB-агаром, содержащей 100 мкг/мл ампициллина и 25 мкг/мл хлорамфеникола. Полученный таким образом штамм был назван Е.coli 9Δ/pMGALl/pHSG-cLal.
13.4. Ферментативная продукция Asp-Phe с помощью Е.coli 9Δ/pMGAL1/ pHSG-cLal
Модифицированный штамм Е.coli 9Δ/pMGAL1/pHSG-cLal и контрольный штамм 9Δ/pMGAL1 каждый выращивали при 25°С в течение 24 часов в среде с LB-агаром (содержащей 100 мкг/мл ампициллина и 25 мкг/мл хлорамфеникола для Е.coli 9Δ/pMGAL1/pHSG-cLal). Штамм Е.coli 9Δ/pMGAL1/pHSG-cLal и контрольный штамм 9Δ/pMGAL1 инокулировали в 20 мл MS среды, содержащей 100 мМ L-Asp, в 500 мл колбе Sakaguchi и выращивали при 25°С в течение 72 часов на возвратно-поступательной качалке при 120 об/мин. Полученную культуру центрифугировали для получения супернатанта культуры. Накопленные в супернатанте культуры Phe и Asp-Phe анализировали методом ВЭЖХ. Результаты представлены в Таблице 12. Как видно из Таблицы 12, Asp-Phe не был получен с использованием штамма 9Δ/pMGAL1, тогда как Asp-Phe продуцировался штаммом 9Δ/pMGAL1, содержащим ген, кодирующий Lal.
Пример 14.
Анализ субстратной специфичности DES
14.1. Конструирование плазмиды pELAC-MBP-DES-HT
14.1.1. Конструирование вспомогательной плазмиды pELAC
Фрагмент ДНК получали с помощью ПЦР с использованием праймеров Р35 (SEQ ID NO:62), Р36 (SEQ ID NO:63) и ДНК-плазмиды pUC18 (GenBank: L08752.1) в качестве матрицы. Полученный ДНК-фрагмент расщепляли с помощью BglII и XbaI и клонировали в плазмиду pET22(b+) (Novagen, каталожный No. 69744-3), разрезанную такими же эндонуклеазами. Так была сконструирована плазмида pELAC.
14.1.2. Конструирование вспомогательной плазмиды pELAC-MBP-HT
Ген malE (без последовательности сигнального пептида) получали с помощью ПЦР с использованием праймеров Р37 (SEQ ID NO:64), Р38 (SEQ ID NO:65) и хромосомной ДНК Е.coli MG1655 в качестве матрицы. Полученный ДНК-фрагмент расщепляли с помощью XbaI и BamHI и клонировали в вектор pELAC-/XbaI-BamHI. Так была сконструирована плазмида pELAC-MBP-HT.
14.1.3. Конструирование плазмиды pELAC-MBP-DES-HT
Для конструирования плазмиды pELAC-MBP-DES-HT был получен ДНК-фрагмент, содержащий ген DES, с использованием праймеров Р39 (SEQ ID NO:66), Р40 (SEQ ID NO:67) и плазмиды pUC57-DES (настоящая патентная заявка) в качестве матрицы. Полученный ДНК-фрагмент расщепляли с помощью BamHI и NotI и лигировали с вектором pELAC-MBP-HT/BamHI-NotI. Так была сконструирована плазмида pELAC-MBP-DES-HT.
14.2. Экспрессия и очистка МВР-гибридного DES с His6-тэгом
Клетки Е.coli 7Δ, содержащие pELAC-MBP-DES-HT, выращивали в LB-среде, содержащей 100 мкг/мл ампициллина, в пробирках при 37°С до достижения OD610нм ~2. Затем 2 мл полученной культуры инокулировали в 100 мл LB-среды, содержащей IPTG (конечная концентрация 0,1 ммоль/л), в 500 мл колбе Sakaguchi при 30°С в течение 8 часов. Индуцированные клетки отбирали из 1,6 л питательного бульона, ресуспендировали в 200-240 мл HT-II буфера (50 мМ Трис-HCl рН 8,0, 0,3 М NaCl, 10 мМ имидазола, 15% глицерина) и обрабатывали ультразвуком с использованием соникатора (INSONATOR 201М, KUBOTA). Дебрис удаляли центрифугированием при 4°С и 14,000 об/мин в течение 15 минут, затем пропускали через фильтр 0,45 мм (Milhpore). Раствор необработанных белков загружали на картридж HisTALON Superflow, 5 мл (Clontech) с использованием AKTA avant 25 (GE Healthcare) в соответствии с рекомендациями производителя. Фракции, содержащие МВР-гибридный DES с Hise-тэгом, объединяли и высаливали с использованием колонок PD-10 (GE Healthcare), калиброванных SC-буфером (50 мМ Трис-HCl рН8,0, 0,3 М NaCl, 15% глицерин).
14.3. Анализ Asp-Phe-синтезирующей активности МВР-гибридного DES с His6-тэгом
Синтезированные с помощью МВР-гибридного DES с His6-тэгом дипептиды определяли с использованием ВЭЖХ-анализа реакционной смеси, которая содержала:
|
Реакции проводили при 37°С в течение 15 часов. Затем проводили ВЭЖХ-анализ реакционной смеси, в которую добавляли 10 мкл 1 М EDTA, рН 9,0 для остановки ферментативной реакции. Условия реакции были такие же, как описано в Примере 4. Было показано, что DES катализирует образование 0,30 мМ Asp-Phe.
Дополнительный пример 1.
Таблица 2 (идентичность, identity) и Таблица 3 (схожесть, similarity) показывают множественные выравнивания белков BBR47_51900 и Staur_4851c известными L-аминокислота-α-лигазами (Lals). Как видно из Таблиц 2 и 3, белки BBR47_51900 и Staur_4851 идентичны на не более чем 25% (Таблица 2) и схожи на не более чем 43% (Таблица 3) с известными Lals.
Дополнительный пример 2.
Таблица 7 (идентичность, identity) и Таблица 8 (схожесть, similarity) показывают значения парного выравнивания для белков BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY. Как видно из Таблиц 7 и 8, белки BBR47_51900, Staur_4851, DES, BUR, ВСЕ, ВТН, АМЕ, SFL и BMY идентичны на не более чем 25% (Таблица 7) и схожи на не более чем 44% (Таблица 8).
Хотя указанное изобретение описано в деталях со ссылкой на наилучшие способы осуществления изобретения, для специалиста в данной области техники очевидно, что могут быть совершены различные изменения и произведены эквивалентные замены и такие изменения и замены не выходят за рамки настоящего изобретения. Каждому из упомянутых выше документов соответствует ссылка, и все цитируемые документы являются частью описания настоящего изобретения.
|
|
|
|
|
|
|
|
|
|
|
|